Populační analýza Streptococcus pneumoniae sérotypu 19A metodou sekvenace celého genomu v České republice a Evropě po jeho zařazení do pneumokokové konjugované vakcíny
Population analysis of Streptococcus pneumoniae serotype 19A by whole genome sequencing in the Czech Republic and in Europe after serotype 19A inclusion in pneumococcal conjugate vaccine
Aim: To present the results of whole genome sequencing (WGS) analysis of Streptococcus pneumoniae serotype 19A and to compare them with the respective data from Europe. The vaccine serotype 19A is widely distributed in the Czech Republic.
Material and methods: WGS was used in this study as the most powerful available method for detailed characterization of S. pneumoniae. Nineteen Czech isolates of S. pneumoniae 19A were analysed and compared with 415 European isolates included in the PubMLST database.
Results: S. pneumoniae serotype 19A causes all types of pathogen - host interaction, from carriage to noninvasive and invasive pneumococcal disease (IPD). In 2010 – 2019, 3872 cases of IPD were reported within the surveillance programme in the Czech Republic, with 323 of these caused by serotype 19A. WGS data of the Czech serotype 19A isolates show a numerous and genetically related subpopulation of three sequencing types: ST-199, ST-416, and ST-3017. Within this subpopulation, the largest is the cluster of nine ST-199 isolates. High relatedness of ST-199 isolates is also confirmed by the fact that all but one isolate, 117/2019 (novel rST - -137805), share the same ribosome sequencing profile - rST-11365. Outside the above-mentioned subpopulation, there are only four isolates that form three separate genetic lines of serotype 19A. A highly similar situation is observed across European countries, where about half of all serotype 19A isolates form a genetically closely related subpopulation (ST-199, ST-416, ST-450, ST-667, ST-3017, and ST-10360) while isolates which are not part of this subpopulation represent a large number of unrelated genetic lines.
Conclusions: The study has shown a mostly homogeneous population of S. pneumoniae serotype 19A to circulate in the post-vaccination era in both the Czech Republic and Europe, with some unrelated isolates located outside this population.
Keywords:
Streptococcus pneumoniae – whole genome sequencing – WGS – vaccine serotype 19A – pneumococcal conjugate vaccine – PCV
Autori:
J. Kozáková
; Z. Okonji
; M. Honskus
Pôsobisko autorov:
Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha
Vyšlo v časopise:
Epidemiol. Mikrobiol. Imunol. 70, 2021, č. 2, s. 110-117
Kategória:
Původní práce
Súhrn
Cíl práce: Využití výsledků sekvenace celého genomu (WGS) Streptococcus pneumoniae sérotypu 19A a jeho porovnání s výsledky v Evropě. Tato publikace prezentuje analýzu vakcinačního sérotypu 19A, který je v České republice hojně zastoupen.
Materiál a metodika: Pro studii byla zvolena metoda WGS jako nejdetailnější aktuálně dostupná metoda pro charakterizaci S. pneumoniae. Analýze bylo podrobeno 19 českých izolátů S. pneumoniae 19A, které byly porovnány se 415 evropskými izoláty, jejichž data jsou dostupná v mezinárodní databázi PubMLST.
Výsledky: S. pneumoniae sérotypu 19A způsobuje všechny typy interakce s hostitelem od nosičství, přes vyvolání neinvazivních onemocnění až po invazivní pneumokokové onemocnění (IPO). V letech 2010–2019 bylo v ČR zjištěno v programu surveillance 3 872 případů IPO, z nichž 323 případů bylo způsobeno sérotypem 19A. Celogenomová data českých izolátů sérotypu 19A ukazují početnou a geneticky blízkou subpopulaci tří sekvenačních typů ST-199, ST-416, ST-3017. V rámci této subpopulace je nejpočetnější klastr devíti izolátů ST-199. Vysokou příbuznost izolátů ST-199 potvrzuje i fakt, že s výjimkou izolátu 117/2019 (nový rST-137805), sdílí všechny stejný ribozomální sekvenační profil – rST-11365. Stranou zmíněné subpopulace stojí pouze čtyři izoláty, které tvoří tři zcela samostatné genetické linie sérotypu 19A. Velice obdobná situace je na evropské úrovni. Zde zhruba polovina všech izolátů sérotypu 19A vytváří geneticky blízkou subpopulaci (ST-199, ST-416, ST-450, ST-667, ST-3017, ST-10360) a izoláty, které nejsou součástí této subpopulace, představují velké množství vzájemně nepříbuzných genetických linií.
Závěry: Studie ukazuje převážně homogenní populaci S. pneumoniae sérotypu 19A kolující v postvakcinační éře v ČR i Evropě se stranou stojícími nepříbuznými izoláty.
Klíčová slova:
Streptococcus pneumoniae – sekvenace celého genomu – metoda WGS – vakcinační sérotyp 19A – pneumokoková konjugovaná vakcína – PCV
ÚVOD
Streptococcus pneumoniae (S. pneumoniae, pneumokok) je grampozitivní extracelulární bakteriální patogen, neustále vykazující celosvětově vysokou nemocnost a smrtnost. Vyskytuje se jako součást normální mikroflóry horních cest dýchacích, ale je současně i nejčastějším bakteriálním původcem běžných infekcí respiračního traktu. Pokud se pneumokok dostane do primárně sterilního prostředí, může způsobit tzv. invazivní pneumokoková onemocnění (IPO) s nejčastějšími klinickými formami: pneumonie, meningitidy či sepse. Všechny případy IPO nahlášené v národní surveillance České republiky odpovídají platné evropské i české definici případu IPO, case definition, kdy je S. pneumoniae prokázán z klinického materiálu, který je za normálních podmínek sterilní [1].
S. pneumoniae je velice variabilní bakterie, kterou na podkladě polysacharidového pouzdra lze zařadit již k více než 100 sérotypům. Díky molekulárním metodám jsou stále definovány nové sérotypy [2]. Podle složení pneumokokových vakcín se sérotypy rozlišují na tzv. vakcinační sérotypy ve vakcínách obsažené a nevakcinační sérotypy ve vakcínách neobsažené. Určení sérotypu S. pneumoniae je velice důležitým krokem a v ČR probíhá pouze v Národní referenční laboratoři pro streptokokové nákazy (NRL) kombinací sérologické Quellung reakce a end-point multiplexové polymerázové řetězové rekce (mPCR) [3].
Vakcinace je jednoznačně nejúčinnější prevencí IPO. Účinná vakcinační strategie je vytvářena na podkladě kvalitní surveillance, která v ČR probíhá od roku 2007. Z dat surveillance IPO vyplývá, že nejohroženějšími skupinami jsou malé děti a senioři [4]. Od roku 2010 je v ČR zavedeno plošné očkování dětí pneumokokovými konjugovanými vakcínami (PCV). Po původní sedmivalentní PCV7 jsou aktuálně v ČR dostupné dva druhy konjugovaných PCV vakcín (desetivalentní PCV10 a třináctivalentní PCV13). Očkování dětí probíhá v režimu doporučeného hrazeného očkování. Od roku 2018 je očkování vakcínou PCV13 rozšířeno i pro pacienty se zdravotní indikací a pro věkovou skupinu seniorů 65 a starších bez poplatku. Dříve byla pro seniory doporučována především pneumokoková polysacharidová vakcína.
19A je vakcinační sérotyp, který se vyskytuje v PCV13. PCV7 ani PCV10 sérotyp 19A neobsahuje, ale obsahují sérotyp 19F. Je popisována imunologická zkřížená reaktivita mezi sérotypy 19F a 19A, která by teoreticky mohla vést ke zkřížené ochraně proti pneumokokovým onemocněním způsobeným sérotypem 19A [5].
I přes možnost vakcinace patří sérotyp 19A mezi jeden z nejčetnějších sérotypů S. pneumoniae způsobujících dlouhodobě v ČR IPO. Proto je populační studie metodou sekvenace celého genomu (WGS) zaměřená na izoláty tohoto sérotypu v období po zahájení vakcinace PCV13 v ČR tzn. po roce 2010. Předkládáme první výsledky studia izolátů z období 2017–2019.
MATERIÁL A METODY
Bakteriální izoláty Streptococcus pneumoniae
Náhodným výběrem z izolátů došlých do NRL v letech po zavedení vakcinace byl připraven soubor 19 izolátů. Jednalo se o S. pneumoniae sérotypu 19A izolovaných v letech 2017–2019 (4krát 2017, 2krát 2018, 13krát 2019) – tabulka 1). Jelikož se do NRL dostávají především invazivní pneumokokové izoláty, tvořily v souboru převážnou část. 17 izolátů pochází z případů IPO (materiál: krev, likvor, sekční materiál) a 2 z neinvazivních pneumokokových onemocnění (materiál: bronchoalveolární laváž, sputum). Izoláty byly uchovány v mrazicím boxu při teplotě -80 °C (KryobankaB, Itest s.r.o.). Kultivace probíhala v CO2 atmosféře při 35 °C společně s optochinovým diskem a v kombinaci s testem rozpustnosti ve žluči. Určení sérotypů S. pneumoniae bylo provedeno pomocí Quellung reakce v kombinaci s reakcí end-point multiplexPCR (mPCR) [3].
Table 1. Molecular characterization of 19 isolates of S. pneumoniae serotype 19A, Czech Republic 2010–2019
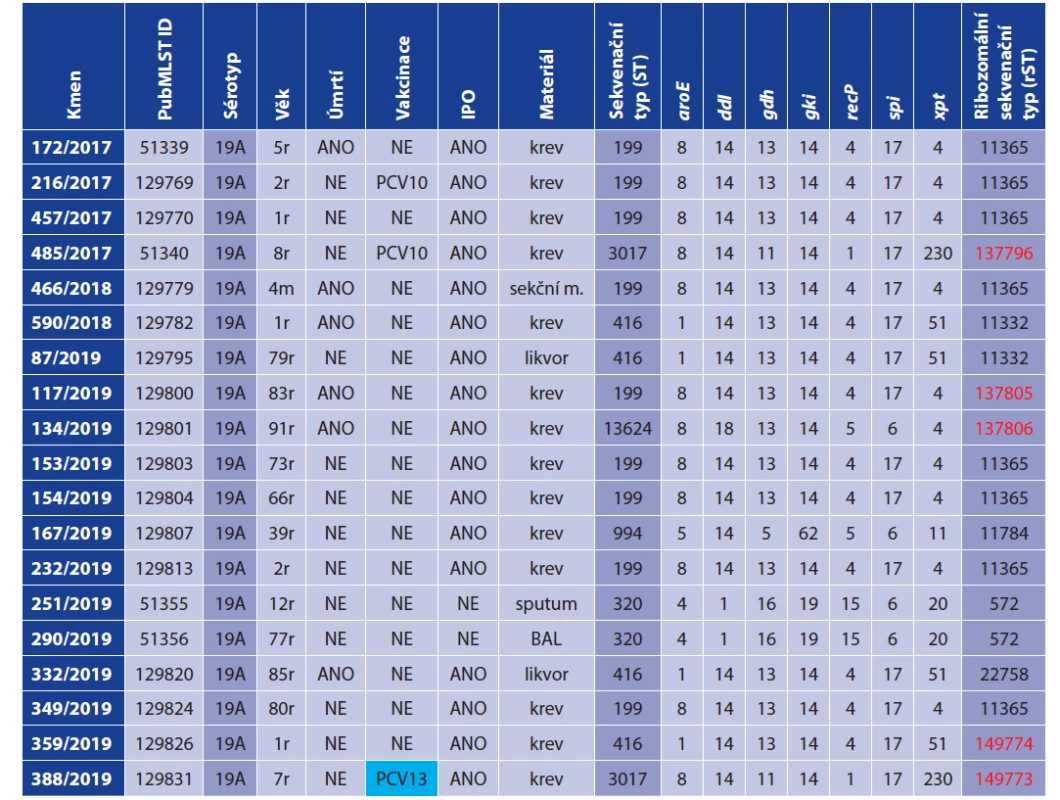
r – year, m – month, BAL – bronchoalveolar lavage; sekční m. – sectional, autopsy material; blue case of vaccination failure; red marked newly described rST
Extrakce DNA
Z izolátů byla izolována deoxyribonukleová kyselina (DNA) pomocí izolačních kitů QIAamp DNA Mini Kit (QIAGEN) a MagAttract HMW DNA Kit (QIAGEN). Postup izolace probíhal podle pokynů výrobce [6, 7].
Celogenomová sekvenace a úprava dat
Celogenomová sekvenace izolátů S. pneumoniae proběhla v EMBL (European Molecular Biology Laboratory, Heidelberg, Německo) s využitím přístroje Illumina Mi - Seq. K sestavení výsledných genomů z primárních raw dat byl v NRL využit software Velvet de novo Assembler (assembly de novo) dle již rutině používaného postupu NRL [8]. Průměrná hodnota parametru “K-mer length“ byla 151 (127–169) a průměrný počet kontigů v sestavených genomech byl 65 (48–142). Získaná celogenomová data byla vložena do databáze PubMLST [9] pod následujícími IDs: 51339, 51340, 129769, 129770, 129779, 129782, 129795, 129800, 129801, 129803, 129804, 129807, 129813, 51355, 51356, 129820, 129824, 129826, 129831.
Analýza a vizualizace WGS dat
Jednotlivé celogenomové izoláty S. pneumoniae byly v databázi PubMLST automaticky skenovány a následně charakterizovány alelovým profilem 53 ribozomálních genů (rpsA-rpsU, rplA-rplF, rplI-rplX, rpmA-rpmJ) a 7 MLST genů (aroE, ddl, gdh, gki, recP, spi, xpt). Na základě alelových variant zmíněných genů byl u každého izolátu následně určen sekvenační typ (ST) a ribozomální sekvenační typ (rST). Nepopsané alelové varianty byly skenovány manuálně, následně anotovány a přidány do PubMLST databáze s novým číselným označením.
Tvorba fylogenetických sítí proběhla s využitím programu Genome Comparator, který je součástí PubMLST databáze, v režimu “all loci“, který srovnává izoláty na nejvyšší úrovni rozlišení (1420 lokusů). “Distance matrix“, které jsou založené na variabilitě všech srovnávaných lokusů napříč izoláty, byly vygenerovány automaticky a na jejich základě byly vytvořeny fylogenetické sítě v programu SplitsTree4, který využívá algoritmus NeighborNet. Editace a úprava grafických výstupů proběhla v programu Inkscape.
Pro populační srovnání našich 19 izolátů S. pneumoniae sérotypu 19A byla využita volně dostupná data v databázi PubMLST. Pro analýzu byly vybrány všechny dostupné evropské izoláty sérotypu 19A s kompletními profily ST a rST, které byly zachyceny od roku 2010 do 2019 a které obsahovaly celogenomová data. Těmto požadavkům odpovídalo ke dni zahájení analýzy (28. 1. 2021) celkem 415 evropských izolátů z následujících zemí: Bělorusko (n = 7), Belgie (n = 1), Finsko (n = 1), Německo (n = 9), Island (n = 97), Irsko (n = 1), Polsko (n = 15), Slovinsko (n = 6), Nizozemsko (n = 6), Velká Británie (n = 272). Ze 415 evropských celogenomových izolátů byla pouze u 119 k dispozici dostatečná epidemiologická data, díky kterým je možné rozlišit jejich primární zdroj a typ interakce s pneumokokem (20krát IPO, 99krát nosiči). U zbylých 296 izolátů nebylo možné zdroj určit [9].
VÝSLEDKY A DISKUSE
V letech 2010–2019 bylo v ČR zjištěno v programu surveillance 3872 případů IPO, z čehož 323 případů bylo způsobeno sérotypem 19A (graf 1). Jedná se o sérotyp S. pneumoniae, který se v ČR za posledních deset let každoročně vyskytuje mezi pěti nejčastěji se vyskytujícími sérotypy. Aktuálně jde o druhý nejčastější sérotyp [4]. Z pohledu věkové distribuce je maximum onemocnění v seniorské věkové skupině nad 65 let, ale pravidelně se objevují i případy dětí pod 4 roky věku (graf 2).
Graph 1. Invasive pneumococcal disease, Czech Republic, 2010–2019

Graph 2. Age distribution of IPD caused by serotype 19A, Czech Republic, 2010–2019

Za sledované období 2010–2019 došlo v jednom případě i k prokázanému selhání vakcinace. Jednalo se o zdravé, řádné očkované dítě, kterému byla aplikována vakcína PCV13 podle souhrnu údajů o přípravku. Přesto v 7 letech onemocnělo IPO s projevem bakteriemické pneumokokové pneumonie (viz tabulka 1 modře zvýrazněný izolát S. pneumoniae z krve číslo 388/2019). Případ byl reportován na farmakovigilanční oddělení Státního ústavu pro kontrolu léčiv.
Sérotyp 19A není problematický pouze v ČR, a proto se na něj zaměřuje mnoho evropských studií [10]. Na - příklad studie ve Velké Británii popisuje, že míra výskytu IPO sérotypu 19A se u dětí ve věku < 5 let po zavedení PCV13 roku 2010 snížila o 91 % ve srovnání s obdobím před PCV13 [11]. Podle dat z Norska po zavedení vakcinace PCV13 v roce 2011 došlo ke snížení celkového počtu IPO a stejně tak k poklesu sérotypu 19A u dětí pod pět let věku i starších [12]. Pokles IPO 19A hlásí i Řecko. Zde se po zavedení PCV13 v roce 2010 procento IPO způsobených 19A snížilo z 22 % v roce 2011 na 11,4 % v roce 2015 [13]. Další studie se zaměřují na prokázání zvýšeného potenciálu sérotypu 19A k vyvolání IPO [14, 15].
Analýza WGS dat českých izolátů
Vybraných 19 izolátů sérotypu 19A patřilo k 6 různým sekvenačním typům: ST-199 (9krát), ST-416 (4krát), ST-320 (2krát), ST-3017 (2krát), ST-994 (1krát) a ST-13624 (1krát). Ve všech případech se jednalo o sekvenační typy, které byly v PubMLST databázi popsané. Známé kombinace ribozomálních genů byly zjištěny u 14 izolátů: rST-11365 (8krát), rST-572 (2krát), ST-11332 (2krát), rST-11784 (1krát) a rST-22758 (1krát). U 5 zbylých izolátů byly nalezeny mutované varianty ribozomálních genů. Po registraci a zařazení těchto nových alel do PubMLST databáze bylo popsáno 5 nových ribozomálních sekvenačních typů (viz tab. 1) – červeně značeno. Izolát 485/2017 (nový rST-137796) nesl nově popsanou alelovou variantu genu rpmC (31553), která se lišila od známé alely 14 jednonukleotidovou substitucí. Izolát 117/2019 (nový rST-137805) obsahoval novou alelu genu rpsL (46666) a izolát 134/2019 s novou alelou genu rplA (57126) definoval nový rST-137806. U izolátu 388/2019 (nový rST-149773) byla nalezena nová alelová varianta genu rplN (47043), která se lišila od známé alely 16 jednonukleotidovou substitucí. Izolát 359/2019 (nový rST-149774) nesl novou alelu genu rplM (49771), která se lišila od nejbližší známé varianty tohoto genu ve dvou nukleotidech.
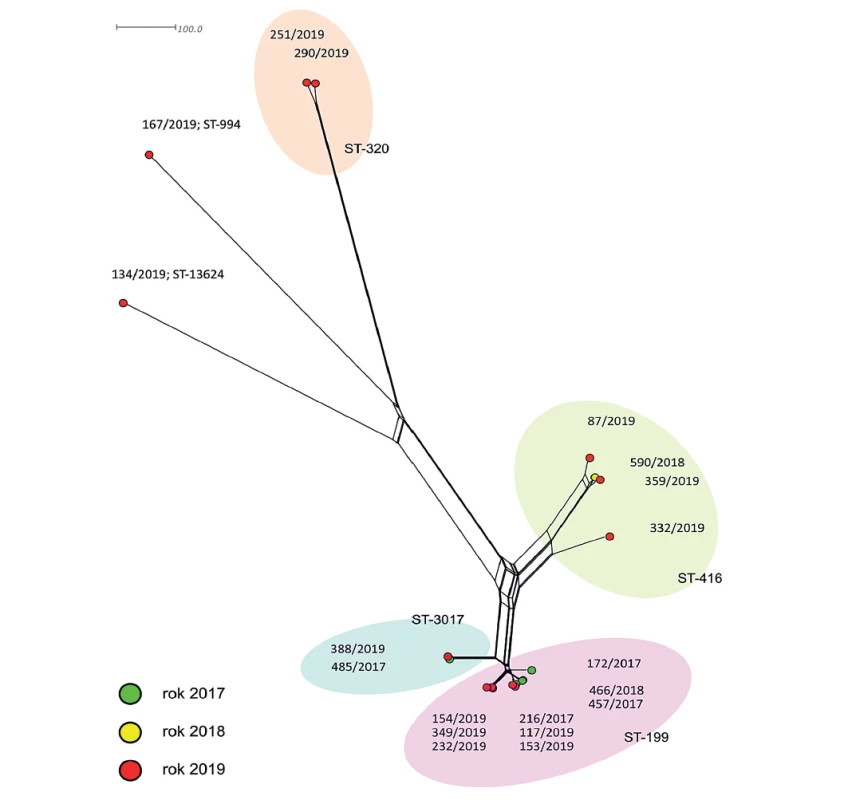
Při pohledu na fylogenetickou síť (obr. 1) izolátů sérotypu 19A z České republiky lze pozorovat především početnou a geneticky blízkou subpopulaci, která obsahuje izoláty tří různých sekvenačních typů (ST-199, ST-416, ST-3017). Nejpočetnější je klastr devíti izolátů ST-199, který je tvořen třemi skupinami téměř identických kmenů a samostatně stojícím izolátem 172/2017. Všechny izoláty jsou geneticky vysoce blízké a rozdíl mezi dvěma nejméně příbuznými tvoří maximálně cca 70 genů. Vysoká příbuznost izolátů ST-199 se zobrazuje i ve faktu, že s výjimkou izolátu 117/2019 (nový rST-137805), sdílí všechny stejný ribozomální sekvenační profil – rST-11365. V těsné blízkosti klastru ST-199 leží dva izoláty ST-3017 z let 2017 a 2019. Genetický rozdíl mezi skupinami izolátů ST-199 a ST-3017, který je cca 100 genů, reprezentují tři odlišné MLST alely (gdh, recP, xpt). Izoláty ST-3017 jsou vzájemně téměř totožné. Jedním z mála genetických rozdílů mezi nimi jsou odlišné mutace u alel ribozomálních genů a tedy i odlišný ribozomální sekvenační typ (oba nově popsané). Alelový profil sekvenačního typu ST-416 se liší od ST-199 pouze ve dvou alelách MLST genů (aroE, xpt). Celková genetická odlišnost izolátů těchto dvou sekvenačních typů je však vyšší, než tomu je v případě izolátů ST-3017, čemuž odpovídá i pozice izolátů ST-416 na fylogenetické síti. Klastr ST-416 je tvořen třemi geneticky blízkými izoláty 590/2018 (rST-11332), 87/2019 (rST-11332), 359/2019 (nový rST-149774) a izolátem 332/2019 (rST - -22758), který je v porovnání s ostatními izoláty ST-416 geneticky vzdálenější. Zbývající izoláty, které nepatří k výše popsané subpopulaci (ST-199, ST-416, ST-3017), tvoří tři zcela samostatné genetické linie sérotypu 19A. První linii reprezentují dva vysoce příbuzné izoláty ST-320 (rST-572) z roku 2019. Jedná se o dva neinvazivní české izoláty zařazené do studie. Druhá linie je zastoupena izolátem 134/2019 (ST-13624, nový rST-137806) a třetí linii vytváří izolát 167/2019 (ST-994, rST-11784). Tyto samostatné linie jsou geneticky nepříbuzné jak k výše popsané subpopulaci, tak k sobě navzájem.
Figure 1. Visualization of genetic diversity of serotype 19A in 19 isolates from the Czech Republic
Srovnání WGS dat českých a evropských izolátů
Druhá fylogenetická síť na obrázku 2 zobrazuje populační srovnání 19 izolátů S. pneumoniae sérotypu 19A z České republiky s dostupnými evropskými celogenomovými izoláty sérotypu 19A v databázi PubMLST z let 2010–2019. Jedná se izoláty z invazivních onemocnění, neinvazivních onemocnění i od zdravých nosičů. Lze pozorovat, že zhruba polovina všech evropských izolátů sérotypu 19A vytváří geneticky blízkou subpopulaci. Detailnější výřez obrázku 2 na obrázku 3 dokumentuje, že se tato subpopulace skládá z izolátů šesti hlavních sekvenačních typů (ST-199, ST-416, ST-450, ST-667, ST-3017 a ST-10360) a jednotek dalších, které byly od těchto ST mutacemi odvozeny. Nejvíce zastoupené jsou izoláty sekvenačního typu ST-199, které na fylogenetické síti vytváří v rámci subpopulace několik oddělených a geneticky poměrně rozmanitých linií. K jedné z linií, kterou můžeme definovat jako druhou nejpočetnější, patří 8 z 9 českých izolátů ST-199. Izolát 172/2017 pak přísluší k sousední, nejpočetnější linii evropských izolátů 19A. Evropské izoláty ST-416 formují na fylogenetické síti dvě geneticky blízké linie. První linie zahrnuje české izoláty 590/2018, 87/2019 a 359/2019, zatímco poslední český izolát 332/2019 přísluší k sesterské linii izolátů ST-416. Sekvenační typ ST-3017 je ve výběru evropských izolátů zastoupen pouze deseti izoláty a vytváří na fylogenetické síti samostatnou, vysoce kompaktní linii, kterou tvoří geneticky téměř identické izoláty. Kromě dvou českých izolátů se v linii ST-3017 nachází 7 izolátů z Islandu a 1 izolát z Velké Británie.
Figure 2. Visualization of genetic diversity of the population of S. pneumoniae serotype 19A from Europe
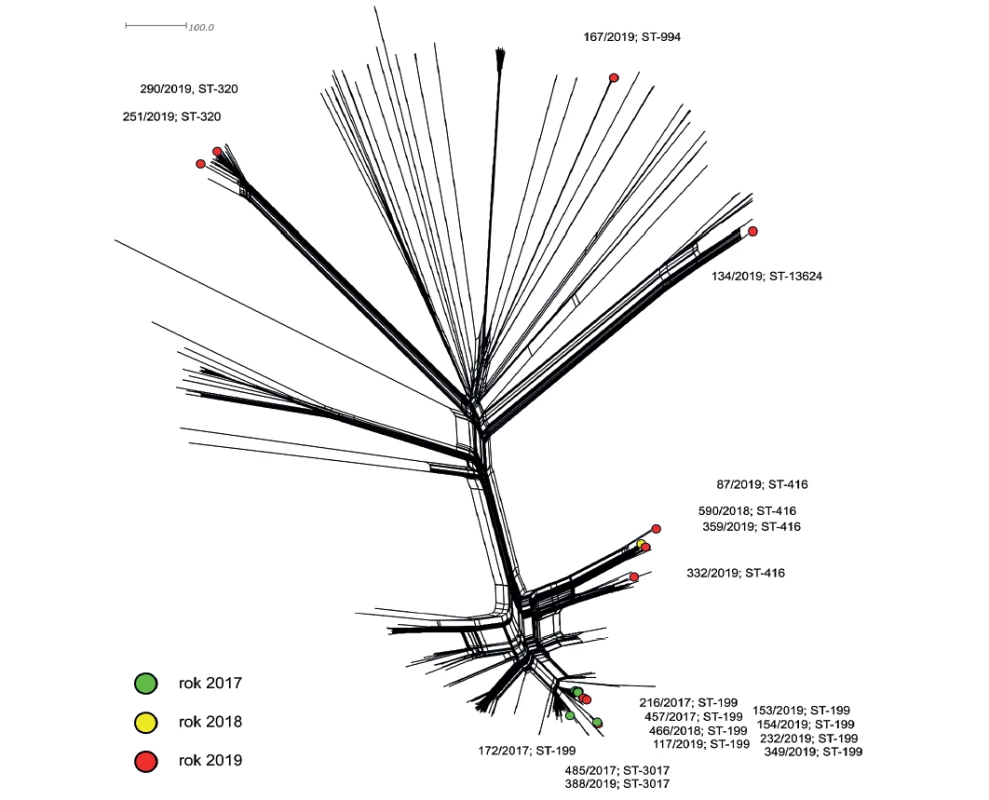
Figure 3. Visualization of genetic diversity of the European subpopulation of S. pneumoniae serotype
19A isolates (ST-199, ST-416, ST-450, ST-667, ST-3017, and ST-10360)
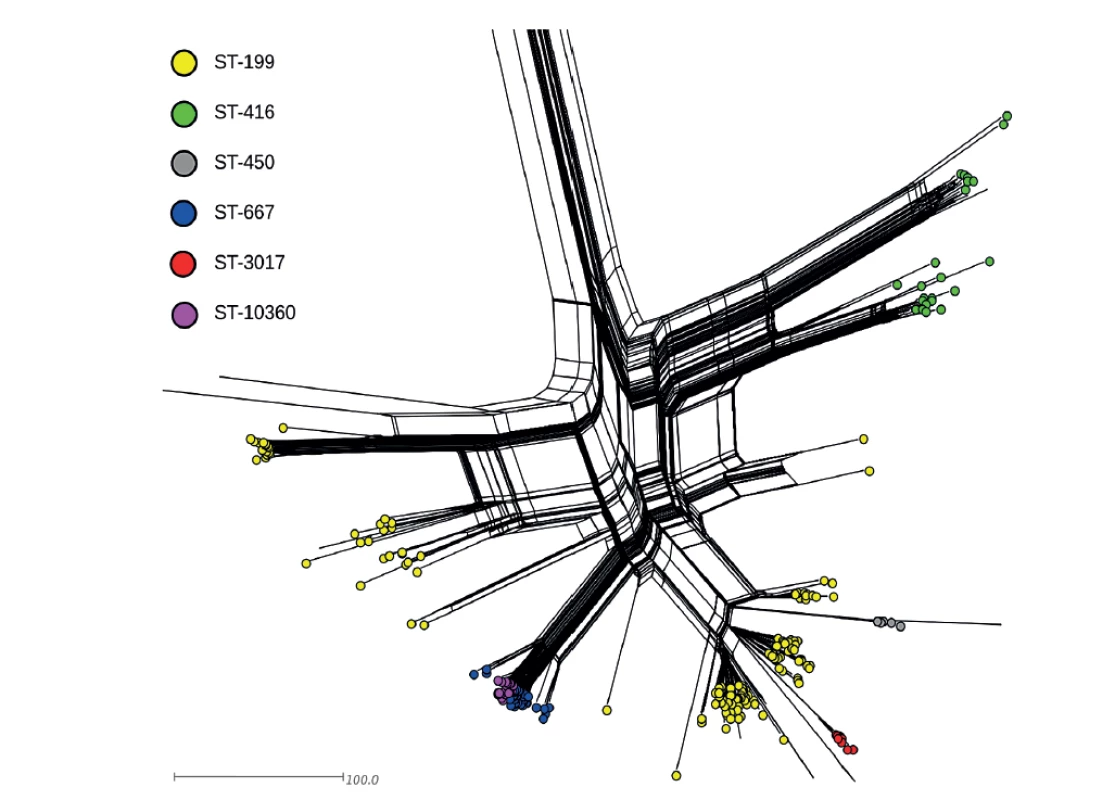
Evropské izoláty sérotypu 19A, které nejsou součástí výše popsané evropské subpopulace (ST-199, ST-416, ST-450, ST-667, ST-3017, ST-10360), vytváří na fylogenetické síti velké množství vzájemně nepříbuzných genetických linií. Tyto genetické linie jsou reprezentovány různým počtem izolátů. Můžeme pozorovat jak linie, které jsou definovány pouze jedním izolátem, tak linie početnější. To je v souladu s výsledky naší předešlé studie, ve které jsme izoláty jednoho sérotypu definovali jako geneticky nehomogenní skupinu o určitém počtu nepříbuzných genetických klastrů, které společně sdílí geny zodpovědné za jejich zařazení ke konkrétnímu sérotypu [8].
Jedna z početnějších genetických linií obsahuje především izoláty sekvenačního typu ST-320 (n = 37) a šest izolátů, které nesou příbuzné sekvenační typy (ST-1451, ST-4768, ST-15674). Společně s dvěma českými neinvazivními izoláty ST-320 tvoří tuto genetickou linii invazivní i neinvazivní izoláty z Velké Británie (n = 23), Polska (n = 10), Běloruska (n = 4), Slovinska (n = 2), Irska (n = 1) a Islandu (n = 1).
Český izolát 167/2019 (ST-994) vytváří další samostatnou linii společně se sedmi téměř identickými izoláty z Velké Británie.
Sekvenační typ ST-13624 sdílí s českým izolátem 134/2019 izolát z Velké Británie z roku 2015, nicméně jejich pozice prozrazuje, že míra jejich vzájemné příbuznosti není tak vysoká, jako tomu bylo v případě izolátů ST-994. Tato genetické linie nese kromě dvou izolátů ST-13624 ještě sesterskou větev, která obsahuje 12 izolátů z Velké Británie (ST-246, ST-695), 2 izoláty z Nizozemska (ST-13983) a jeden izolát ze Slovinska (ST-246).
ZÁVĚRY
Celogenomová data izolátů sérotypu 19A z České republiky ukazují početnou subpopulaci, která je zastoupena třemi sekvenačními typy – ST-199, ST-416, ST-3017. Všechny izoláty jsou geneticky vysoce blízké. Nejpočetnější je klastr devíti izolátů ST-199. Vysokou příbuznost izolátů ST-199 dokládá i fakt, že s výjimkou izolátu 117/2019 (nový rST-137805), sdílí všechny stejný ribozomální sekvenační profil – rST-11365. Čtyři izoláty, které nepatří k popisované subpopulaci, tvoří tři zcela samostatné genetické linie sérotypu 19A. Jedná se o izolát ST-994, ST-13624 a dva izoláty ST-320. Velice obdobná situace je na evropské úrovni. Zde zhruba polovina všech izolátů sérotypu 19A vytváří geneticky blízkou subpopulaci (ST-199, ST-416, ST-450, ST-667, ST-3017, ST-10360) a izoláty, které nejsou součástí této subpopulace, představují velké množství vzájemně nepříbuzných genetických linií.
Díky získání celogenomových dat izolátů S. pneumoniae 19A se může jejich studium dále zabývat invazivitou či rezistencí na antibiotika, to všechno jsou u sérotypu 19A velice zajímavá témata.
Poděkování
Podpořeno z programového projektu Ministerstva zdravotnictví ČR s reg. č. 17-29256A, 2017-2020. Veškerá práva podle předpisů na ochranu duševního vlastnictví jsou vyhrazena.
Do redakce došlo dne 5. 3. 2021.
Adresa pro korespondenci:
MUDr. Jana Kozáková
SZÚ Praha Šrobárova 48 100 42 Praha 10
e-mail: jana.kozakova@szu.cz
Zdroje
1. Vyhláška č. 275/2010 Sb., kterou se mění vyhláška č. 473/2008 Sb., o systému epidemiologické bdělosti pro vybrané infekce, příloha č. 21 invazivní pneumokoková onemocnění.
2. Ganaie F, Saad JS, McGee L, Tonder AJ, Bentley SD, Lo SW, Gladstone RA, Turner P, Keenan JD, Breiman RF, Nahm MH. A New Pneumococcal Capsule Type, 10D, is the 100th Serotype and Has a Large cps Fragment from an Oral Streptococcus. mBio, 2020;11(3).
3. Vacková Z, Klímová M, Kozáková J. Nová metoda a schéma typizace Streptococcus pneumoniae. Epidemiol Mikrobiol Imunol, 2013;62(2):50–58.
4. Kozáková J, Okonji Z, Šebestová H, Klímová M, Křížová P. Invazivní pneumokokové onemocnění v České republice v roce 2019. Zprávy CEM (SZÚ, Praha), 2019;28(7):277–282.
5. Hausdorff WP, Hoet B, Schuerman L. Do pneumococcal conjugate vaccines provide any cross-protection against serotype 19A? BMC Pediatr, 2010;10 : 4.
6. Manuál QIAamp DNA Mini Kit. Dostupné na www: https://www. qiagen.com/us/resources/resourcedetail?id=62a200d6-faf4 - 469b-b50f-2b59cf738962&lang=en
7. MagAttract HMW DNA Kit. Dostupné na www: https://www.qiagen. com/us/resources/resourcedetail?id=3e331b6c-0742-483b -b74f-a7be2d4c1116&lang=en.
8. Kozáková J, Honskus M, Okonji Z. Implementace a využití metody sekvenace celého genomu (WGS) v surveillance invazivního pneumokokového onemocnění, Česká republika, 2017–2019. Epidemiol Mikrobiol Imunol, 2020;69(3):134–141.
9. https://pubmlst.org/organisms/streptococcus-pneumoniae.
10. Isturiz R, Sings HL, Hilton B, Arguedas A, Reinert RR, Jodar L. Streptococcus pneumoniae serotype 19A: worldwide epidemiology. Expert Review of Vaccines, 2017;16(10):1007–1027.
11. Waight PA, Andrews NJ, Ladhani SN, et al. Effect of the 13-valent pneumococcal conjugate vaccine on invasive pneumococcal disease in England and Wales 4 years after its introduction: an observational cohort study. Lancet Infect Dis, 2015;15 : 535–543.
12. Steens A, Bergsaker MA, Aaberge IS, Rønning K, Vestrheim DF. Prompt effect of replacing the 7-valent pneumococcal conjugate vaccine with the 13-valent vaccine on the epidemiology of invasive pneumococcal disease in Norway. Vaccine, 2013;16,31(52):6232–6238.
13. Koutouzis EI, Daikos GL, Chatzichristou P, Michos AG, Koutouzi FI, Chrousos GP, Syriopoulou VP. Characteristics of Streptococcus pneumoniae serotype 19A isolates from children in the pre and post Conjugate Vaccine Era. Single center experience 1986 – 2015. Vaccine, 2018;23,36(35):5245–5250.
14. Varon E, Cohen R, Béchet S, Doit C, Levy C. Invasive disease potential of pneumococci before and after the 13-valent pneumococcal conjugate vaccine implementation in children. Vaccine, 2015;17,33(46):6178–6185.
15. Weinberger DM, Harboe ZB, Sanders EA, Ndiritu M, Klugman KP, Rückinger S, Dagan R, Adegbola R, Cutts F, Johnson HL, O’Brien KL, Scott JA, Lipsitch M. Association of serotype with risk of death due to pneumococcal pneumonia: a meta-analysis. Clin Infect Dis, 2010;15,51(6):692–699.
Štítky
Hygiena a epidemiológia Infekčné lekárstvo MikrobiológiaČlánok vyšiel v časopise
Epidemiologie, mikrobiologie, imunologie

2021 Číslo 2
- Parazitičtí červi v terapii Crohnovy choroby a dalších zánětlivých autoimunitních onemocnění
- Očkování proti virové hemoragické horečce Ebola experimentální vakcínou rVSVDG-ZEBOV-GP
- Koronavirus hýbe světem: Víte jak se chránit a jak postupovat v případě podezření?
Najčítanejšie v tomto čísle
- Necholerová vibria – výskyt nejen v Evropě v posledních letech
- Borrelia miyamotoi – další emergentní patogen přenášený klíšťaty
- Substance use and addictive behaviours during COVID-19 confinement measures increased in intensive users: Results of an online general population survey in the Czech Republic
- Séroprevalence protilátek proti spalničkám u zaměstnanců Fakultní nemocnice Olomouc před přeočkováním a po přeočkování – pilotní studie
