Možnosti zoonotického přenosu rotavirových infekcí
The possibilities of zoonotic transmission of rotaviruses
Rotaviruses are significant enteric pathogens of humans and animals. Rotavirus-associated mortality is still high in children, especially in developing countries. Rotaviruses also account for important economic losses due to gastrointestinal disease of livestock animals, notably of young cattle and pigs. Increasing numbers of different rotavirus strains stress the necessity of their uniform nomenclature and detailed classification with the use of whole-genome sequencing. Phylogenetic analyses of the rotavirus genome reveal repeated intersections between the evolution of human and animal rotavirus strains which is probably a consequence of multiple events of transmission among various animal species. The interspecies transmission and subsequent gene reassortment are important mechanisms driving the diversity of rotaviruses and enabling the emergence of new pathogenic strains.
Keywords:
rotavirus – gastroenteritis – zoonotic transmission – gene reassortment
Authors:
R. Moutelíková; J. Prodělalová
Authors‘ workplace:
Výzkumný ústav veterinárního lékařství, oddělení virologie, Brno
Published in:
Epidemiol. Mikrobiol. Imunol. 64, 2015, č. 2, s. 66-71
Category:
Review Article
Overview
Rotavirus představuje významného původce gastroenteritid u lidí i zvířat. Mortalita dětí v důsledku rotavirové infekce je hlavně v rozvíjejících se zemích stále vysoká. Rotaviry způsobují také významné ekonomické ztráty v souvislosti s gastroenteritidami hospodářských zvířat, zvláště u telat a selat. Přibývající počty odlišných kmenů rotavirů ukazují na nutnost používat jednotnou nomenklaturu a podrobnou klasifikaci založenou na sekvenci celého genomu viru. Fylogenetická analýza genomu rotavirů poukazuje na časté prolínání vývoje lidských a zvířecích kmenů, pravděpodobně v důsledku mnohonásobných přenosů mezi různými živočišnými druhy. Mezidruhový přenos a následné přeskupení genů je jedním z mechanismů přispívajících k velké genetické rozmanitosti rotavirů, což představuje riziko vzniku nových patogenních kmenů.
Klíčová slova:
rotavirus – gastroenteritida – zoonotický přenos – reasortment genů
ÚVOD
Rotaviry patří celosvětově mezi nejčastější původce akutní gastroenteritidy (přibližně 1/3 všech případů) u dětí mladších 5 let. Fatální případy rotavirové gastroenteritidy se podílejí na úmrtnosti dětí do 5 let z 5 %, nejvíce ohrožené jsou děti z nízkopříjmových zemí [12].
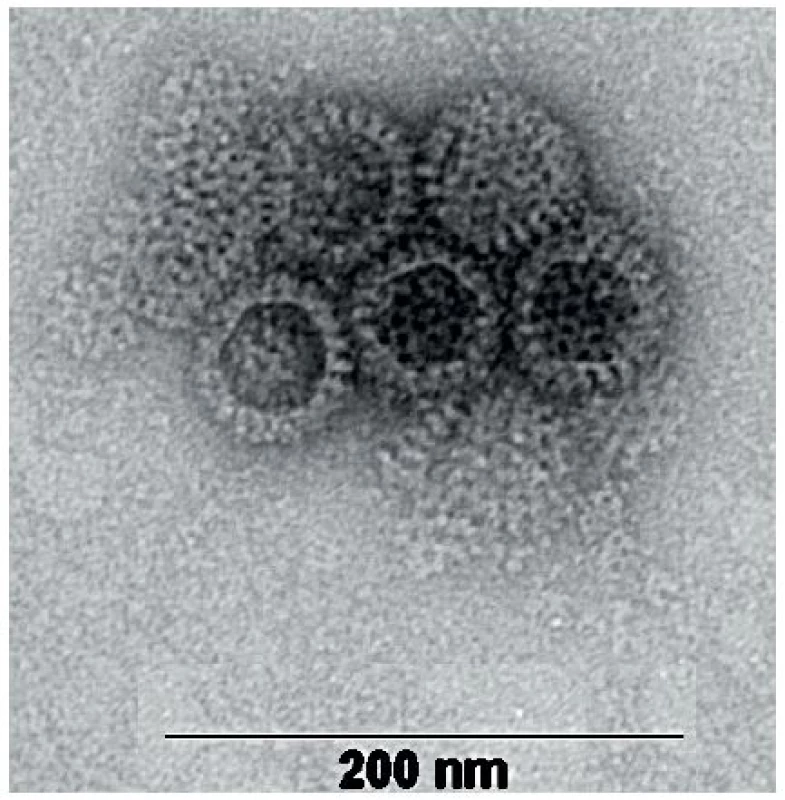
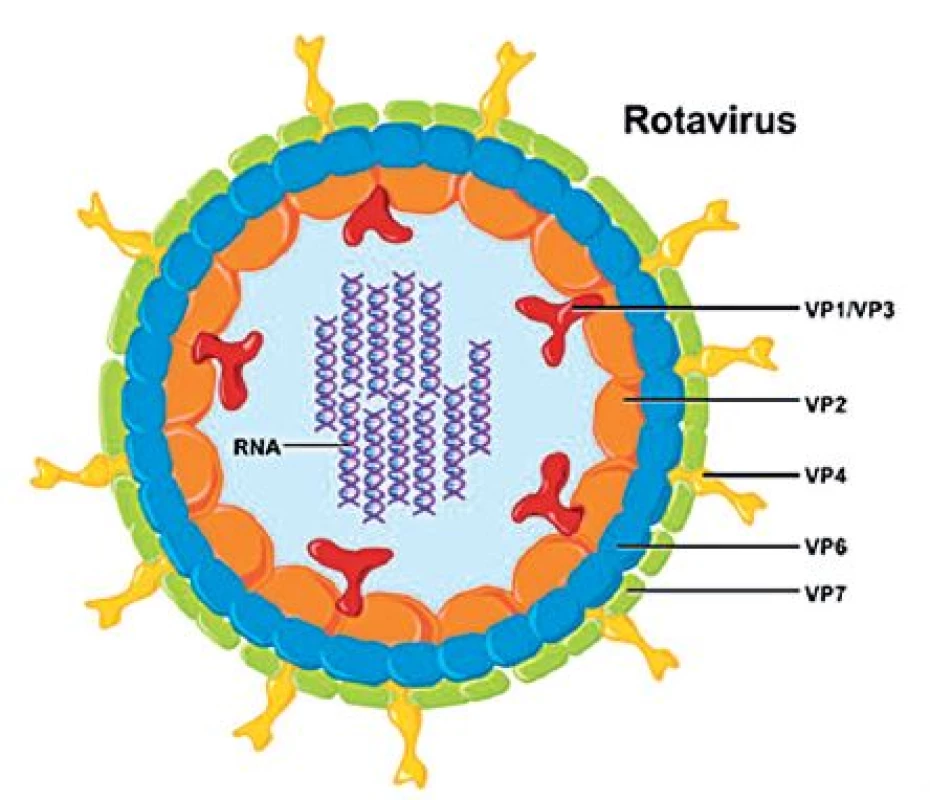
Rotaviry jsou neobalené viry z čeledi Reoviridae s poměrně složitou morfologií. Kompletní virion má v průměru 70–75 nm a je tvořen jádrem s 11 segmenty dvouřetězcové (ds) RNA a ostře ohraničenou trojvrstevnou kapsidou s typickou strukturou, která se v elektronovém mikroskopu jeví jako ozubené kolo s loukotěmi, což dalo tomuto rodu jméno (kolo – lat. rota) – obrázek 1. Jednotlivé segmenty RNA genomu kódují šest strukturálních virových proteinů (VP1–VP4, VP6 a VP7) a pět (případně šest u některých rotavirů A) nestrukturálních proteinů (NSP1–NSP5/NSP6) [52]. Stavba rotavirové částice je vidět na obrázku 2.
KLASIFIKACE ROTAVIRŮ
Rod Rotavirus zahrnuje v současnosti 5 druhů (Rotavirus A – Rotavirus E) a 3 prozatím nezařazené druhy (Rotavirus F, Rotavirus G a Rotavirus H) [3].
Pro zařazení rotavirů do jednotlivých druhů (běžněji označovaných jako skupiny) je nutné potvrdit molekulárně-biologickými metodami podobnost nukleotidové sekvence genu pro VP6 vyšší než 75 % [27]. Diagnostické soupravy detekující přímo rotavirový antigen jsou zaměřeny na virový protein VP6. Běžně používanými diagnostickými kity proto není možné odhalit infekci jinými druhy (skupinami) rotavirů, než je RVA.
Od roku 1989 se klasifikace RVA opírá o binární klasifikační systém, který vznikl na základě reaktivity s hyperimunními séry obsahujícími neutralizační protilátky proti dvěma hlavním povrchovým antigenům VP4 a VP7. Takto byly rotavirové kmeny zařazovány do P (VP4) sérotypu a G (VP7) sérotypu. Názvy sérotypů byly vytvořeny podle hlavních charakteristik antigenů – P jako proteáza-senzitivní a G jako glykoprotein [14]. Vzhledem k tomu, že antigenní charakterizace kmenů je časově náročná, a díky stále větší dostupnosti sekvenačních metod byla původní klasifikace do sérotypů nahrazena klasifikačním systémem VP4 a VP7 genotypů, které jsou určeny na základě sekvenační analýzy a podobnosti mezi jednotlivými genovými segmenty. Dosud bylo popsáno nejméně 27 G genotypů a 35 P genotypů [27]. G sérotypy jsou ve většině případů shodné s určenými G genotypy. Naproti tomu pro antigenní a genetickou klasifikaci VP4 bylo nutné použít dvě čísla. P sérotyp, je-li známý, bývá označen číslicí, za níž někdy následuje velké písmeno, a P genotyp je označen číslem v hranaté závorce (P1A[8], P2C[6] apod.). U nově popsaných kmenů rotavirů se uvádí už jen číslo genotypu.
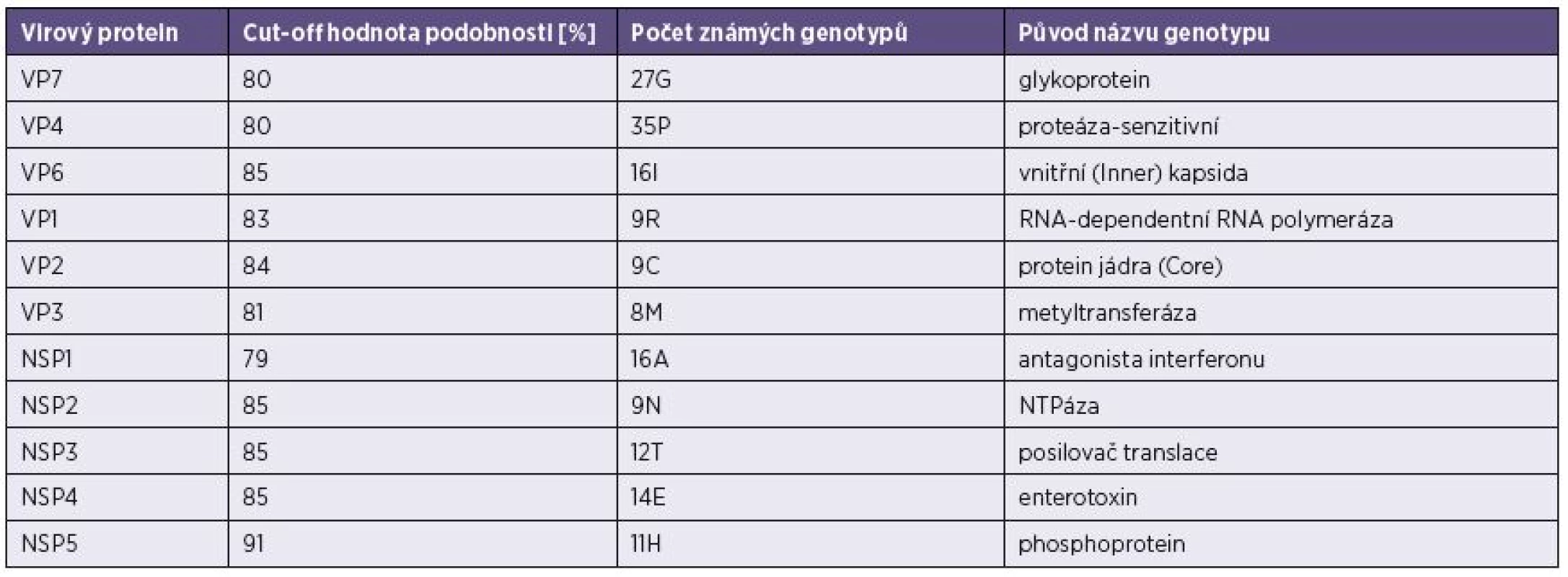
S přibývajícím počtem izolovaných RVA kmenů se ukázalo jako nezbytné vytvoření jednotného klasifikačního systému, který by zahrnul všech 11 segmentů rotavirového genomu a rozdělil jednotlivé geny do genotypů na základě definovaných cut-off hodnot procentní podobnosti nukleotidové sekvence jednotlivých genů. Na základě fylogenetických analýz dostupných RVA sekvencí byl v roce 2008 vytvořen systém klasifikace RVA [26] – tabulka 1. Tento systém zařazuje každý segment genomu do jednoho genotypu, takže schematický popis jednotlivých RVA kmenů vypadá následovně: Gx-P[x]-Ix-Rx-Cx-Mx-Ax-Nx-Tx-Ex-Hx, kde jsou seřazeny jednotlivé genotypy (VP7-VP4-VP6-VP1-VP2-VP3-NSP1-NSP2-NSP3-NSP4-NSP5/6) a x znamená číslo daného genotypu.
Obdobný přehledný systém klasifikace je v současné době zaváděn i pro rotaviry skupin B a C [24, 25].
Nový systém klasifikace rotavirů umožňuje snadněji odhalit a popsat nezvyklé či nové kombinace genotypů, a tak postihnout obrovskou genetickou rozmanitost rotavirů. Tato rozmanitost je způsobena několika různými mechanismy, z nichž největší význam má genový reasortment (přeskupení). Tento unikátní mechanismus výměny RNA segmentů mezi různými kmeny rotavirů může nastat v případě smíšené infekce dvěma či více kmeny, kdy vznikají nové genetické a antigenní varianty rotavirů. Reasortment RNA segmentů je ovlivněn mnoha faktory, jako jsou původ virových kmenů, druh hostitele či vliv okolního prostředí [22]. Tento jedinečný evoluční mechanismus může vyústit v překročení mezidruhové bariéry a výměnu genových segmentů mezi rotaviry původně druhově specifickými.
ROTAVIROVÉ INFEKCE LIDÍ
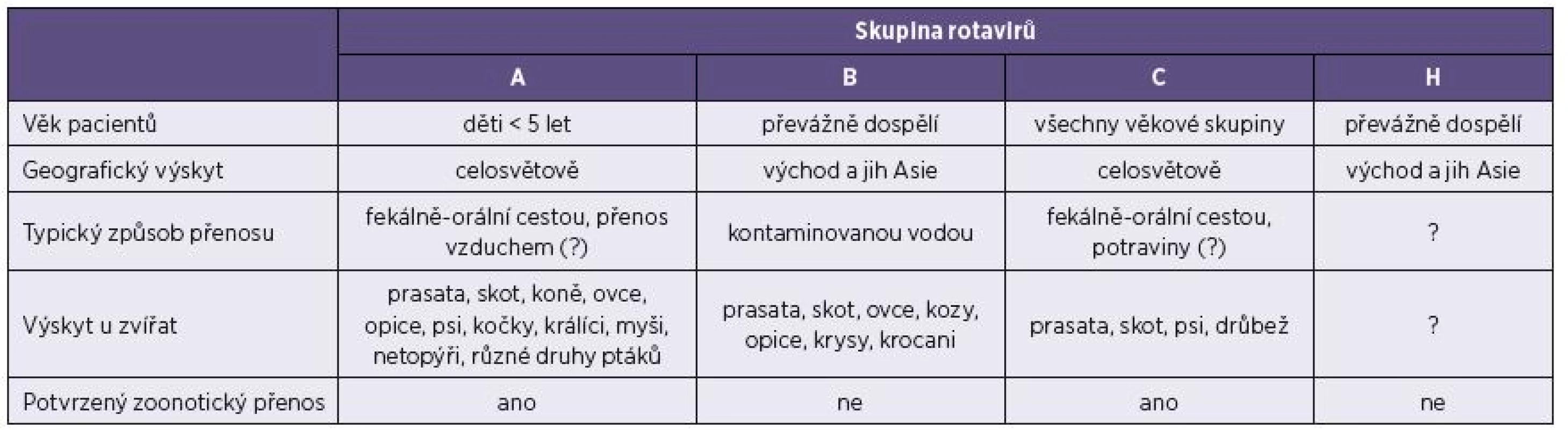
Člověk může být infikován rotaviry skupin A, B, C a H – tabulka 2. První popis rotavirové infekce u lidí se datuje k roku 1973 [9], kdy byly ve stolici dětí postižených akutní gastroenteritidou odhaleny metodou elektronové mikroskopie negativně barveného preparátu typické virové částice. Rozvoj diagnostických metod (především přímá detekce virového antigenu ve vzorcích stolice) odhalil rotavirus A (RVA) jako nejčastější příčinu gastroenterálního onemocnění kojenců a malých dětí [39]. Patogenita lidských rotavirů je značná, v rozvojových zemích se rotaviry uvádějí jako příčina více než 400 000 úmrtí ročně u dětí mladších 5 let, což představuje 37 % úmrtí následkem průjmového onemocnění. Z epidemiologického hlediska je významný také fakt, že téměř každé dítě do 5 let věku nejméně jedenkrát prodělá rotavirovou infekci (80 % dětí je infikováno v kojeneckém věku mezi 6.–9. měsícem) [2, 12]. V České republice jsou rotaviry A v současnosti podle oficiálních údajů z databáze Epidat třetím nejčastějším původcem gastroenteritid – 11,2 % průjmových onemocnění celkem (u dětí do 5 let 23,0 %). Skutečný výskyt je však pravděpodobně několikanásobně vyšší, neboť většina nákaz je hlášena na základě laboratorního vyšetření hospitalizovaných pacientů a při objasňování epidemických výskytů [40].
Epidemiologie RVA je značně komplikovaná. Přenos rotavirů se uskutečňuje – obdobně jako u dalších enterálních virů – především fekálně-orální cestou. Charakteristickou vlastností neobalených virů je značná stabilita v prostředí a vysoká odolnost vůči běžným dezinfekčním prostředkům. V tepelně neupravených potravinách a v pitné i užitkové vodě zůstávají rotaviry stabilní po dobu až několika týdnů [28, 55]. Aktivita RVA v tropických oblastech je celoroční, kdežto v zemích mírného pásu vykazují RVA vyhraněnou sezonnost s maximální aktivitou během zimních a jarních měsíců [41].
Více než 90 % vzorků lidských RVA izolovaných v zemích Evropy, které se zúčastnily programu EuroRotaNet mezi roky 2005 a 2009, patřilo mezi pět nejběžnějších G a P kombinací (G1P[8], G2P[4], G3P[8], G4P[8] a G9P[8]) [21]. Kromě těchto běžných G a P genotypů se v posledních letech začal s vyšší frekvencí vyskytovat genotyp G12, který byl dosud izolován v různých zemích Evropy, v Asii i v USA [16].
Další skupiny rotavirů způsobující onemocnění u lidí jsou rotaviry B, C a H, někdy také souhrnně označované non-A rotaviry. Rotaviry skupiny B (RVB) byly poprvé popsány v souvislosti s rozsáhlou epidemií gastroenteritidy v roce 1982 v Číně, která postihla přibližně jeden milion převážně dospělých osob [46]. V nedávné době byl RVB popsán jako původce průjmových onemocnění u dospělých i u dětí mimo území Číny (Bangladéš, Barma), přičemž tyto novější izoláty se geneticky odlišují od původního čínského izolátu [1, 4].
Rotaviry C (RVC) byly nejdříve popsány jako příčina gastroenterálního onemocnění u sajícího selete v roce 1980 v USA [45]. První zpráva o RVC jako původci gastroenteritidy u člověka pochází z Austrálie [44], následně byl tento virus prokázán v mnoha zemích Evropy, Asie a Ameriky. Podle dostupných studií se RVC podílí na všech gastroenterálních onemocněních 0,4 % v Maďarsku, 4,4 % ve Španělsku, 10,2 % v Japonsku a až 16,1 % v USA [8, 13, 42, 47]. Soustavné sledování výskytu RVC u lidí se neprovádí, údaje o podílu RVC na případech onemocnění GIT se liší v závislosti na použité metodice a výchozím souboru vzorků (děti, dospělí). Obecně se předpokládá, že RVC způsobuje méně než 10 % všech případů gastrointestinálních infekcí, což je srovnatelné s počtem infekcí způsobených astroviry či adenoviry. Vzhledem k odlišným povrchovým antigenům jednotlivých rotavirových skupin není možné non-A rotaviry detekovat běžně používanými diagnostickými soupravami zaměřenými na průkaz RVA antigenu metodou ELISA, imunochromatograficky či latexovou aglutinací. RVC ve srovnání s lidskými RVA má nižší patogenitu, většinou způsobuje izolované případy gastroenteritid nebo epidemie malého rozsahu a nevykazuje sezonní výskyt. Infekce RVC postihuje obvykle starší děti a dospělé [36]. Genetická variabilita lidských RVC je ve srovnání s prasečími kmeny velmi nízká. Převážná většina dosud známých lidských RVC je fylogeneticky blízce příbuzných a zařazených do stejných genotypů. Naproti tomu prasečí RVC kmeny zahrnují nejméně 8 G genotypů a 5 P genotypů. [23, 31]. Nabízí se otázka, zda tato nízká rozmanitost lidských kmenů neukazuje na kratší dobu výskytu RVC u lidí než u zvířat, např. prasat, tzn. na možné zavlečení do lidské populace ze zvířecího rezervoáru v minulosti. Překročení mezidruhové bariéry bylo u RVC – stejně jako u RVA – již dříve prokázáno [15, 33].
Rotavirus H (RVH) prozatím zahrnuje virus NADRV, který byl popsán jako původce epidemie gastroenteritidy dospělých v Číně roku 1997 [51]. Dále je do této skupiny zařazen kmen B219, který byl izolován při výskytu průjmového onemocnění v Bangladéši v roce 2002 [35].
ROTAVIROVÉ INFEKCE HOSPODÁŘSKÝCH ZVÍŘAT A DOMÁCÍCH MAZLÍČKŮ
Rotaviry jsou jednou z nejčastějších příčin průjmu u prasat, skotu i koní. Postižená bývají převážně mláďata, u kterých silná dehydratace může vést až k úhynu, čímž dochází ke značným ekonomickým ztrátám [6, 10, 54]. Nejčastějším původcem průjmů bývá RVA, další druhy rotavirů bývají diagnostikovány méně často. Infekce RVC u prasat často probíhá bezpříznakově [32].
U domácích mazlíčků je vzhledem k mnohdy těsnému kontaktu majitelů se zvířaty nutné dobře poznat zvířecí patogeny, zvláště jedná-li se o zoonotické mikroorganismy. Počty doma chovaných psů a koček jsou vysoké. Odhaduje se, že ve světě je chováno 100 miliónů psů a nejméně 80 miliónů koček. Infekce rotaviry se u psů a koček vyskytují běžně, infekce však mívají mírnější průběh než u lidí a hospodářských zvířat, popřípadě probíhají asymptomaticky [37].
Rotaviry infikují také různé druhy ptáků. RVA byl zjištěn u kuřat, krocanů, perliček, bažantů, křepelek, holubů a pštrosů [38, 48]. Ptačí RVA jsou s lidskými rotaviry příbuzné jen velmi vzdáleně, naproti tomu rotaviry izolované z prasat, skotu, koček a psů vykazují v některých případech vysokou míru podobnosti s izoláty humánního původu.
MEZIDRUHOVÝ PŘENOS
Jednotlivé kmeny rotavirů přednostně infikují živočišný druh, pro který byly popsány jako homologní. Infekce různých živočichů či člověka heterologním kmenem nicméně může nastat jak za experimentálních podmínek, tak i v normálním prostředí. Například v průběhu klinických zkoušek různých typů rotavirových vakcín byly testovány opičí nebo bovinní kmeny, které se v lidském organismu množily sice v nižší míře než ve svém homologním hostiteli, ale přesto byly schopny vyvolat imunitní odpověď [49].
Za experimentálních podmínek bylo též prokázáno, že rotavirus může vyvolat infekci a způsobit onemocnění u heterologního zvířecího modelu. Virulentní lidské kmeny RVA jsou schopné způsobit průjem a virémii u selat [5].
Mezidruhová bariéra může být v případě rotavirů překročena dvěma hlavními způsoby:
- přímým mezidruhovým přenosem a
- přenosem spojeným s reasortmentem.
Mezidruhový přenos rotavirových genů pomocí reasortmentu je považován za důležitý zdroj rozmanitosti kmenů v rámci jednotlivých hostitelů [29]. Podrobný průzkum genotypů lidských RVA v rámci projektu EuroRotaNet ukázal, že přibližně 2 % všech typizovaných kmenů, kterých bylo více než 36 000, představují potenciálně zoonotické rotaviry, popř. hybridní lidské-zvířecí kmeny [21]. V literatuře je možné nalézt mnoho studií zmiňujících případy infekce člověka zvířecími RVA kmeny. Wang et al. popisuje detekci G4P[6] kmene rotaviru u dítěte hospitalizovaného s akutní gastroenteritidou [50]. Tento RVA kmen vznikl reasortmentem prasečích a bovinních kmenů. Ve východní Indii byl popsán neobvyklý G8P[4] rotavirus způsobující akutní gastroenteritidu u 3–14měsíčních dětí. Celogenomové sekvenování zde prokázalo zoonotický přenos rotaviru od přežvýkavců [34]. Rotavirové kmeny izolované od pacientů s akutní gastroenteritidou v Thajsku byly popsány jako genotyp G9P[19]. Jejich podrobná genetická analýza odhalila prasečí původ nejméně 9 genových segmentů [17].
Bylo by chybné se domnívat, že k zoonotickému přenosu dochází především v zemích s nižšími hygienickými standardy, kde je běžný užší kontakt s hospodářskými zvířaty. V Maďarsku byl prováděn podrobný průzkum RVA kmenů izolovaných od dětí mladších 15 let s akutní rotavirovou infekcí. Genetická analýza kmenů s neobvyklými kombinacemi povrchových antigenů odhalila blízkou příbuznost s bovinními, prasečími, kočičími, koňskými a králičími rotaviry [7]. Podobně byl v Belgii popsán RVA kmen G9P[6], který byl izolován od 1měsíčního kojence hospitalizovaného s akutním průjmovým onemocněním. Celogenomové sekvenování a následná fylogenetická analýza ukázaly, že se jednalo o přímý přenos rotaviru od prasete na člověka, což bylo potvrzeno i zjištěním, že otec postiženého chlapce byl zaměstnán na prasečí farmě [53]. Podobně přímý přenos rotaviru od skotu na člověka byl prokázán v Izraeli, kde bovinní RVA genotypu G6P[1] způsobil akutní gastroenteritidu u ročního chlapce [11]. Ve studii mapující diverzitu rotavirových kmenů cirkulujících mezi dětmi mladšími 5 let v Dánsku byla u 1 % vzorků zjištěna přítomnost vzácných genotypů, jako např. G6P[14] a G6P[5]. Předpokládá se, že původní hostitelé genotypu P[14] byly kopytníci (ovce), zatímco genotyp G6P[5] bývá běžně nalézán u dánského skotu [30].
Lidské rotaviry genotypů G9 a G12, které se začaly s vyšší frekvencí objevovat v poslední dekádě, mají původ pravděpodobně u zvířecích druhů. Sekvence genu kódujícího povrchový antigen VP7 genotypů G9 a G12 bývají běžně nalézány u selat [18, 19]. Tyto novější genotypy G9 a G12 nyní cirkulují u lidí na všech kontinentech, v některých epidemiologických studiích jsou dokonce RVA kmeny G9 převažujícím genotypem [43].
Rotaviry skupiny C, ačkoliv způsobují přibližně 1–10 % gastroenteritid virové etiologie, nejsou běžně diagnostikovány. Také množství celogenomově sekvenovaných kmenů RVC je výrazně menší, než je tomu u RVA. Informací o potenciálně zoonotických RVC kmenech je proto méně. Nicméně bylo prokázáno, že prasečí RVC kmeny jsou schopné způsobit infekci u člověka [15]. O pravděpodobném zoonotickém potenciálu RVC se již dříve diskutovalo na základě výsledků séroepidemiologického průzkumu ve Velké Británii, kdy byla zjištěna vyšší prevalence protilátek proti RVC ve venkovských oblastech ve srovnání s oblastmi městskými [20].
ZÁVĚR
Důkazy potvrzující mezidruhový přenos a genetický reasortment mezi lidskými a zvířecími rotaviry (zvláště RVA) se v odborné literatuře objevují stále častěji. Je zřejmé, že především některé živočišné druhy, jako jsou prasata, krávy, kočky a psi, se kterými se lidé nacházejí v těsnějším kontaktu, běžně přispívají k rozšiřování obrovské antigenní a genetické rozmanitosti lidských rotavirů. Nové kmeny RVA vzniklé reasortmentem, a tak nesoucí smíšené genomy (RNA segmenty pocházející jak z lidských, tak ze zvířecích rotavirů), se zdají být při šíření v lidské populaci úspěšnější než čistě heterologní kmeny, které obsahují všechny genomové segmenty jen ze zvířecích virů. Úspěšné šíření těchto nově vzniklých kmenů může být usnadněno nulovou specifickou imunitou populace proti nově introdukovaným antigenům.
Je zcela nezbytné provádět souvislé epidemiologické sledování rotavirů na molekulární úrovni, neboť nově se objevující kmeny by v budoucnu mohly snižovat účinnost používaných vakcín. Ekologie rotavirů by mohla být také ovlivněná probíhající vakcinací, nicméně dosud nebyly pozorovány žádné zásadní změny v prevalenci jednotlivých RVA genotypů, které by mohly být způsobeny použitými vakcinačními kmeny.
Poděkování
Tato práce byla připravena za finanční podpory od Ministerstva školství, tělovýchovy a sportu České Republiky (projekt LO1218 v rámci programu NPU I).
Střet zájmů
Autorky prohlašují, že nedošlo k žádnému střetu zájmů.
Do redakce došlo dne 28. 11. 2014.
Adresa pro korespondenci:
Mgr. Romana Moutelíková
Hudcova 70
621 00 Brno
e-mail: moutelikova@vri.cz
Sources
1. Ahmed MU, Kobayashi N, Wakuda M, et al. Genetic analysis of group B human rotaviruses detected in Bangladesh in 2000 and 2001. J Med Virol, 2004;72 : 149–155.
2. Agócs MM, Serhan F, Yen C, et al. WHO global rotavirus surveillance network: a strategic review of the first 5 years, 2008–2012. Morb Mortal Wkly Rep, 2014;63(29):634–637.
3. Attoui H, Mertens PPC, Becnel J, et al. Reoviridae. In King AMQ, Adams MJ, Carstens EB, Lefkowitz EJ. Virus Taxonomy: Classification and Nomenclature of Viruses: Ninth Report of the International Committee on Taxonomy of Viruses. Amsterdam: Elsevier Academic Press;2012;541–637.
4. Aung TS, Kobayashi N, Nagashima S, et al. Detection of group B rotavirus in an adult with acute gastroenteritis in Yangon, Myanmar. J Med Virol, 2009;81 : 1968–1974.
5. Azavedo MS, Yuan L, Jeong KI, et al. Viremia and nasal and rectal shedding of rotavirus in gnotobiotic pigs inoculated with Wa human rotavirus. J Virol, 2005;79 : 5428–5436.
6. Bailey KE, Gilkerson JR, Browning GF. Equine rotaviruses – Current understanding and continuing challenges. Vet Microbiol, 2013;167(1-2):135–144.
7. Bányai K, Bogdán Á, Domonkos G, et al. Genetic diversity and zoonotic potential of human rotavirus strains, 2003-2006, Hungary. J Med Virol, 2009;81 : 362–370.
8. Bányai K, Jiang B, Bogdán Á, et al. Prevalence and molecular characterization of human group C rotaviruses in Hungary. J Clin Virol, 2006;37 : 317–322.
9. Bishop RF, Davidson GP, Holmes IH, Ruck BJ. Virus particles in epithelial cells of duodenal mucosa from children with acute non-bacterial gastroenteritis. Lancet, 1973;2(7841):1281–1283.
10. Blanchard PC. Diagnostics of dairy and beef cattle diarrhea. Vet Clin Food Anim, 2012;28(3):443–464.
11. Doan YH, Nakagomi T, Aboudy Y, et al. Identification by full-genome analysis of a bovine rotavirus transmitted directly to and causing diarrhea in a human child. J Clin Microbiol, 2013;51 : 182–189.
12. Doležel J, Pazdiora P. Kolik v ČR stojí hospitalizace pro rotavirové gastroenteritidy [online]. Medical Tribune CZ 26/2013 [cit. 2015-01-16]. Dostupný na www: <http://www.tribune.cz/clanek/31812>.
13. Esona MD, Humphrey CD, Dennehy PH, et al. Prevalence of group C rotavirus among children in Rhode Island, United States. J Clin Microbiol, 2008;42 : 221–224.
14. Estes MK. Rotaviruses and their replication. In: Knipe DM, Howley PM, Griffin DE, et al. Fields Virology. 4th ed. Philadelphia: Lipincott William and Wilkins;2001; 1747–1785.
15. Gabbay YB, Borges AA, Oliveira DS, et al. Evidence for zoonotic transmission of group C rotaviruses among children in Belém, Brazil. J Med Virol, 2008;80 : 1666–1674.
16. Ghosh S, Kobayashi N. Whole-genomic analysis of rotavirus strains: current status and future prospects. Future Microbiol, 2011;6(9):1049–1065.
17. Ghosh S, Urushibara N, Taniguchi K, Kobayashi N. Whole genomic analysis reveals the porcine origin of human G9P[19] rotavirus strains Mc323 and Mc345. Infect Genet Evol, 2012;12 : 471–477.
18. Ghosh S, Varghese V, Samajdar S, et al. Molecular characterization of a porcine Group A rotavirus strain with G12 genotype specificity. Arch Virol, 2006;151 : 1329–1344.
19. Hoshino Y, Honma S, Jones RW, et al. A porcine G9 rotavirus strain shares neutralization and VP7 phylogenetic sequence lineage 3 characteristics with contemporary human G9 rotavirus strains. Virology, 2005;332 : 177–188.
20. Iturriza-Gómara M, Clarke I, Desselberger U, et al. Seroepidemiology of group C rotavirus infection in England and Wales. Eur J Epidemiol, 2004;19 : 589–595.
21. Iturriza-Gómara M, Dallman K, Bányai K, et al. Rotavirus genotypes co-circulating in Europe between 2006 and 2009 as determined by EuroRotaNet, a pan-European collaborative strain surveillance network. Epidemiol Infect, 2011;139(6):895–909.
22. Kobayashi N, Alam MM, Kojima K, et al. Genomic diversity and evolution of rotaviruses: an overview. In: Genomic Diversity and Molecular Epidemiology of Rotaviruses. Kobayashi N (ed). Research Signpost, Trivandrum, India;2003; 75–89.
23. Martella V, Bányai K, Lorusso E, et al. Genetic heterogeneity in the VP7 of group C rotaviruses. Virology, 2007;367 : 358–366.
24. Marthaler D, Rossow K, Culhane M, et al. Identification, phylogenetic analysis and classification of porcine group C rotavirus VP7 sequences from the United States and Canada. Virology, 2013;446 : 189–198.
25. Marthaler D, Rossow K, Gramer M, et al. Detection of substantial porcine group B rotavirus genetic diversity in the United States, resulting in a modified classification proposal for G genotypes. Virology, 2012;433 : 85–96.
26. Matthijnssens J, Ciarlet M, Rahman M, et al. Recommendations for the classification of group A rotaviruses using all 11 genomic RNA segments. Arch Virol, 2008;153 : 1621–1629.
27. Matthijnssens J, Otto PH, Ciarlet M, et al. VP6-sequence-based cutoff values as a criterion for rotavirus species demarcation. Arch Virol, 2012;157 : 1177–1182.
28. Meleg E, Bányai K, Martella V, et al. Detection and quantification of group C rotaviruses in communal sewage. Appl Environ Microbiol, 2008;74 : 3394–339.
29. Midgley SE, Bányai K, Buesa J, et al. Diversity and zoonotic potential of rotaviruses in swine and cattle across Europe. Vet Microbiol, 2012;156 : 238–245.
30. Midgley S, Böttiger B, Jensen TG, et al. Human group A rotavirus infections in children in Denmark; detection of reassortant G9 strains and zoonotic P[14] strains. Infect Genet Evol, 2014;27 : 114–120.
31. Moutelíková R, Prodělalová J, Dufková L. Prevalence study and phylogenetic analysis of group C porcine rotavirus in the Czech Republic revealed a high level of VP6 gene heterogeneity within porcine cluster I1. Arch Virol, 2014;159(5):1163–1167.
32. Moutelíková R, Bendová J, Prodělalová J. Detection of group B and C porcine rotaviruses in the Czech Republic. Veterinářství, 2012;2 : 87–89.
33. Mukherjee A, Ghosh S, Bagchi P, et al. Full genomic analyses of human rotavirus G4P[4], G4P[6], G9P[19] and G10P[6] strains from North-Eastern India: evidence for interspecies transmission and complex reassortment events. Clin Microbiol Infect, 2011;17 : 1343–1346.
34. Mukherjee A, Mullick S, Deb AK, et al. First report of human rotavirus G8P[4] gastroenteritis in India: evidence of ruminants-to-human zoonotic transmission. J Med Virol, 2013;85(3):537–545.
35. Nagashima S, Kobayashi N, Ishino M, et al. Whole genomic characterization of a human rotavirus strain B219 belonging to a novel group of the genus Rotavirus. J Med Virol, 2008;80 : 2023–2033.
36. Nilsson M, Svenungsson B, Hedlund K-O, et al. Incidence and genetic diversity of group C rotavirus among adults. J Infect Dis, 2000;182 : 678–684.
37. Ng TFF, Mesquita JR, Nascimento MSJ, et al. Feline fecal virome reveals novel and prevalent enteric viruses. Vet Microbiol, 2014;171 : 102–111.
38. Otto PH, Ahmed MU, Hotzel H, et al. Detection of avian rotaviruses of groups A, D, F and G in diseased chickens and turkeys from Europe and Bangladesh. Vet Microbiol, 2012;156 : 8–15.
39. Parashar UD, Hummelman EG, Bresee JS, et al. Global illness and deaths caused by rotavirus disease in children. Emerg Infect Dis, 2003;9 : 565–72.
40. Pazdiora P. Current rotavirus infections, options of prevention through vaccination. Postgraduální medicína, 2011;9 : 964–967.
41. Pazdiora P, Beneš Č. Rotavirus gastroenteritis in the Czech Republic before the start of vaccination. Epidemiol Mikrobiol Imunol, 2013;4 : 131–137.
42. Phan TG, Nishimura S, Okame M, et al. Virus diversity and an outbreak of group C rotavirus among infants and children with diarrhea in Maizuro City, Japan, during 2002-2003. J Med Virol, 2004;74 : 173–179.
43. Rahman M, Matthijnssens J, Goegebuer T, et al. Predominance of rotavirus G9 genotype in children hospitalized for rotavirus gastroenteritis in Belgium during 1999-2003. J Clin Virol, 2005;33 : 1–6.
44. Rodger SM, Bishop RF, Holmes IH. Detection of a rotavirus-like agent associated with diarrhea in an infant. J Clin Microbiol, 1982;16 : 724–726.
45. Saif LJ, Bohl EH, Theil KW, et al. Rotavirus-like, calicivirus-like, and 23-nm virus-like particles associated with diarrhea in young pigs. J Clin Microbiol, 1980;12 : 105–111.
46. Saif LJ, Jiang B. Nongroup A rotaviruses of humans and animals. Curr Top Microbiol Immunol, 1994;185 : 339–371.
47. Sánchez-Fauquier A, Roman E, Colomina J et al. First detection of group C rotavirus in children with acute diarrhea in Spain. Arch Virol, 2003;148 : 399–404.
48. Silva LC, Sanches AA, Gregori F, et al. First description of group A rotavirus from fecal samples of ostriches (Struthio camelus). Res Vet Sci, 2012;93 : 1066–1069.
49. Vesikari T. Trials of oral bovine and rhesus rotavirus vaccines in Finland: a historical account and present status. Arch Virol, 1996;12 : 177–186.
50. Wang Y-H, Kobayashi N, Nagashima S, et al. Full genomic analysis of a porcine-bovine reassortant G4P[6] rotavirus strain R479 isolated from an infant in China. J Med Virol, 2010;82 : 1094–1102.
51. Yang H, Makeyev EV, Kang Z, et al. Cloning and sequence analysis of dsRNA segments 5, 6 and 7 of a novel non-group A, B, C adult rotavirus that caused an outbreak of gastroenteritis in China. Virus Res, 2004;106 : 15–26.
52. Yuan L, Stevenson GW, Saif LJ. Rotavirus and reovirus. In: Straw BE, Zimerman JJ, D´Alaire S, Taylor DJ. Diseases of Swine. 9th ed. Ames, IA: Blackwell Publishing; 2006 : 435–454.
53. Zeller M, Heylen E, De Coster S, et al. Full genome characterization of a porcine-like human G9P[6] rotavirus strain isolated from an infant in Belgium. Infect Genet Evol, 2012;12 : 1492–1500.
54. Zhang Q, Hu R, Tang X, et al. Occurence and investigation of enteric viral infections in pigs with diarrhea in China. Arch Virol, 2013;158(8):1631–1636.
55. Ziemer CJ, Bonner JM, Cole D, et al. Fate and transport of zoonotic, bacterial, viral, and parasitic pathogens during swine manure treatment, storage, and land application. J Anim Sci, 2010;88:E84-E94.
Labels
Hygiene and epidemiology Medical virology Clinical microbiologyArticle was published in
Epidemiology, Microbiology, Immunology

2015 Issue 2
-
All articles in this issue
- Prevalence výskytu nosního nosičství kmenů Staphylococcus aureus a meticilin rezistentních kmenů S. aureus (MRSA) u studentů všeobecného lékařství LF UP v Olomouci
- Sekvenace enterovirů jako nový přístup k laboratorní diagnostice pro klinické i epidemiologické účely
- Toxoplasmóza u imunokompromitovaných pacientů
- Možnosti zoonotického přenosu rotavirových infekcí
- Hepatitida E – přehled současných poznatků
- Rizika šíření viru západonilské horečky v České republice
- Diagnostika infekcí vyvolaných Clostridium difficile v České republice – dostupnost, možnosti, interpretace laboratorních nálezů
- Prevalenční studie kmenů Escherichia coli a Klebsiella pneumoniae produkujících karbapenemázu v českých nemocnicích – výsledky české části studie EuSCAPE (European Survey on Carbapenemase-Producing Enterobacteriaceae)
- Epidemiology, Microbiology, Immunology
- Journal archive
- Current issue
- About the journal
Most read in this issue
- Možnosti zoonotického přenosu rotavirových infekcí
- Hepatitida E – přehled současných poznatků
- Prevalence výskytu nosního nosičství kmenů Staphylococcus aureus a meticilin rezistentních kmenů S. aureus (MRSA) u studentů všeobecného lékařství LF UP v Olomouci
- Toxoplasmóza u imunokompromitovaných pacientů
