-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Efektivní schéma využívané v diagnostice nádorů CNS
A rational approach to the CNS tumors diagnostics
The new WHO classification of CNS tumors is largely based on molecular diagnostic. Without molecular methods some entities can no longer be diagnosed. We are trying to show a rational approach to the CNS tumors diagnostics, which is based on conventional molecular methods such as RT-PCR, Sanger sequencing, MLPA, extended by the next generation sequencing (NGS) and methylation SNP array.
Keywords:
next generation sequencing (NGS) – RT-PCR – WHO classification – CNS tumors – MLPA – Methylation SNP array
Autori: Lenka Krsková 1; Barbora Šípalová 1; Tereza Němečková 1; Martina Strnadová 1; Tereza Kalendová 1; Alena Kalfusová 1; Alexandra; Malkusová 1; Monika Šandová 1; Miroslav Koblížek 1; Jan Balko 1; Aleš Vícha 2; Petr Brož 2,3; Pavla Jenčová 2; Lucie Štolová 2; Dagmar; Voříšková 2; Marie Belhajová 2; Michal Zápotocký 2; David Sumerauer 2; Josef Zámečník 1
Pôsobisko autorov: Ústav patologie a molekulární medicíny, 2. LF UK a FN Motol, Praha 1; Klinika dětské hematologie a onkologie, 2. LF UK a FN Motol, Praha 2; Bioxsys s. r. o., Ústí nad Labem 3
Vyšlo v časopise: Čes.-slov. Patol., 58, 2022, No. 3, p. 135-137
Kategória: Přehledový článek
Súhrn
Nová WHO klasifikace nádorů CNS se do značné míry opírá o molekulární diagnostiku, bez které již nelze některé jednotky prakticky diagnostikovat. Snažíme se ukázat racionální přístup k diagnostice nádorů CNS, který se opírá o konvenční molekulární metody jako je RT-PCR, Sangerovo sekvenování, MLPA, rozšířené o sekvenování nové generace (NGS) a metylační SNP array.
Klíčová slova:
sekvenování nové generace (NGS) – RT-PCR – WHO klasifikace – nádory CNS – MLPA – Metylační SNP array
Nová WHO klasifikace nádorů CNS se ve značném rozsahu opírá o molekulární diagnostiku, bez které již nelze některé jednotky prakticky diagnostikovat (1,2). Z tohoto důvodu vyplývá i důraz na racionální nicméně maximálně efektivní molekulární diagnostiku těchto nádorů.
Na našem pracovišti ve FN Motol máme již několik let zavedenou molekulární diagnostiku nádorů CNS, kdy velmi těsně spolupracuje na molekulární diagnostice Laboratoř molekulární patologie Ústavu patologie a molekulární medicíny s Laboratoří biologie solidních nádorů Kliniky dětské hematologie a onkologie. Společná diagnostika se opírá o morfologický nález, který nasměruje molekulárního patologa k tomu, co racionálně vyšetřovat. Důraz klademe na údaj o procentu nádorových buněk ve vyšetřované tkáni, což je údaj pro molekulární vyšetření zcela nezbytný, jelikož se od něj odvíjí limitace řady molekulárních metod (viz Tabulka č. 1).
Tab. 1. Molekulární metody a jejich limitace. 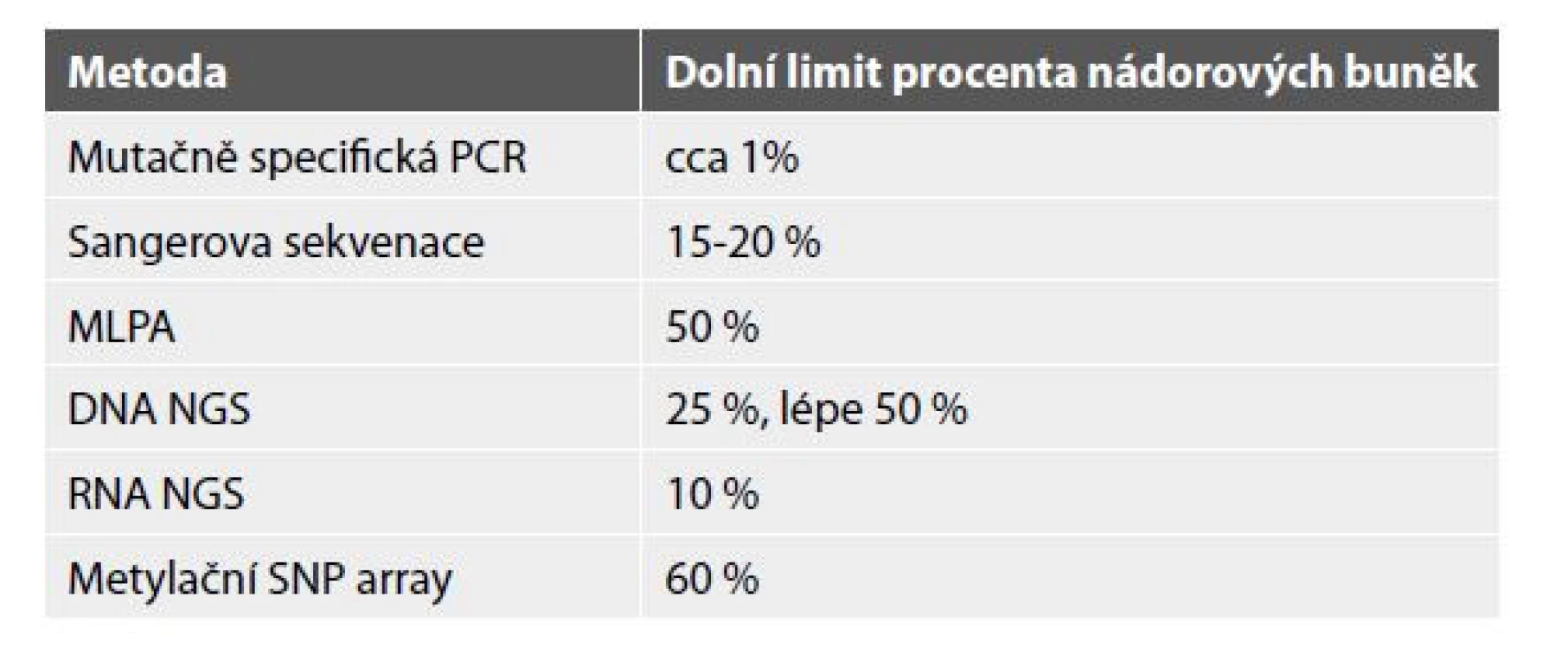
Dále se diferenciálně diagnosticky propojují data ze zobrazovacích metod spolu s klinickými údaji, která např. napoví, kde je daný nádor lokalizovaný, zda se nádor sytí po podání kontrastní látky na zobrazení MRI, či zda je v nádoru cystická komponenta apod., a tím může upřesnit další molekulární vyšetření.
U low-grade gliomů (LGG) CNS tedy k diagnostice přistupujeme tak, že vzorek nejprve vyšetříme pomocí PCR a popř. Sangerovy sekvenace a hlavně RT-PCR molekulární změny, které bychom u daného nádoru v dané lokalizaci očekávali. Například u LGG zadní jámy lební vyšetřujeme nejčastěji se vyskytující fúzní gen KIAA1549::BRAF. Oproti tomu z našich zkušeností víme, že začít vyšetřování fúze KIAA1549::BRAF u nádoru stejné morfologie v hemisferální oblasti, kde se setkáváme s celou řadou jiných a velmi často netradičních fúzních partnerů genu BRAF, nemá tato koncepce kýžený přínos.
Nicméně velmi stručně a zjednodušeně u LGG jiných lokalizací než hemisferálních primárně většinou vyšetřujeme fúzní gen KIAA1549::BRAF spolu s mutacemi genu BRAF a v případě neúspěchu dle morfologie pokračujeme s vyšetřením interní tandemové duplikace FGFR1 (ITD FGFR1) u diagnózy dysembryoplastického neuroepiteliálního nádoru (DNET), mutace genu BRAF či FGFR2 fúzní geny u suspektní diagnózy polymorfního low-grade neuroepiteliálního nádoru mladých (PLNTY), apod. V tomto úseku diagnostiky hraje ústřední roli ladění molekulární diagnostiky právě s morfologickým nálezem a s pohledem klinického onkologa. V případě negativních výsledků volíme dvě varianty a to vyšetření pomocí multiplexní na ligaci závislé PCR reakce (MLPA), která nás může efektivně nasměrovat k odhalení správné molekulární změny, např. k fúznímu genu SRGAP::RAF1, k fúznímu genu FGFR1::TACC1, apod. Nicméně čím dál častěji volíme v rámci zrychlení celého procesu druhou variantu a tou je sekvenování nové generace (NGS) k odhalení fúzních genů (3). U nádorů s negativním molekulárním nálezem a/nebo u nádorů s neobvyklým molekulárním nálezem (např. fúzní gen GOPC::ROS1 u LGG, různé fúze genů NTRK, či FGFR) volíme metylační SNP array k doplnění a k odhalení dalších molekulárních změn jako je určení CNV (Copy Number Variation neboli delece, či zisk celého nebo části chromozómu) a zařazení nádoru dle jeho metylačního profilu (4,5).
V rámci diagnostiky high-grade gliomů (HGG) se v korelaci s věkovým zasazením, lokalizací nádoru apod. zaměřujeme u pediatrických pacientů na molekulární diagnostiku mutací genu H3-3A (H3F3A) a to mutací K27M, či G34, popř. mutace genu BRAF a genů IDH rozšířené v dalším kroku o NGS s cílem najít potenciální terapeutický cíl. U pediatrických pacientů je rovněž standardem vyšetření pomocí metylační SNP array. Infantilní typ je novým typem high-grade gliomu, který se vyskytuje u novorozenců a kojenců a má odlišný molekulární profil s fúzními geny zahrnujícími ALK, ROS1, NTRK1/2/3 nebo MET (6,7), který se pro širokou škálu fúzních genů standartně vyšetřuje pomocí NGS a metylační SNP array. U dospělých pacientů se standartně vyšetřují mutace genů IDH (IDH1 a IDH2) spolu s promotorovou oblastí genu TERT rozšířenou o deleci CDKN2A, amplifikaci genu EGFR a detekci chromozomálních změn +7/-10 (2).
Oproti tomu v diagnostice meduloblastomů primárně vyšetřujeme mutaci genu CTNNB1 (WNT podtyp meduloblastomů) a mutaci v promotorové oblasti genu TERT, kterou často nacházíme u SHH typu meduloblastomů. Paralelně s tímto vyšetřením probíhá metylační profilování, které v současnosti společně s profilováním pomocí exprese mRNA jako jediné metody jednoznačně určí daný typ meduloblastomu i s jeho podtypy. Dále nám analýza CNV z metylační array pomůže odhalit prognostické alterace jako MYC a MCN amplifikace, ztrátu 6. chromosomu a i17q. To je zásadní pro stratifikaci léčby pacientů s meduloblastomem a určení jejich prognózy.
V rámci diagnostiky ependymomů opět u pediatrických pacientů probíhá standardně metylační profilování k určení biologické podskupiny onemocnění. U supratentoriálních ependymomů vyšetřujeme fúzi ZFTA::C11orf95 (dříve RELA::C11orf95). V případě negativního nálezu, popř. po zařazení nádoru na základě metylačního profilu do skupiny supratentoriálních ependymomů s YAP1 alterací, vyšetřujeme fúzi YAP1::MAMLD1 a další fúze pomocí RT-PCR či pomocí NGS.
V rámci dalších nicméně raritních nádorů CNS využíváme obecně metylační SNP array spolu se sekvenováním nové generace ať už na úrovni DNA a/nebo RNA panelů.
Závěrem lze shrnout, že diagnostika CNS nádorů je v současné době proces vysoce komplexní, kde dochází k propojení mnoha metod počínaje metodami zobrazovacími, morfologickými, imunohistochemickými spolu s širokým spektrem metod molekulárních doplněných o pohled klinického onkologa, neurochirurga apod. Výsledkem by měla být co nejpřesnější diagnóza, která umožní stanovit prognózu onemocnění a v optimálním případě nalézt vhodný terapeutický cíl.
KAZUISTIKA
V následující kazuistice udáváme příklad molekulární diagnostiky pacientky s LGG.
K histologickému i molekulárnímu vyšetření jsme dostali biopsii tříleté dívky se středočárovým nádorem v oblasti mozkového pedunklu a thalamu vlevo s extenzí do mozkového kmene. Dle peroperační biopsie se jednalo o low-grade gliom. Nádor byl z morfologického hlediska středně buněčný. Přítomné byly oblasti z kompaktně uspořádaných protáhlých astrocytů s početnými fibrilami v intersticiu. Dále byly přítomné oblasti z drobných monotónních buněk s kulatým jádrem vzhledu oligodendroglie. Nádorové buňky byly bez atypií, mitotická aktivita ani nekróza nebyly prokázány.
Nádorové buňky byly pozitivní v imunohistochemickém průkazu GFAP. Imunohistochemický průkaz IDH1 a CD34 byl v nádorových buňkách negativní. Podobně byl negativní i průkaz neuronálních markerů (neuN, synaptofyzin, neurofilamenta). Proliferační aktivita nádorových buněk stanovená imunohistochemickým průkazem jaderného antigenu Ki-67 byla nízká, pozitivních bylo méně než 2-3 % jader nádorových buněk.
Nádor byl pomocí RT-PCR vyšetřen na fúzní gen KIAA1549::BRAF s negativním výsledkem, mutační analýza exonu 15 genu BRAF a exonů 12 a 14 genu FGFR1 neprokázala patogenní variantu vyšetřovaných genů. Dále jsme vyšetřili tandemovou duplikaci genu FGFR1 rovněž s negativním výsledkem. V extraktu DNA ze vzorku tkáně jsme nedetekovali mutaci genu H3-3A (H3F3A).
Vzorek byl dále vyšetřen pomocí NGS s použitím Archer FusionPlex Lung kitu, kde byl prokázán fúzní gen GOPC::ROS1 s fúzí exonu 8 genu GOPC s exonem 35 genu ROS1 (obr. 1A). Tato fúze byla následně potvrzena i pomocí RT-PCR a následné sekvenace (obr. 1B).
Obr. 1. A: Průkaz fúzního genu GOPC::ROS1 pomocí Archer Fusion Plex NGS u dětské pacientky s LGG. Analýza pomocí Arriba software https:// github.com/suhrig/arriba. B: Sekvenační analýza fúzního produktu GOPC::ROS1 pomocí Sangerovy sekvenace. 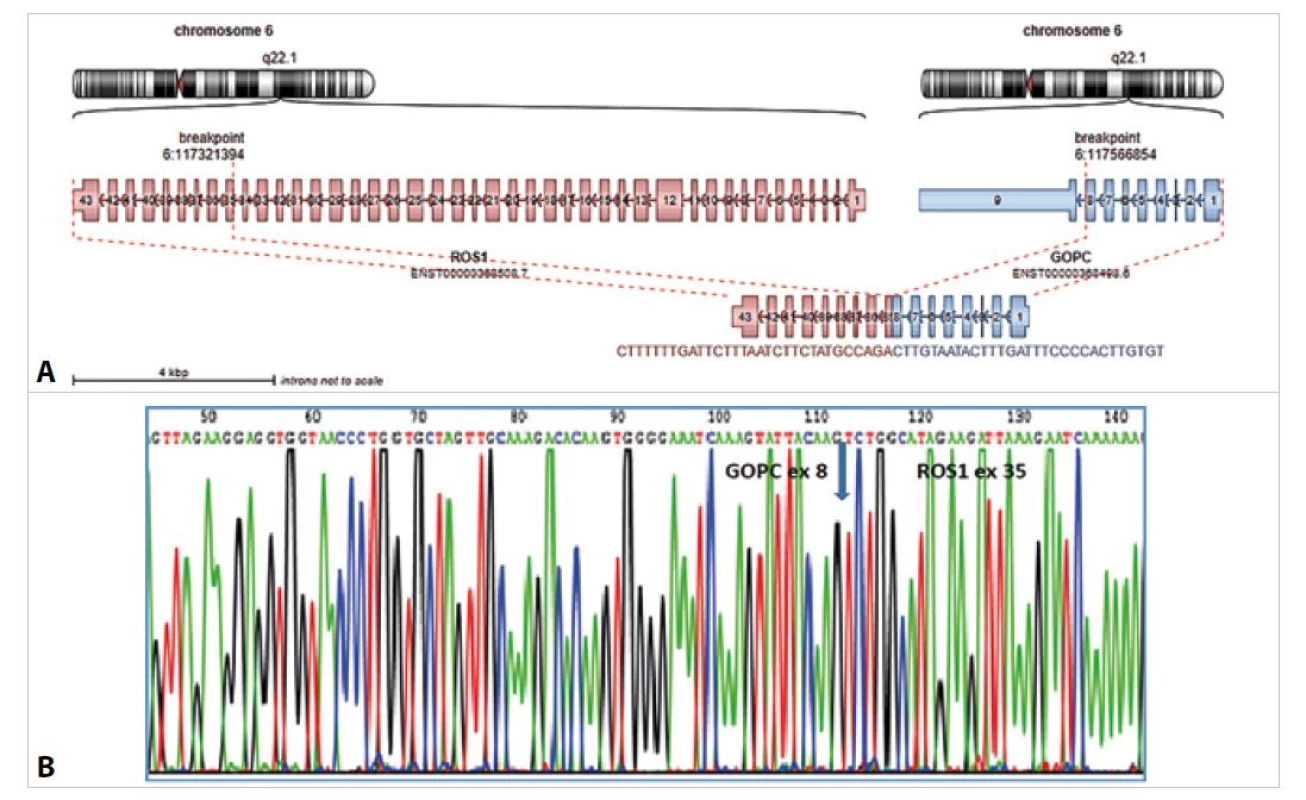
Nádor jsme klasifikovali jako low grade gliom s prokázanou fúzí GOPC::ROS1, podle proliferační aktivity WHO grade I.
Pro neobvyklý molekulární nález jsme u pacientky provedli metylační SNP array, která tumor zařadila jako Low grade glioma, pilocytic astrocytoma subtype, midline s jistotou 0,984 (pro jistotu zařazení je nutné dosáhnout hodnoty 0,9, což zde bylo splněno). Ve vyšetřovaném tumoru nebyly prokázány chromozomální změny, včetně změn které by upozornily na přítomnost fúze GOPC::ROS1. Nález z metylačního profilování byl ve shodě se stanovenou diagnózou low grade gliomu.
V současné době je pacientka pro progresi nádoru indikovaná k cílené terapii Rozlytrekem.
PROHLÁŠENÍ
Autor práce prohlašuje, že v souvislosti s tématem, vznikem a publikací tohoto článku není ve střetu zájmů a vznik ani publikace článku nebyly podpořeny žádnou farmaceutickou firmou. Toto prohlášení se týká i všech spoluautorů.
Adresa pro korespondenci:
RNDr. Lenka Krsková, Ph.D.
Ústav patologie a molekulární medicíny 2. LF UK a FN Motol
V Úvalu 84
Praha 5, 15006
Tel: 224435634
Fax: 224435620
Zdroje
1. Louis DN, Perry A, Wesseling P, et al. The 2021 WHO Classification of Tumors of the Central Nervous System: a summary. Neuro Oncol 2021; 23(8): 1231-1251.
2. IARC Publications Website - Central Nervous System Tumours. Accessed March 30, 2022.
3. Krsková L, Kalinová M, Němečková T, et al. Next generation sequencing - a science tool or routine pathology? Cesk Patol 2021; 57(3): 136-143.
4. Vícha A, Štolová L, Jenčová P, et al. Methylation pattern in the diagnosis and prognosis of brain cancer. Cesk Patol 2021; 57(3): 154-160.
5. Capper D, Stichel D, Sahm F, et al. Practical implementation of DNA methylation and copy-number-based CNS tumor diagnostics: the Heidelberg experience. Acta Neuropathol 2018; 136(2): 181-210.
6. Clarke M, Mackay A, Ismer B, et al. Infant High-Grade Gliomas Comprise Multiple Subgroups Characterized by Novel Targetable Gene Fusions and Favorable Outcomes. Cancer Discov 2020; 10(7): 942-963.
7. Guerreiro Stucklin AS, Ryall S, Fukuoka K, et al. Alterations in ALK/ROS1/NTRK/MET drive a group of infantile hemispheric gliomas. Nat Commun 2019; 10(1): 1-13.
Štítky
Patológia Súdne lekárstvo Toxikológia
Článek 'NEUROPATOLOGIEČlánek 'NEFROPATOLOGIEČlánek 'HEPATOPATOLOGIEČlánek 'ORTOPEDICKÁ PATOLOGIEČlánek 'KARDIOPATOLOGIEČlánek 'HEMATOPATOLOGIEČlánek 'CYTODIAGNOSTIKAČlánek 'PATOLOGIE GITČlánek 'PATOLOGIE ORL OBLASTIČlánek 'PULMOPATOLOGIEČlánek 'UROPATOLOGIEČlánek Molekulárně patologické profilování vybraných nádorů centrálního nervového systému metodou MLPAČlánek Mukormykóza: kazuistika
Článok vyšiel v časopiseČesko-slovenská patologie

2022 Číslo 3-
Všetky články tohto čísla
- Novinky ve WHO klasifikaci nádorů CNS 2021
- … obor paleontologie se v roce 1989 otevíral jenom v Leningradě …
- 'NEUROPATOLOGIE
- 'NEFROPATOLOGIE
- 'HEPATOPATOLOGIE
- 'ORTOPEDICKÁ PATOLOGIE
- 'KARDIOPATOLOGIE
- 'HEMATOPATOLOGIE
- 'CYTODIAGNOSTIKA
- 'PATOLOGIE GIT
- 'PATOLOGIE ORL OBLASTI
- 'PULMOPATOLOGIE
- 'UROPATOLOGIE
- Novinky ve WHO klasifikaci nádorů centrálního nervového systému 2021
- Efektivní schéma využívané v diagnostice nádorů CNS
- Molekulárně patologické profilování vybraných nádorů centrálního nervového systému metodou MLPA
- Nádory CNS – klinické a radiologické aspekty
- Obrovskobuněčný fibroblastom: kazuistika
- Mukormykóza: kazuistika
- Česko-slovenská patologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Novinky ve WHO klasifikaci nádorů centrálního nervového systému 2021
- Nádory CNS – klinické a radiologické aspekty
- Mukormykóza: kazuistika
- Obrovskobuněčný fibroblastom: kazuistika
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



