-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Analýza fosfoproteínov a signálnych dráh kvantitatívno‑ proteomickými metódami
Ananlysis of Phosphoproteins and Signalling Pathwaysby Quantitative Proteomics
Protein phosphorylation is a key regulator in cellular signaling pathways. It is involved in most cellular events in which interplay between phosphatases and kinases strictly controls biological processes, such as differentiation, proliferation and apoptosis. Altered or defective signaling pathways often result in various diseases, emphasizing the importance of studying the phosphoproteome. The abundance of phosphoproteins in the proteome is often very low, which requires specific and highly sensitive approaches. By using quantitative proteomics methods, we are able to analyze changes in abundance of proteins and their posttranslational modifications and then changes in signaling pathways. In this review, we describe quantitative proteomics methods, which could be used for study of phosphoproteins and their connection in signaling pathways.
Key words:
proteomics – phosphoproteins – signaling pathways
This work was supported by the European Regional Development Fund and the State Budget of the Czech Republic (RECAMO, CZ.1.05/2.1.00/03.0101) and by MH CZ – DRO (MMCI, 00209805).
The authors declare they have no potential conflicts of interest concerning drugs, products, or services used in the study.
The Editorial Board declares that the manuscript met the ICMJE “uniform requirements” for biomedical papers.Submitted:
30. 1. 2014Accepted:
14. 4. 2014
Autori: M. Pjechová 1; L. Hernychová 1; P. Tomašec 1,2; G. W. Wilkinson 1,2; B. Vojtěšek 1
Pôsobisko autorov: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno 1; Department of Medical Microbiology, Institute of Infection and Immunity, School of Medicine, Cardiff University, United Kingdom 2
Vyšlo v časopise: Klin Onkol 2014; 27(Supplementum): 116-120
Súhrn
Fosforylácia proteínov má kľúčovú úlohu v regulácii bunkových signálnych dráh. Je zahrnutá vo väčšine bunkových dejov, v ktorých súhra medzi kinázami a fosfatázami prísne kontroluje bunkové deje ako napr. proliferáciu, diferenciáciu a apoptózu. Chybné alebo pozmenené signálne dráhy sa mnohokrát podieľajú na vzniku rôznych chorôb, čo iba zdôrazňuje dôležitosť štúdia fosfoproteómu. Abundancia fosfoproteínov je v proteóme často veľmi nízka a na ich analýzu sú potrebné vysoko citlivé a špecifické prístupy. Metódami kvantitatívnej proteomiky je možné analyzovať zmeny v abundancii jednotlivých proteínov a ich posttranslačných modifikáciách a následne i v signálnych dráhach buniek. V tomto článku sa venujeme kvantitatívno ‑ proteomickým metódam, ktoré je možné použiť pri štúdiu fosfoproteínov a ich zapojení do signálnych dráh.
Kľúčové slová:
proteomika – fosfoproteíny – signálne dráhyÚvod
Fosforylácia proteínov je jedným z najrozšírenejších regulačných mechanizmov v prírode. Je kľúčovým regulátorom vnútrobunkových biologických procesov a momentálne najštudovanejšou posttranslačnou modifikáciou proteínov. Fosforyláciou je obvykle iniciovaná signálna transdukcia a šírené signály v signálnych dráhach buniek. Je to dočasná a reverzibilná posttranslačná modifikácia, ktorá vedie k zmenám konformácie, aktivity a interakcií proteínov vo veľmi krátkom časovom úseku. Proteínovú fosforyláciu riadia dve skupiny proteínov: kinázy (fosforylujú proteíny) a fosfatázy (defosforylujú proteíny). Často sú fosforylované viaceré reziduá proteínu, a to väčšinou rôznymi kinázami. Spomedzi aminokyselín sa u eukaryotov môže fosforylovať serín, treonín, tyrozín a histidín, ktorý je však veľmi labilný a zriedka identifikovaný vo fosfoproteomických štúdiách.
Prístupy kvantitatívnej proteomiky nám dovoľujú analyzovať zmeny v expresii proteínov (a fosfoproteínov) za rôznych podmienok a taktiež aj určiť zmenu fosforylácie jednotlivých proteínov, ktoré tvoria základnú funkčnú jednotku signálnych dráh.
V porovnaní s ostatnými proteínmi sú fosfoproteíny v bunkách prítomné vo veľmi nízkych množstvách, čo komplikuje ich detekciu hmotnostným spektrometrom a následné analýzy. Z tohto dôvodu sú potrebné techniky, ktoré umožňujú obohatenie vzoriek o fosfopeptidy (po proteolytickom štiepení proteínov na peptidy) pred hmotnostne spektrometrickou analýzou. „Strong cation exchange chromatography“ (SCX) [1], „hydrophilic interaction liquid chromatography“ (HILIC) [2], obohatenie fosfopeptidov oxidom titaničitým (TiO2) [3] či „immobilized metal ion affinity chromatography“ (IMAC) [1] patria medzi techniky pomocou ktorých sme schopní úspešne izolovať fosfopeptidy pred MS analýzou. Následná kvapalinová chromatografia v spojení s tandemovou MS analýzou (LC ‑ MS/ MS) je vhodný prístup, ktorý dovoľuje identifikáciu a kvantifikáciu tisícok fosforylačných miest počas jednej analýzy. Tieto, ale aj ďalšie metódy a postupy používané vo fosfoproteomických experimentoch budeme bližšie rozoberať v nasledujúcej časti článku.
Metódy kvantifikácie fosfoproteínov
Na kvantifikáciu zmien fosfoproteínov je možné použiť proteomické prístupy ako napr. „stable isotope labeling with amino acids in cell culture“ (SILAC) [4], ktorý sa dá použiť iba na živé bunkové kultúry. Jedná sa o metabolické značenie aminoyselín, pri ktorom v priebehu kultivácie dochádza k inkorporácii ťažkých aminokyselín (značené stabilné izotopy lyzínu a arginínu) do novosyntetizovaných proteínov. Pri tejto metóde je možné použiť ťažké izotopy lyzínu a arginínu (R10K8), stredne ťažké izotopy lyzínu a arginínu (R6K4) a neznačené, ľahké médium (R0K0).
Medzi izotopické značenia fosfopeptidov patria napr. ICAT, dimetylové a 18O značenie.
ICAT (isotope ‑ coded affinity tags), teda thiol ‑ reaktívne molekuly obsahujúce ťažké alebo ľahké izotopy, predstavuje jednu z najstarších techník [5]. ICAT značka pozostáva z reaktívnej skupiny, ktorá je orientovaná na cysteín, polymerového linkera s ôsmimi deutériami a biotínovej skupiny, ktorá dovoľuje purifikáciu značených peptidov. V ICAT experimente sú dva proteínové pooly denaturované a redukované, cysteínové reziduá proteínov sú následne značené ľahkým alebo ťažkým reagentom. Značené pooly sú potom zmiešané, prečistené od nadbytečného reagentu a štiepené vhodnou proteázou. Peptidy obsahujúce cysteín, nesúce ľahkú alebo ťažkú značku sú následne zachytené pomocou avidínovej kolóny, cez biotínovú skupinu na značke. Peptidy sú potom vyeluované z kolóny a analyzované na MS [6].
Pri dimetylovom značení sú proteíny najprv štiepené vhodnou proteázou, ako napr. trypsín, následne sú peptidy z rôznych vzoriek označené dimetylovými značkami a analyzované na LC/ MS, pričom rozdiel abundancie dimetylových značiek je použitý na porovnanie rozdielov v abundancii peptidov vo vzorkách. Toto značenie je založené na reakcii primárnych peptidových amínov (N ‑ konce peptidov a epsilonové amino skupiny lyzínových reziduí) s formaldehydom a vytvorení Schiff bázy, ktorá je rapídne redukovaná pridaním cyanoborohydridu. Dimetylové značenie je možné použiť ako triplex, kde je pri značení „ľahkou značkou“ použitý obyčajný formaldehyd a cyanoborohydrid, čo spôsobí zvýšenie hmotnosti o 28 Da na primárny amín v peptide. Použitie denaturovaného formaldehydu a obyčajného cyanoborohydridu spôsobí zvýšenie hmotnosti o 32 Da, čo je stredná značka [7]. Inkorporácia ťažkej značky je dosiahnutá kombináciou denaturovaného a 13C ‑ značeného formaldehydu s cyanoborodeuteridom, čo vedie k zvýšeniu hmotnosti o 36 Da [8].
Značenie pomocou 18O je relatívne jednoduchá, enzýmovo ‑ sprostredkovaná technika značenia peptidov pomocou H2 18O. Tento prístup je spojený s proteázami 2. triedy, napr. trypsínom, ktoré katalyzujú výmenu dvoch 16O atómov za dva 18O atómy na C‑terminálnej karboxylovej skupine proteolitického peptidu, čo vedie k posunu hmotnosti o 4 Da medzi 1 - krát nabitými rôzne značenými peptidmi v MS1 móde [6].
TMT (tandem mass tag) [9] a iTRAQ (the isobaric tag for relative and absolute quantitation) [10] sú izobarické značenia fungujúce na veľmi podobnom princípe. V porovnaní s tradičnými kvantifikačnými metódami sú schopné použitím tandemovej MS analýzy simultánne identifikovať a kvantifikovať 6 – 8 vzoriek. TMT molekula pozostáva z reaktívnej skupiny, ktorá značí primárne amínové skupiny; reportérovej skupiny, ktorá udáva v MS/ MS móde abundanciu peptidu po zmiešaní peptidov; odštiepiteľného linkeru, ktorý uvoľnuje TMT reporterový fragment v MS/ MS analýze a hmotnostne normalizačnej skupiny, ktorá vyrovnáva hmotnostné rozdiely medzi jednotlivými reportérovými fragmentami na udržanie konštantnej celkovej hmotnosti značiek v sete [11]. Keďže sa jedná o izobarické značky, kvantifikácia prevedená v MS/ MS móde porovnaním plôch píkov označených iónov vychádza z fragmentácie značky, teda rovnaké spektrum je použité na kvantifikáciu a identifikáciu fosfopeptidu [12].
Pri label free kvantifikácii sú proteínové zmesi analyzované priamo a vzorky sú navzájom porovnané až po samostatných analýzach. Pri tejto metóde nie sú vzorky zmiešané, teda je možné dosiahnuť vyššieho proteómového pokrytia a neexistuje žiaden limit v počte vzoriek, ktoré môžu byť porovnávané [13]. Nevýhodou tohto prístupu je neprítomnosť formálneho vnútorného štandardu, čo môže viesť k významným chybám v individuálnych datasetoch. Tento nedostatok je však minimalizovaný analýzou niekoľkých biologických replikátov [6].
Proteolytické štiepenie
Pred hmotnostnou analýzou je potrebné vzorky upraviť tak, aby boli kompatibilné s meraním na hmotnostnom spektrometri. Prvým krokom je štiepenie proteínov na peptidy použitím vhodnej proteázy. Na to, aby mohla proteáza proteíny efektívne štiepiť, je potrebná ich denaturácia, redukcia a alkylácia. Na denaturáciu sa používajú rôzne reagencie, napr. β-merkaptoetanol, SDS. Medzi najviac používané redukčné činidlo patrí dithiotreitol (DTT) alebo Tris (2 - carboxyethyl) phosphine (TCEP), ktoré redukujú disulfidové mostíky medzi cysteínovými skupinami. Nasleduje alkylácia thiolových skupín pomocou jódacetamidu (IAA), ktorý zabraňuje ich reoxidácii. Proteíny sú potom štepené proteázami na peptidy, ktoré je možné analyzovať na hmotnostnom spektrometri. Používajú sa proteázy s vysokou špecificitou štiepenia, aminokyselinové reťazce štiepia iba za určitými aminokyselinami. Jednou z najbežnejšie používaných proteáz je trypsín, ktorý štiepi reťazec aminokyselín na karboxylovej strane lyzínu a arginínu, ak za nimi nenasleduje prolín. Existuje viacero postupov, ako proteíny pripraviť na proteolytické štiepenie. Ich výber závisí na type a množstve vzorky, keďže pri niektorých postupoch sú starty vzoriek väčšie než pri iných.
Pred vlastným proteolytickým štiepením je potrebné odstrániť denaturačný roztok, ktorý nie je kompatibilný s proteolytickými enzýmami, a preto sa zamieňa za roztok močoviny. Pri „filter aided sample preparation“ (FASP) protokole sa tento krok deje na „cut off“ filtri (10 kDa, alebo 30 kDa), kde prebieha aj následná redukcia, alkylácia a vlastné proteolytické štiepenie. Medzi nevýhody tohto postupu patrí čiastočná strata vstupného materiálu na filtri [14].
Štiepenie v roztoku je postup, pri ktorom sa do skúmavky so vzorkou proteínov postupne pridávajú jednotlivé roztoky zaisťujúce denaturáciu, redukciu a alkyláciu (napr. močovina, DTT, IAA). Prebytok alkylačného činidla je následne odstránený pridaním redukčného činidla, aby sa v hmotnostnom spektre neobjavovali satelitné peptidové píky nesúce vzniknuté modifikácie zvyšujúce ich m/ z. Nakoniec sa pridáva proteáza a roztok NH4HCO3, ktorý je s ňou kompatibilný a poskytuje vhodné podmienky na štiepenie. V prípade tohto postupu vstupný materiál síce zostáva zachovaný, ale všetky pridané reagencie zostávajú v roztoku spolu so vzorkou a pred ďalším krokom je nutné získané peptidy purifikovať [15].
Pri štiepení proteínov v géli sa bunkový lyzát najprv rozseparuje na polyakrylamidovom géli. Zväčša sa používajú denaturačné 1D alebo 2D elektroforézy, ktoré separujú denaturované proteíny podľa molekulovej hmotnosti (proteíny s najvyššou hmotnosťou putujú v géli najpomalšie), čím zároveň umožňujú presné stanovenie molekulovej hmotnosti. Pri 1D elektroforéze sa proteíny separujú iba podľa molekulovej hmotnosti. Pri 2D elektroforéze sa proteíny rozdeľujú najprv podľa izoelektrického bodu, čo je pH, pri ktorom majú molekuly nulový náboj, a nepohybujú sa v elektrickom poli [16]. Nasleduje ich delenie v druhom smere podľa molekulovej hmotnosti. Najväčšou výhodou týchto postupov je prečistenie proteínov od nízkomolekulových nečistôt, ako napr. detergentov a zložiek pufrov, ktoré často nie sú kompatibilné s následnou MS analýzou. Na vizualizáciu proteínov sa používajú napr. trifenylmetánové farbivá Coomassie brilliant blue (R250, G250), ktoré sa viažu na proteíny prostredníctvom van der Waalesových a elektrostatických interakcií [17]. Najbežnejšie používanou je Coomassie brilliant blue G250, ktorá je schopná vizualizovať už 0,3 ng proteínu v géli. Komerčne sú dostupné aj fluorescenčné značky, napr. Sypro Ruby, Flamingo, či Deep Purple, ktoré majú senzitivitu okolo 1 ng detekovaného proteínu. Veľmi citlivým prístupom je farbenie striebrom, pomocou ktorého je možné vizualizovať 0,3 ng proteínu. Po farbení sú jednotlivé bandy vyrezané z gélu, odfarbené a proteíny štiepené proteázou. Nevýhoda tejto metódy spočíva v strate proteínov nerozpustných v SDS pufri a proteínov s nízkou molekulovou hmotnosťou [18].
Separácia peptidov pomocou kvapalinovej chromatografie
Na rozdelenie komplexných vzoriek peptidov získaných FASP protokolom alebo štiepením v roztoku, ako aj na odstránenie zložiek, ktoré môžu interferovať s ďalšími postupmi, je možné použiť kvapalinovú chromatografiu. High‑performance liquid chromatography (HPLC) môže byť použitá na separáciu peptidov na základe hydrofobicity, náboja alebo veľkosti. Kombináciou vhodných analytických kolón a gradientov je možné vybrať frakcie obsahujúce peptidy a fosfopeptidy a ďalej ich analyzovať. Vybrané typy kolón a ich vlastnosti budú popísané v ďalšej časti:
- Na negatívne nabitých funkčných skupinách kolóny strong cation exchange chromatography (SCX) sa pri pH 2,7 zachytávajú najmä 1 - krát fosforylované +1 kladne nabité fosfopeptidy. V eluovanej frakcii sa nachádzajú multifosforylované peptidy, preto je nutné eluát zachytiť [1].
- Hydrophilic interaction liquid chromatography (HILIC) je vysoko rozlišovacia separačná technika, pri ktorej sa využívajú vodíkové interakcie medzi peptidom a neutrálnou, hydrofilnou stacionárnou fázou. Retencia vzrastá so zvyšujúcou sa polaritou peptidu. Vzorky sú nanášané vo fáze s vysokým podielom organických zlúčenín a eluované zvyšujúcou sa polaritou mobilnej fázy [2].
- Electrostatic repulsion ‑ hydrophilic interaction chromatography (ERLIC) je separácia založená na anion exchange chromatography v kombinácii s hydrofilnými interakciami. Fosfátové skupiny sú čiastočne ionizované a elektrostaticky priťahované k stacionárnej fáze. Ako prvé sú eluované nefosforylované, monofosforylované a difosforylované peptidy, keďže multifosforylované peptidy obsahujú viacero negatívnych nábojov na fosfátových skupinách a eluujú sa neskôr [19].
Techniky obohacovania vzoriek o fosfopeptidy
Obohatenie fosfopeptidov je kľúčový krok v príprave vzoriek na identifikovanie fosforylovaných aminokyselín. Koncentrácia fosfoproteínov je v bunke v porovnaní s ostatnými proteínmi veľmi nízka a sú takmer nemerateľné dostupnými technikami hmotnostnej spektrometrie. Fosfopeptidy zle ionizujú, čo je dôsledkom prítomnosti fosfátovej skupiny, ktorá je záporne nabitá. Tým ovplyvňuje efektivitu ionizácie a detekcie pri kyslých podmienkach, ktoré sa používajú pri analýze v pozitívnom móde [20]. Fosfopeptidy bývajú v spektrách prekryté fragmentmi nefosforylovaných/ abundantnejších peptidových iónov, ktoré majú oveľa vyššiu intenzitu signálu. Na získanie/ obohatenie fosfopeptidov zo vzoriek bolo vyvinutých viacero metód, ktoré využívajú väzbu fosfátovej skupiny na matricu. Medzi najviac používané obohacovacie metódy patria obohatenie fosfopeptidov oxidom titaničitým (TiO2), immobilized metal ion affinity chromatography (IMAC) a sequentional elution from IMAC (SIMAC).
V prípade TiO2 (voľné častice s priemerom 10 µm) dochádza k väzbe fosfopeptidu cez fosfátové OH skupiny za kyslých podmienok. Uvoľnenie tejto väzby nastáva za zásaditých podmienok, pričom uvoľnené sú najmä monofosforylované peptidy (multifosforylované peptidy sa na TiO2 viažu s príliš vysokou afinitou a ich elúcia je veľmi obtiažna). Na TiO2 sa môžu viazať aj kyslé peptidy, čomu sa dá predísť znížením pH kyselinou trifluóroctovou alebo pridaním kyseliny dihydrobenzoovej, či mliečnej, ktoré sa na TiO2 viažu s vyššou afinitou než kyslé peptidy, ale s nižšou než fosfopeptidy [3].
Pri metóde IMAC sú ióny kovov (Fe3+, Al3+, Ga3+, Co2+) chelatované na častice pokryté nitrilotrioctovou alebo iminodioctovou kyselinou, čím vytvárajú stacionárnu fázu, na ktorú sa môžu naviazať negatívne nabité fosfopeptidy v mobilnej fáze. Táto metóda je vhodná hlavne pre obohatenie multifosforylovaných peptidov. Vážnym problémom tohto postupu je nešpecifická väzba nefosforylovaných peptidov obsahujúcich viacnásobne kyslé reziduá aminokyselín, čo znižuje selektivitu. Jednou z možností ako zvýšiť selektivitu tejto metódy je rovnako ako pri TiO2 zníženie pH [1].
SIMAC metóda kombinuje výhody TiO2 a IMAC prístupu. Keďže pri IMAC sú preferenčne naviazané multifosforylované peptidy a pri TiO2 monofosforylované, kombináciou týchto dvoch metód je možné dosiahnuť jednoduchého obohatenia mono ‑ i multifosforylovaných peptidov. IMAC je použitý ako prvý krok, kde po väzbe nasleduje elúcia monofosforylovaných peptidov za kyslých podmienok (pH 1,0) a potom elúcia multifosforylovaných peptidov za bázických podmienok (pH 11,3). Ďalším krokom je obohatenie monofosforylovaných peptidov TiO2 časticami [1].
Hmotnostne spektrometrická analýza
Peptidy (fosfopeptidy) je možné identifikovať pomocou MALDI ‑ MS (matrix ‑ aassisted laser desorption/ ionization – mass spectrometry, tj. hmotnostného spektrometru s ionizáciou pomocou laseru za účasti matrice) alebo prostredníctvom LC ‑ MS/ MS (liquid chromatography – mass spectrometry) s elektrosprejovou ionizáciou. Pri hlbších fosforylačných štúdiách je dôležité určiť špecifickú aminokyselinu, ktorá bola fosforylovaná (miesto fosforylácie) v danom biologickom experimente. Preto sa využíva tandemová MS analýza s collision‑induced dissociation (CID) a electron ‑ transfer dissociation (ETD) fragmentáciou. Pri CID fragmentácii dochádza k strate fosfátovej skupiny H3PO4 pri fosfoseríne a fosfotreoníne a HPO3 pri fosfotyrozíne. Nedostatkami tejto metódy sú nízka ionizačná efektivita a skorá strata labilnej fosfátovej skupiny, čo má za následok nízky počet peptidových fragmentov na identifikáciu. ETD štiepi peptidovú kostru bez odštiepenia labilných posttranslačných modifikácií, ako napr. fosforylácie. ETD má vyššiu pravdepodobnosť úspešnej fosfopeptidovej identifikácie pre prekurzory s nízkym m/ z a nábojovým stavom > 2, kým CID je úspešnejšia pre dvojnásobne nabité peptidy a tie s vysokým m/ z [21]. Niektoré štúdie porovnávajúce ETD a CID navrhujú využitie oboch metód fragmentácie na dosiahnutie komplexnej analýzy fosforylácie [21 – 23].
Analyzovanie dát
Kritickou súčasťou všetkých proteomických štúdií je analýza dát, pri ktorej sa využívajú bioinformatické a štatistické prístupy. Získané MS/ MS dáta sú porovnané s databázou Uniprot proteome [24] s použitím softwarov Mascot (Matrix Science Inc.) alebo Sequest [25]. Na spracovanie dát, prehľadanie databáz a kvantitatívne vyhodnotenie rozdielov medzi fosfoproteínmi v jednotlivých vzorkách je možné použiť program Proteome Discoverer 1.4 (Thermo Fisher Scientific) s algoritmom PhosphoRS 3.1 [26] umožňujúcim predikciu lokalizácie fosforylovaných miest. K dispozícii sú aj ďalšie podobné programy MaxQuant [27] a SkyLine [28].
Pri identifikovaných fosfoproteínoch je potrebné brať do úvahy zmenu fosforylácie oproti kontrolnej vzorke a celkovej expresii proteínu. Proteíny so signifikantnými zmenami fosforylácie môžu byť ďalej použité na štúdium signálnych dráh pomocou špecializovaných programov ako napr. Ingenuity Pathway Analysis (IPA) [29], David [30] alebo PathVisio [31]. Tieto programy sú schopné graficky znázorniť signálne dráhy na základe informácií získaných z databáz a vedeckých publikácií. Umožňujú tiež porovnanie jednotlivých dráh, a teda definovanie hlavných rozdielov medzi signálnymi dráhami v rôznych bunkách.
Záver
Na štúdium zmien v signálnych dráhach buniek je možné využiť proteomické prístupy. Kvantitatívna proteomika umožňuje určiť zmeny v signálnych dráhach zahŕňajúce zmeny v expresii proteínov, ako aj zmeny v posttranslačných modifikáciách (fosforyláciách), ktoré sú základným mechanizmom pri zapínaní/ vypínaní signálnych dráh. Vzhľadom na nízku abundanciu fosfoproteínov v bunkách a ich problematické zachytenie hmotnostným spektrometrom je správna voľba metód kľúčovým krokom (tab. 1.).
Tab. 1. Schéma postupu spracovania vzorky proteomickými metódami. 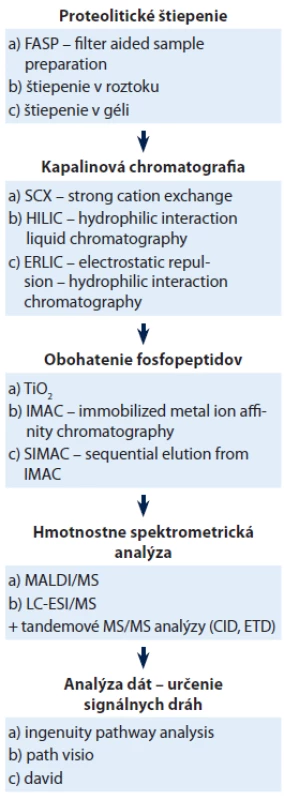
Nedostatky pri momentálne používaných postupoch poskytujú široký priestor na vylepšenie metód. Zamedzenie straty materiálu pri príprave vzoriek, či väzbe kyslých peptidov na častice pri obohacovacích technikách sú problémy, ktoré je nutné riešiť. Nemenej dôležitými sú i obmedzenia v MS analýze a bioinformatických aplikáciách. Rovnako aj zdokonaľovanie starých a vývoj nových metód a programov na analýzu môže rapídne prispieť k vylepšeniu detekcie a identifikácie proteínov.
Práca bola podporená Európskym fondom pre regionálny rozvoj a štátnym rozpočtom Českej republiky (OP VaVpI – RECAMO, CZ.1.05/2.1.00/03.0101) a MZ ČR – RVO (MOÚ, 00209805).
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
Mgr. Mariana Pjechová
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: mariana.pjechova@mou.cz
Obdržené: 30. 1. 2014
Přijaté:14. 4. 2014
Zdroje
1. Thingholm TE, Jensen ON, Larsen MR. Analytical strategies for phosphoproteomics. Proteomics 2009; 9(6): 1451 – 1468. doi: 10.1002/ pmic.200800454.
2. Alpert AJ. Hydrophilic ‑ interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. J Chromatogr 1990; 499 : 177 – 196.
3. Larsen MR, Thingholm TE, Jensen ON et al. Highly selective enrichment of phosphorylated peptides from peptide mixtures using titanium dioxide microcolumns. Mol Cell Proteomics 2005; 4(7): 873 – 886.
4. Oda Y, Huang K, Cross FR et al. Accurate quantitation of protein expression and site ‑ specific phosphorylation. Proc Natl Acad Sci USA 1999; 96(12): 6591 – 6596.
5. Gygi SP, Rist B, Gerber SA et al. Quantitative analysis of complex protein mixtures using isotope ‑ coded affinity tags. Nature Biotechnol 1999; 17 : 994 – 999.
6. Sap KA, Demmers JA (eds). Labeling Methods in Mass Spectrometry Based Quantitative Proteomics [monograph on the Internet]. Integrative Proteomics; 2012 [cited 2014 January 3]. Available from: http:/ / cdn.intechopen.com/ pdfs ‑ wm/ 29631.pdf.
7. Hsu JL, Huang SY, Chow NH et al. Stable ‑ isotope dimethyl labeling for quantitative proteomics. Anal Chem 2003; 75(24): 6843 – 6852.
8. Boersema PJ, Aye TT, van Veen TA et al. Triplex protein quantification based on stable isotope labeling by peptide dimethylation applied to cell and tissue lysates. Proteomics 2008; 8(22): 4624 – 4632. doi: 10.1002/ pmic.200800297.
9. Thompson A, Schafer J, Kuhn K et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/ MS. Anal Chem 2003; 75(8): 1895 – 1904.
10. Ross PL, Huang YN, Marchese JN et al. Multiplexed protein quantitation in saccharomyces cerevisiae using amine ‑ reactive isobaric tagging reagents. Mol Cell Proteomics 2004; 3(12): 1154 – 1169.
11. Licker V, Patel V, Ward M (eds). Characterisation of human cerebrospinal fluid (CSF) after tandem mass tag (TMT0) labelling [monograph on the Internet]. Universtité de Genéve; 2008 [cited 2014 January 3]. Available from: http:/ / www.mpb.unige.ch/ reports/ rap_VirginieLicker.pdf.
12. Nita ‑ Lazar A, Saito ‑ Benz H, White FM. Quantitative phosphoproteomics by mass spectrometry: past, present, and future. Proteomics 2008; 8(21): 4433 – 4443. doi: 10.1002/ pmic.200800231.
13. Bantscheff M, Schirle M, Sweetman G et al. Quantitative mass spectrometry in proteomics: a critical review. Anal Bioanal Chem 2007; 389(4): 1017 – 1031.
14. Wiśniewski JR, Zougman A, Nagaraj N et al. Universal sample preparation method for proteome analysis. Nat Methods 2009; 6(5): 359 – 362. doi: 10.1038/ nmeth.1322.
15. Btk.fi [homepage on the Internet]. Turku: Centre for Biotechnology, Finland [cited 2014 January 5]. Available from: http:/ / www.btk.fi/ fileadmin/ Page_files/ proteomics/ PDF ‑ files/ Protocols/ In_solution_trypsin_digestion.pdf.
16. Vgn.uvm.edu [homepage on the Internet]. Vermont Genetics Network. University of Vermont [cited 2014 January] Available from: http:/ / vgn.uvm.edu/ outreach/ documents/ Garfin_IEF_WebArticle9 – 07.pdf.
17. Nationaldiagnostics.com [homepage on the Internet]. Atlanta: National Diagnostics; Georgia; c2011 [cited 2014 January 5]. Available from: https:/ / www.nationaldiagnostics.com/ electrophoresis/ article/ staining ‑ protein‑gels ‑ coomassie ‑ blue.
8. Shevchenko A, Tomas H, Havlis J et al. In ‑ gel digestion for mass spectrometric characterization of proteins and proteomes. Nat Protoc 2006; 1(6): 2856 – 2860.
19. Alpert AJ. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Anal Chem 2008; 80(1): 62 – 76.
20. Steen H, Jebanathirajah JA, Rush J et al. Phosphorylation analysis by mass spectrometry. Mol Cell Proteomics 2006; 5(1): 172 – 181.
21. Swaney DL, McAlister GC, Coon JJ. Decision tree‑driven tandem mass spectrometry for shotgun proteomics. Nat Methods 2008; 5(11): 959 – 964. doi: 10.1038/ /nmeth.1260.
22. Molina H, Horn DM, Tang N et al. Global proteomic profiling of phosphopeptides using electron transfer dissociation tandem mass spectrometry. Proc Natl Acad Sci USA 2007; 104(7): 2199 – 2204.
23. Sweet SM, Bailey CM, Cunningham DL et al. Large scale localization of protein phosphorylation by use of electron capture dissociation mass spectrometry. Mol Cell Proteomics 2009; 8(5): 904 – 912. doi: 10.1074/ mcp.M800451 - MCP200.
24. Uniprot.org [homepage on the Internet] Uniprot, c2002 – 2014 [cited 2014 January 5]. Available from: http:/ / www.uniprot.org/ .
25. Eng JK, McCormack AL, Yates JR. Sequest algorithmpublication: an approach to correlate tandem mass spectral data of peptides with amino acid sequences in a protein database. J Am Soc Mass Spectrom 1994; 5(11): 976 – 989. doi: 10.1016/ 1044 - 0305(94)80016 - 2.
26. Taus T, Köcher T, Pichler P et al. Universal and confident phosphorylation site localization using phosphoRS. J Proteome Res 2011; 10(12): 5354 – 5362. doi: 10.1021/ pr200611n.
27. MaxQuant.org [homepage on the Internet]. MaxQuant. Max planck institute of biochemistry. Martinsried: Germany; c2013 [cited 2014 January 7]. Available from: http:/ / www.maxquant.org/ .
28. Skyline.gs.washington.edu [homepage on the Internet]. MacCoss Lab Software. Washington: University of Washington, Seattle; c2014 [cited 2014 January 7]. Available from: https:/ / skyline.gs.washington.edu/ labkey/ wiki/ home/ software/ Skyline/ page.view?name=defaude.
29. Ingenuity.com [homepage on the Internet]. Ingenuity. CA: Redwood City [cited 2014 January 7]. Available from: http:/ / www.ingenuity.com/ .
30. David.abcc.ncifcrf.gov [homepage on the Internet]. DAVID Bioinformatics Resources 6.7. National institute of allergy and infectious diseases, Frederick, MD [cited 2014 January 7]. Available from: http:/ / david.abcc.ncifcrf.gov/ home.jsp.
31. van Iersel MP, Kelder T, Pico AR et al. Presenting and exploring biological pathways with PathVisio. BMC Bioinformatics 2008; 9 : 399. doi: 10.1186/ 1471 - 2105 - 9-399.
Štítky
Detská onkológia Chirurgia všeobecná Onkológia
Článok vyšiel v časopiseKlinická onkologie
Najčítanejšie tento týždeň
2014 Číslo Supplementum- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Kombinace metamizol/paracetamol v léčbě pooperační bolesti u zákroků v rámci jednodenní chirurgie
- Antidepresivní efekt kombinovaného analgetika tramadolu s paracetamolem
- Srovnání analgetické účinnosti metamizolu s ibuprofenem po extrakci třetí stoličky
-
Všetky články tohto čísla
- Analýza proteinů pomocí hmotnostní spektrometrie
- p‑ SRM, SWATH a HRM – cílené proteomické přístupy na hmotnostním spektrometru TripleTOF 5600+ a jejich aplikace v onkologickém výzkumu
- Analýza fosfoproteínov a signálnych dráh kvantitatívno‑ proteomickými metódami
- Nové trendy ve studiu glykosylace proteinů u onkologických onemocnění
- Současné trendy ve využívání PET radiofarmak k onkologické diagnostice
- „Techneciová krize“ – příčiny, možná řešení a dopad na diagnostiku planární scintigrafií a SPECT
- Vitamin D jako významný steroidní hormon u karcinomu prsu
- Využití průtokové cytometrie pro analýzu mitochondriální buněčné smrti
- Soutěž o nejlepší práci
- Metody studia buněčné migrace a invazivity nádorových buněk
- Možnosti studia transendoteliální migrace in vitro
- Editorial
- Mechanizmy lékové rezistence a nádorové kmenové buňky
- Funkční testy pro detekci nádorových kmenových buněk
- Mikroprostředí nádoru – možnosti výzkumu v podmínkách in vitro
- Elektrochemická analýza nukleových kyselin, bílkovin a polysacharidů v biomedicíně
- Sekvenování nové generace a možnosti jeho využití v onkologické praxi
- Vývoj metod založených na PCR a jejich aplikace v onkologickém výzkumu a praxi
- Programovaná buněčná smrt v nádorových buňkách
- Metody pro studium protein‑proteinových a protein‑ligandových interakcí
- Detekce protein‑proteinových interakcí metodami FRET a BRET
- Detekce proteinů, proteinových interakcí a modifikací s využitím „proximity ligation assay“ in situ
- Exprese a purifikace proteinů
- Kvantitativní hmotnostní spektrometrie a její využití v onkologii
- Klinická onkologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Exprese a purifikace proteinů
- Metody studia buněčné migrace a invazivity nádorových buněk
- Sekvenování nové generace a možnosti jeho využití v onkologické praxi
- Analýza proteinů pomocí hmotnostní spektrometrie
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



