-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Vliv mikrobiomu na vznik a vývoj kolorektálního karcinomu
Role of the Microbiome in the Formation and Development of Colorectal Cancer
Background: The clinical, histopathological, and molecular characteristics of colorectal cancer vary considerably. Factors associated with the heterogeneity of this disease and with understanding the effects of heterogeneity on disease progression and response to therapy are critical for the better stratification of patients and the development of new therapeutic methods. Although studies have focused mainly on tumor molecular profiling, current molecular predictive and prognostic factors are relevant to specific groups of colorectal cancer patients and are mostly used to predict the applicability of targeted biological agents rather than to predict their benefits. Molecular profiling fails to capture aspects important for tumor growth and aggressiveness, including the tumor microenvironment. The gut microbiome, consisting of specific communities of all commensal, symbiotic, and pathogenic microorganisms, has been shown to have a significant impact on the development of many diseases, including Crohn’s disease, type II diabetes, and obesity. Recent studies have indicated that long-term dysbiosis of the intestinal microflora can influence the development and progression of colorectal cancer, as well as tumor aggressiveness and response to treatment.
Conclusion: This review article summarizes current knowledge of the gut microbiome in colorectal cancer, including the various mechanisms by which the gut microbiome affects the intestinal wall, thereby contributing to the development and progression of colorectal cancer.
This work was supported by Ministry of Health of the Czech Republic (project AZV 16-31966A), project of Ministry of Education, Youth and Sports of the Czech Republic – NPU I – LO1413 a Ministry of Health of the Czech Republic – RVO (MMCI, 00209805).
The authors declare they have no potential conflicts of interest concerning drugs, products, or services used in the study.
The Editorial Board declares that the manuscript met the ICMJE recommendation for biomedical papers.
Submitted: 15. 4. 2019
Accepted: 17. 6. 2019
Keywords:
dysbiosis
Autori: Barbora Zwinsová; Veronika Brychtová; Martina Hrivňáková; Lenka Zdražilová-Dubská; Beatrix Bencsiková; Roman Šefr; Rudolf Nenutil; Petra Vídeňská; Eva Budinská
Pôsobisko autorov: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
Vyšlo v časopise: Klin Onkol 2019; 32(4): 261-269
Kategória: Přehled
doi: https://doi.org/10.14735/amko2019261Súhrn
Východiska: Kolorektální karcinom je velice heterogenní onemocnění z klinického, histopatologického i molekulárního hlediska. Detailní charakterizace této heterogenity a její vliv na patologii tohoto onemocnění je nezbytným krokem k lepší stratifikaci pacientů a vývoji nových léčebných postupů. V posledních dvou desetiletích se pozornost vědců zaměřovala na studium molekulárních nádorových procesů pro prediktivní, diagnostické a prognostické účely. Avšak i přes veškeré úsilí jsou existující molekulární prediktivní a prognostické testy aplikovatelné pouze pro menší specifické skupiny pacientů a spíše pomáhají při rozhodování o nasazení specializované cílené biologické léčby, než by předpovídaly její úspěšnost. Samotné molekulární profilování není schopné zachytit řadu dalších faktorů významně ovlivňujících růst a agresivitu nádoru. Mezi tyto aspekty patří i mikroprostředí nádoru, jehož nejméně prostudovanou částí je střevní mikrobiom – specifické společenství všech komenzálních, symbiotických a patogenních mikroorganizmů. Střevní mikrobiom hraje klíčovou roli u řady onemocnění, jako je např. Crohnova choroba, diabetes II. typu a obezita a podle nejnovějších studií může dlouhodobá dysbióza střevní mikroflóry ovlivňovat i vznik a další vývoj kolorektálního karcinomu, jeho agresivitu nebo úspěšnost léčby.
Závěr: Tato přehledová studie sumarizuje dosavadní poznatky studia střevního mikrobiomu u kolorektálního karcinomu, vč. různých mechanizmů, jakými střevní mikrobiom ovlivňuje poškození stěny střeva a tím se může podílet na vzniku a progresi kolorektálního karcinomu.
Klíčová slova:
heterogenita – střevní mikrobiom – dysbióza
Úvod
Zhoubný novotvar tlustého střeva a konečníku (colorectal cancer – CRC) je vysoce heterogenní onemocnění, kterému v celosvětovém měřítku patří druhá (ženy) a třetí (muži) příčka v incidenci nádorových onemocnění [1]. V ČR se jedná o jednu z nejčastějších onkologických diagnóz a celosvětově má ČR šestou nejvyšší incidenci CRC [2]. V etiologii tohoto onemocnění hrají významnou roli environmentální vlivy, jako jsou stravovací návyky, konzumace alkoholu a kouření a také rizikové faktory, jako je věk, rodinná anamnéza, chronická zánětlivá onemocnění trávicího traktu a přítomnost polypů tlustého střeva. Přestože incidence CRC aktuálně zaznamenává ve vyspělých zemích mírný pokles především díky zavedení preventivních a screeningových programů [3], jedná se stále o onemocnění s komplikovanou léčbou a vysokou úmrtností. Důvodem je vysoká heterogenita tohoto onemocnění, a to jak interindividuální, tak heterogenita samotného nádoru jediného pacienta. Nádorová heterogenita CRC zahrnuje rozdíly na úrovni fenotypu nádorových buněk (jak morfologické, tak molekulární) a jejich interakci s nádorovým mikroprostředím projevující se přítomností buněk nenádorové tkáně (tzv. stromální reakce), variabilní infiltrací buňkami imunitního systému a – jak naznačují nejnovější výzkumy – také přítomností bakterií v samotném nádoru nebo na jeho povrchu z luminální strany tlustého střeva. Posledních 15 let se vědecký výzkum prognostických a prediktivních markerů CRC (a nádorového onemocnění obecně) zaměřoval zejména na molekulární profilování nádorových buněk a hodnocení faktorů nádorového mikroprostředí. Výsledky tohoto úsilí vedly k zavedení několika molekulárních biomarkerů, které v kombinaci se standardně používanými klinickými proměnnými slouží k individualizaci léčby. Jako příklad můžeme uvést stanovení mikrosatelitové nestability jako pozitivního prognostického markeru při indikaci adjuvantní chemoterapie na bázi 5-fluoruracilu u II. klinického stadia CRC nebo testování mutací genů KRAS, NRAS a BRAF při indikaci léčby metastatického CRC terapeutickými monoklonálními protilátkami cílenými proti receptoru pro epidermální růstový faktor [4]. Dále existuje několik vícegenových prognostických panelů určených pro predikci relapsu u časných stadií CRC jako Oncotype DX Colon Recurrence Score Test [5] a ColoPrint [6], které byly validovány retrospektivně v rámci řady klinických studií [7]. Jejich využití v klinické praxi však není rutinní, protože tyto testy nepredikují benefit adjuvantní chemoterapie. Podobně je na tom prognostický nástroj Immunoscore® [8] založený na kvantifikaci tumor-infiltrujících T buněk v parafinových řezech, který demonstruje význam složení nádorového mikroprostředí pro agresivitu CRC. Ve snaze blíže charakterizovat molekulární procesy CRC se pozornost zaměřila na definici molekulárních podtypů a v roce 2013 bylo publikováno několik klasifikačních systémů vycházejících z transkriptomických profilů CRC [9–12], které byly následně sjednoceny v koordinovaném úsilí za definice konsenzuálních molekulárních podtypů (consensual molecular subtypes – CMS) CRC s rozdílnou genovou expresí a silnou prognostickou hodnotou [13]. V roce 2017 pak byl navržen alternativní systém klasifikace CRC, Current Research Information System [14], založený na transkripčních profilech čisté populace nádorových buněk (bez nádorového stromatu). Oba systémy se vzájemně doplňují, ale neposkytují dostatečná kritéria, která by pomohla s nastavením specifické léčby na míru pacienta. Po 15 letech molekulárních výzkumů v oblasti CRC jsme tedy ještě stále vzdáleni svatému grálu personalizované medicíny. Jedním z pomyslných chybějících střípků mozaiky heterogenity CRC může být právě střevní mikrobiom, který, jak se ukazuje, má významný vliv na vznik a vývoj CRC.
Střevní mikrobiom
Lidské tělo je osídleno řádově triliony symbiotických mikroorganizmů vyskytujících se např. v trávicím traktu, na kůži, sliznici dutiny ústní a slinách, spojivce, dýchacích cestách, urogenitálním systému a dalších [15]. Pojem mikrobiom označuje specifické společenství všech komenzálních, symbiotických i patogenních mikroorganizmů v hostitelském organizmu a jejich genom. Patří sem bakterie, viry, archea a některé eukaryotní organizmy (prvoci, plísně a kvasinky). Každý člověk hostí více mikroorganizmů, než je počet jeho vlastních buněk, a také genetická informace lidského mikrobiomu je několikanásobně větší než genetická informace samotného člověka [16–18]. Z hlediska zdraví je nejvýznamnější mikrobiom tlustého střeva, zejména bakterie, které se podílejí na metabolizmu komplexních složek potravy, které by jinak nebylo možné využít v přeměně na energii, dále na syntéze esenciálních i neesenciálních aminokyselin, enzymů a vitaminů (vitamin K, B12, kyselina listová), můžou neutralizovat potenciálně karcinogenní sloučeniny [19], anebo naopak škodlivé sloučeniny produkovat [20]. Významnými metabolity jsou např. mastné kyseliny s krátkým řetězcem (short chain fatty acids – SCFA), které slouží jako zdroj energie nejen pro kolonocyty, ale i pro ostatní buňky a orgány [21]. Střevní mikrobiom tak ovlivňuje i morfologii střeva – architekturu sliznice, množství a složení hlenu, prokrvenost a proliferaci epiteliálních buněk [22]. V posledních letech se studium střevního mikrobiomu orientuje zejména na jeho interakci s imunitním systémem, která je důležitá obzvláště v období po narození, kdy se významně podílí na vývoji imunity. Pokud nedojde ke správnému vývoji imunitní reakce, dochází k vývoji alergií či autoimunitních chorob. Interakce mezi mikrobiomem a imunitním systémem později napomáhá k rovnováze mezi eliminací patogenů napadajících trávicí trakt a udržením tolerance ke zdravé tkáni střeva [23,24]. Složení střevního mikrobiomu se nejvíce mění od narození přibližně do 3 let věku [25]. V pozdějším věku je složení mikrobiomu stabilní, pokud není narušena jeho rovnováha a nedojde k prudkému snížení diverzity mikrobiomu nebo dominanci bakterií, které nepatří mezi přínosné komenzální zástupce. Tento nerovnovážný stav mikrobiomu nazýváme dysbióza a může být vyvolán např. léčbou antibiotiky, nevhodnou stravou nebo onemocněním. Dysbióza je spojována s vývojem různých chronických a autoimunitních chorob, jako je např. Crohnova choroba, diabetes II. typu, obezita apod. [26].
Střevní mikrobiom je tvořen zejména striktně anaerobními kmeny bakterií, které převažují nad bakteriemi fakultativně anaerobními a aerobními. Protože je velice obtížné tyto bakterie charakterizovat klasickými kultivačními metodami, jejich podrobnější studium umožnil až vývoj molekulárních metod. Vzhledem k velkému množství genetických informací se ve studiu mikrobiomu nejvíce uplatňují sekvenační techniky nové generace, které umožňují číst velké množství genetických informací najednou, a tak identifikovat složení a funkci mikrobiomu. Nejrozšířenější je stále analýza bakteriálního složení pomocí genu kódujícího 16S ribozomální ribonukleové kyseliny (rRNA) se specifickými oblastmi pro jednotlivé bakterie a analýza funkce mikrobiomu pomocí celometagenomového sekvenování [27].
Složení mikrobiomu a abundance jednotlivých bakteriálních taxonů odrážejí fyziologické rozdíly jednotlivých částí gastrointestinálního traktu, a není tedy ve střevním lumen rovnoměrné. Nejvíce bakterií se nachází v tlustém střevě, kde dochází k významnému rozkladu zbytků tráveniny bakteriemi. U zdravého člověka jsou z více než 90 % dominujícími bakteriálními kmeny gram-pozitivní Firmicutes a gram-negativní Bacteroidetes [28,29], jejichž poměr se významně interindividuálně liší [30]. Mezi další významně zastoupené kmeny patří Actinobacteria, Verrucomicrobia a Proteobacteria [31,32]. Na úrovni řádů jsou dominujícími sacharolytické Bacteriodales (kmen Bacteroidetes) a Clostridiales (kmen Firmicutes).
Dopad složení střevního mikrobiomu na zdraví člověka je studován zejména porovnáním složení mikrobiomu u nemocných a zdravých jedinců. Rozsáhlé populační studie jako US National Institute of Health Human Microbiome Project [18] nebo European Metagenomics of the Human Intestinal Tract project [33] ukazují, že ve složení střevního mikrobiomu existuje velká variabilita jak mezi etniky, tak i jedinci, a není proto jednoduché definovat složení „zdravého“ mikrobiomu. Snaha o stratifikaci mikrobiomu vyústila v roce 2011 v definování tří základních enterotypů lišících se dominancí jednoho z bakteriálních druhů – Bacteroides, Prevotella a Ruminococcus [34]. Navazující práce ukázaly, že řazení jedinců do enterotypů je velice závislé na použité metodice a není dostatečně ostře vymezené. Nicméně budoucí studie založené na velkých datových souborech s normovanou metodikou mohou tuto problematiku posunout k využití v léčbě a diagnostice nejenom střevních onemocnění [35].
Mikrobiom a kolorektální karcinom
Střevní mikrobiom může, ať už v podobě individuálních bakteriálních zástupců, nebo spolupůsobením mikrobiální komunity, potencovat nebo zmírňovat riziko vzniku CRC. Řada publikací ukazuje, že dysbióza střevní mikroflóry anebo poškození stěny střeva v důsledku interakce mikrobiomu s buňkami imunitního systému hostitele může ovlivňovat rozvoj zánětlivých a nádorových onemocnění střeva, progresi nádorů [36,37] a jejich odpověď na léčbu [38,39]. Bakterie a jejich produkty se mohou podílet na vzniku nebo progresi sporadického CRC řadou různých mechanizmů, jako je indukce prozánětlivých a prokarcinogenních drah v epiteliálních buňkách, produkce genotoxinů a reaktivních forem kyslíku [40,41] nebo metabolická přeměna prokarcinogenních výživových faktorů na karcinogeny [42]. Za významný rizikový faktor vzniku nádorových onemocnění střeva je považována zánětlivá reakce ve střevním epitelu, která může být způsobena dlouhodobou dysbiózou a působením patogenních nebo oportunně (podmíněně) patogenních bakterií [43–45].
Zdravý střevní epitel má řadu obranných mechanizmů pro boj s mikrobiálními narušiteli. Patří sem hlenová vrstva a udržování epiteliální integrity zamezující vniku mikroorganizmů, rychlá výměna epiteliálních buněk, a tedy odstraňování buněk infikovaných, autofagie a vrozená imunitní odpověď. Neméně důležitá je také ochranná funkce komenzálních bakterií, které svojí přítomností na sliznici za normálních okolností zabraňují průniku patogenů nebo jejich produktů. Nicméně mnoho bakteriálních patogenů vyvinulo téměř dokonalé infekční strategie, kterými tyto obranné mechanizmy obcházejí. Během svého působení vylučují různé toxiny a efektory, kterými mohou ovlivňovat hostitelské buněčné funkce a využívat je pro svoje přežití. Zároveň udržují rovnováhu v narušené epiteliální bariéře tak, aby ji mohli kolonizovat dlouhodobě [46]. Vedle patogenních bakterií mohou svými metabolity přispívat ke vzniku CRC také bakterie primárně nepatogenní, jejichž prokarcinogenní potenciál může být umocněn poškozením střevní sliznice následkem zranění nebo infekce [46]. Všechny mechanizmy, kterými střevní mikrobiom jako celek přispívá k udržení zdravého prostředí, nebo naopak rozvoji patologických stavů vč. tumorigeneze v mikrobiálně bohatém a imunologicky komplexním prostředí střeva, ovšem stále nejsou zcela objasněny.
Látky, kterými bakterie negativně působí na buňky epitelu nebo imunitní buňky hostitele, mají cyklomodulační (buněčný cyklus ovlivňující) nebo genotoxický účinek. V důsledku toho může u epiteliálních buněk docházet k poškození deoxyribonukleové kyseliny (DNA), akumulaci mutací a nekontrolované proliferaci. Produkty bakterií se můžou podílet na zastavení dělení imunitních buněk, čímž prakticky umožní sobě i nádorovým buňkám uniknout imunitní odpovědi. Z tohoto pohledu se jeví pro vznik CRC jako nejvýznamnější enterotoxické kmeny Escherichia coli, které produkují hned čtyři skupiny cyklomodulinů – cytotoxický nekrotizující faktor (CNF), faktor inhibující buněčný cyklus (Cif), cytoletální distendující toxin (CDT) a kolibaktin, který má také genotoxické účinky. CDT a Cif a kolibaktin inhibují proliferaci, CNF zase proliferaci spouští. CDT kromě enteropatogenních kmenů E. coli produkují také Shigella dysenteriae, Campylobacter spp., Salmonella typhi, Haemophilus ducreyi nebo Actinobacillus actinomycetemcomitans [47]. Enterotoxické kmeny Bacteroides fragilis produkují toxin ze skupiny metaloproteáz, který narušuje spojení střevních epiteliálních buněk štěpením transmembránového proteinu E-cadherinu [48–50]. Degradace E-cadherinu má za následek aktivaci -kateninové signální dráhy v buňkách epitelu, což způsobuje jejich zvýšenou proliferaci. Je zajímavé, že enteropatogenní kmeny E. coli jsou také schopny dramaticky snížit expresi klíčových DNA mismatch-repair proteinů MSH2 a MLH1 [51], a mohou dokonce aktivovat senescentní buňky k produkci růstových faktorů, které ovlivňují nádorový růst [52]. Ke komenzálním bakteriím s prokarcinogenním účinkem patří např. Enterococcus faecalis, která může poškozovat DNA epiteliálních buněk svou produkcí extracelulárních superoxidů [53,54]. Sulfidogenní bakterie jako Fusobacterium, Desulfovibrio a Bilophila wadsworthia se mohou účastnit procesu nádorového bujení produkcí sirovodíku, jenž je známý svým genotoxickým účinkem [55].
Dalším faktorem, jak se mohou bakterie úspěšněji podílet na progresi procesu tumorigeneze, je tvorba biofilmu – homogenního nebo heterogenního společenstva mikroorganizmů tvořícího vyšší strukturu obklopenou extracelulárními polymerními látkami, které mikroorganizmy v biofilmu vylučují a chrání je tak před nepříznivými podmínkami a imunitou [56,57]. Bakterie schopné adherence na různé povrchy jsou tzv. primární kolonizátoři, ke kterým se později mohou připojit bakterie, které by jinak nebyly samotné adherence schopné – sekundární kolonizátoři [58,59]. Přítomnost biofilmu pozměňuje metabolizmus v nádorové tkáni, produkci regulátorů buněčné proliferace a potenciálně ovlivňuje vývoj a progresi nádoru [60].
Bakteriální model „driver-passenger“
Známý model „adenom–karcinom“ vývoje CRC, který byl navržen Fearonem & Vogelsteinem [61], má svou paralelu i v bakteriálním působení na proces tumorigeneze. V modelu adenom–karcinom je pojem „driver–passenger“ běžně používán ve spojení s genovými mutacemi, přičemž „driver“ jsou mutace řídící, tedy zodpovědné za vznik a vývoj nádoru, a „passenger“ jsou mutace vzniklé sekundárně v procesu nádorového vývoje [62]. Tjalsma et al přišli s myšlenkou aplikace tohoto modelu na bakterie a vznik CRC. Hlavní myšlenka tohoto modelu je taková, že vznik a vývoj CRC je iniciován „driver“ bakteriemi, které svou aktivitou přímo nebo nepřímo způsobují poškození DNA a které v důsledku mohou přispívat k hromadění mutací charakteristických pro adenomy a karcinomy a k procesu tumorigeneze [63]. Proces nádorového bujení pak mění lokální mikroprostředí, zvyšuje se propustnost střevní stěny a mění se buněčný metabolizmus, což umožní uchycení a pomnožení oportunních „passenger“ bakterií. Změna mikroprostředí tedy způsobí, že patogenní bakteriální „drivery“ mohou být postupně nahrazeny „passenger“ bakteriemi, které buď dále podněcují, nebo naopak pozastaví proces tumorigeneze [63] (schéma 1).
Schéma 1. Bakteriální „driver-passenger“ model. 
Za bakteriální „drivery“ jsou tedy považovány střevní bakterie s prokarcinogenními vlastnostmi. Do této kategorie patří výše uvedené střevní patogenní bakterie. Jako bakteriální „passengers“ jsou definované ty střevní bakterie, které relativně málo kolonizují střevo zdravých lidí, ale dokážou se úspěšně množit v mikroprostředí nádoru. Mohou to být oportunní patogeny, komenzály nebo probiotické bakterie a podle toho může dojít buď k progresi, nebo potlačení tumorigeneze [63].
Tento model vznikl na základě řady studií, které porovnávají bakteriální složení ze stolice nebo stěrů střevní tkáně u pacientů s CRC, adenomem a zdravých kontrol. V tab. 1 je uveden přehled aktuálních studií a jejich výsledků porovnávajících změny ve stolici a nádorové tkáni. Omezením těchto studií je, že neberou v úvahu možnou rozdílnost v mikrobiálním složení danou podtypy nádorů.
Tab. 1. Aktuální přehled studií porovnávajících změny ve střevním mikrobiomu ve vzorcích stolice a stěrech nádrové tkáně. 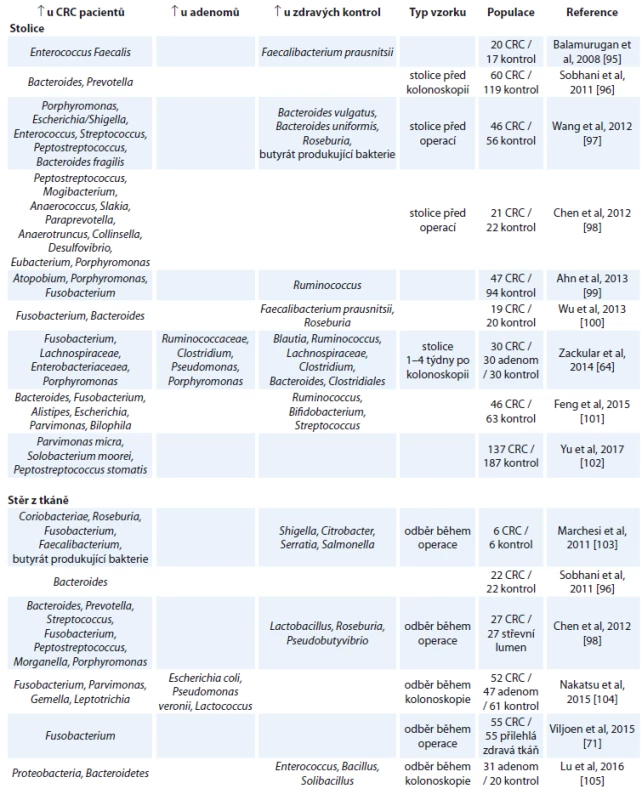
CRC – kolorektální karcinom Studie porovnávající bakteriální složení stolice u pacientů s CRC, adenomem a zdravými kontrolami obvykle cílily na využití mikrobiomu stolice jakožto přídatného neinvazivního testu na CRC. V roce 2014 dva vědecké týmy nezávisle na sobě publikovaly studie pojednávající o možnosti využití mikrobiomu jako screeningového nástroje pro diagnostiku začínajícího CRC [64,65]. Oba týmy dospěly k závěru, že kombinací základních rizikových faktorů pro vznik CRC (body mass index, věk a rasa) a mikrobiálního složení stolice lze lépe odlišit skupiny pacientů s CRC, pacientů s adenomem a zdravých lidí.
Mikrobiom, klinické a molekulární proměnné
Další typ studií se zaměřuje na korelaci bakteriálního složení na povrchu nádorové tkáně nebo ve stolici pacientů s CRC s klinickými proměnnými ve snaze pochopit odlišnosti mezi různými podtypy nádorů a podchytit možné biologické pozadí jejich heterogenity.
Lokalizace primárního CRC se ukazuje jako významný klinický faktor, který ovlivňuje charakteristiku nádorů tlustého střeva, a to nejen histopatologickou, ale i molekulární [66]. Je známo, že proximální část střeva je odlišného embryonálního vývoje než jeho distální část [67] a s tím se pojí i rozdíl v cévním zásobování, v expresi antigenů a metabolizmu glukózy, což má za následek odlišné osídlení bakteriemi i u zdravých jedinců [68]. Tento efekt se projevuje také ve významném rozdílu ve složení mikrobiomu mezi pravostrannými a levostrannými nádory tlustého střeva [68]. Proximální část zdravého střeva např. obsahuje větší počet bakterií než část distální [67], ale relativní prevalence dominantních rodů (Lactococcus, Fusobacterium, Pseudomonas a Flavobacterium) je v pravé a levé části zdravého střeva podobná. U nádorů je ovšem rod Fusobacterium více zastoupen v levé, distální části tlustého střeva [68]. Zde je také více zastoupena bakterie Escherichia-Shigella (tyto dva druhy bakterií jsou natolik příbuzné, že je nelze od sebe odlišit), jejíž přítomnost může napomáhat rozvoji karcinogeneze, ale u zdravých jedinců její výskyt převažuje v proximální části. Bakterie rodu Prevotella, které jsou spojované se zvýšenou produkcí IL-17 ve střevní sliznici pacientů, dále pak Selenomonas, Peptostreptoccus a kmen Firmicutes jsou více zastoupeny v nádorech s proximální lokalizací CRC [68]. Rozdíl mezi pravostrannými a levostrannými nádory je i ve výskytu bakteriálního biofilmu. Ve studii Johnsona et al se biofilm vyskytoval v 89 % nádorových tkání a ve všech vzorcích polypů vyskytujících se pravostranně, zatímco u levostranných nádorů se vyskytoval pouze ve 12 % nádorů a v žádném polypu [60].
Další asociace byla nalezena mezi bakteriálním složením střevní mukózy a sporadickými CRC asociovanými s kolitidou, u nichž byl zaznamenán vyšší výskyt bakterií z čeledi Enterobacteriaceae a rodu Sphingomonas a nižší výskyt Fusobacterium a Ruminococcus v porovnání se sporadickými CRC neasociovanými s kolitidou [69].
Studií asociujících molekulární proměnné s mikrobiomem zatím není mnoho. Existují publikace, které porovnávají složení mikrobiomu mezi mikrosatelitně stabilními (MSS) CRC a nádory s mikrosatelitní nestabilitou (MSI). MSS nádory vykazují zvýšený výskyt E. coli produkující kolibaktin [70], zatímco MSI nádory zvýšený výskyt Fusobacterium nucleatum a E. coli neprodukující kolibaktin [70–72].
V roce 2017 první a zatím jediná výzkumná skupina publikovala výsledky korelace mikrobiomu s konsenzuálními molekulárními podtypy CRC [73]. Analýza bakteriálního složení proběhla cílenou sekvenací genu pro 16S rRNA z nádoru a zároveň zkoumáním RNA sekvencí samotného nádoru, které nebyly přiřazené k referenčnímu lidskému genomu. Validace pak proběhla pomocí kvantitativní polymerázové řetězové reakce. Tímto způsobem byly identifikovány bakterie, které byly asociovány s konsenzuálními molekulárními podtypy CRC [13]. Zvýšený výskyt bakterií Fusobacterium hwasooki a Porphyromonas gingivalis je spojován s molekulárním podtypem CMS1 [73]. U tohoto podtypu se také ve větším množství vyskytovaly bakterie Treponema denticola a Tannerrella forsythia, které se společně s Porphyromonas gingivalis podílejí na tvorbě již výše zmíněného biofilmu [74,75]. Druhý molekulární podtyp (CMS2 Canonical) obsahoval zvýšený výskyt bakterií Selenomonas a Prevotella. Bacillus coagulants je asociován s třetím molekulárním podtypem (CMS3 Metabolic). Tato studie je ovšem limitovaná malým množstvím vzorků (n = 34), na kterých byla analýza provedena, a faktem, že ani jeden vzorek nebyl zařazen do čtvrtého konsenzuálního podtypu (CMS4 Mesenchymal), který je navíc prognosticky nejvýznamnější [73].
Funkční studie bakterie Fusobacterium nucleatum
Často pozorované asociace zvýšeného výskytu bakterií Fusobacterium nucleatum ve stolici i nádorové tkáni u pacientů s CRC vedl k řadě následných detailnějších studií, a tak se z bakterie F. nucleatum stal zřejmě nejlépe popsaný druh tohoto bakteriálního rodu z pohledu CRC. Jedná se o bakterie podporující zánět, které se abundantně vyskytují ve střevním mikrobiomu pacientů se zánětlivým střevním onemocněním [76]. Přítomnost F. nucleatum podporuje expresi celé řady zánětlivých cytokinů, např. IL-6, IL-8, IL-10, IL-18, TNF- [72–74]. F. nucleatum usnadňuje díky ojedinělým dvěma typům adherence spojení primárních a sekundárních kolonizátorů a podílí se tak výrazným způsobem na vzniku biofilmu [58]. Schopnost F. nucleatum zachytit se na střevní sliznici nebo do ní invadovat je závislá na formě adhezivního proteinu FadA, který umožňuje jejich přilnutí k epitelovým buňkám střevní sliznice. Současně dochází k sérii událostí, které vyústí ve stimulaci proliferace CRC buněk [77]. Kromě uvedeného dochází k snížení sekrece mucinů pohárkovými buňkami střeva jakožto hlavní složky slizniční ochrany střeva [78–81].
Vyšší výskyt F. nucleatum v nádorové tkáni koreluje s nižší infiltrací nádorových lézí T lymfocyty, vyšším stadiem onemocnění a horší prognózou z důvodu vyšší agresivity těchto nádorů [72,82]. Zdá se, že jedním z důvodů, proč dochází k potlačování imunitní odpovědi organizmu v místě nádorové léze napadené F. nucleatum, je skutečnost, že metabolity této bakterie jsou krátké peptidy a SCFA, které slouží jako chemoatraktant pro myeloidní supresorové buňky [83]. Je zajímavé, že výskyt F. nucleatum je spojován s molekulárním podtypem CRC, který je charakteristický vysokou MSI, mutací BRAF a s pravostrannou anatomickou lokalizací [72,84], dále pak s hypermetylací MLH1 a vysokou metylací CpG (CIMP-H) [72].
V roce 2017 provedli Bullman et al [85] pokus na myších, jimž byl zaveden štěp pocházející z primárního CRC získaného od pacientů. Tyto myši byly následně léčeny antibiotiky s cílem snížit množství F. nucleatum. Následně bylo pozorováno zpomalení proliferace nádorových buněk a růstu nádoru [85].
Nedávno byl identifikován nový druh bakterie Fusobacterium, Fusobacterium hwasookii, která byla ve studiích založených na cílené sekvenaci 16S rRNA genu, která má menší přesnost určení do druhů, vždy určována jako F. nucleatum [86]. Na základě podobnosti v sekvencích F. hwasookii a F. nucleatum a výskytu vysoce konzervovaného genu FadA u F. hwasookii se zdá, že F. hwasookii má podobný význam v karcinogenezi jako F. nucleatum.
Mikrobiom a léčba kolorektálního karcinomu
Rovnováha mezi bakteriemi je důležitá pro ochranu sliznice střeva a změny vyvolané chemoterapií mohou zvyšovat riziko výskytu infekce. Chemoterapie i radiační léčba ovlivňuje složení střevního mikrobiomu a působí toxicky na střevní sliznici, čímž umožňuje prostup bakterií do vrstvy epitelu. Zwielehner et al [87] ukázali, že množství bakterií klesá u pacientů ihned po aplikování chemoterapie a k obnovení dochází po 5–9 dnech. Chemoterapie významně ovlivnila výskyt komenzálních bakterií z rodu Bacteroides a Bifidobacteria a potenciálních patogenů z rodu Clostridium skupina IV [87]. Přemnožení potenciálních patogenů, jako je např. Clostridium difficile, může vést u pacientů s nádorovým onemocněním k závažným komplikacím [88,89].
Protinádorová léčba má vliv na složení mikrobiomu a i složení mikrobiomu může mít vliv na účinnost léčby. Studie na myších modelech odhalují, že střevní mikrobiom má vliv na úspěšnost chemoterapie tím, že ovlivňuje diferenciaci a funkci myeloidních buněk v mikroprostředí nádoru [38]. V této studii Goldszmid et al zkoumali vliv mikrobiomu na léčbu platinovými deriváty, jako je např. oxaliplatina nebo cisplatina. Tyto sloučeniny se vážou na DNA buněk, narušují její strukturu a tím inhibují syntézu a proliferaci proteinů a indukují apoptózu [90]. Oxaliplatina navíc indukuje imunogenní buněčnou smrt, která pak aktivuje protinádorovou imunitu zprostředkovanou T lymfocyty [91]. Léčebný efekt cisplatiny a oxaliplatiny byl mnohem nižší u myší léčených antibiotiky a bezmikrobních myší. U myší léčených antibiotiky se vytvořily sloučeniny platiny a DNA nádorových buněk na stejné úrovni jako u kontrolních myší, avšak již 48 hod po léčbě došlo k významnému poklesu cytotoxicity. Antibiotická léčba myší potlačila veškerou modifikaci genové exprese indukovanou oxaliplatinou [38].
Závěr
Interakce hostitel-mikrobiom se zvláštním důrazem na roli střevního mikrobiomu nejen v imunitní homeostáze a autoimunitních onemocněních, ale i ve spojení se vznikem nádorů bude velmi diskutovaným tématem následujících let. Poslední desetiletí ve výzkumu CRC bylo věnováno především studiu prognostických a prediktivních markerů na základě genové exprese. Nicméně tyto poznatky oproti standardním klinickým faktorům v porovnání s vynaloženým úsilím na jejich získání jen nepatrně zasáhly do klinické praxe. Vezmeme-li v potaz nejnovější znalosti o vlivu mikrobiomu na vznik a vývoj CRC, dá se předpokládat, že studium mikrobiomu může vést k lepšímu porozumění heterogenity CRC a přiblíží nás více k efektivní diagnostice a léčbě. Navíc se v posledních pár letech ukazuje, že nejen bakterie v nádoru, ale i bakterie na jeho povrchu tvořící bakteriální biofilm ovlivňují vývoj CRC. Výsledky studií je však třeba interpretovat velmi opatrně, neboť výsledky jsou závislé na typu odběru, uchování vzorků a izolace bakteriální DNA ze vzorku [92–94].
Z klinického hlediska existuje několik rozdílných terapeutických přístupů, které by potenciálně mohly být použity jako prevence vzniku nádorových onemocnění, jako podpůrná terapie při léčbě nádorových onemocnění anebo ke zvýšení odpovědi na léčbu. Mezi tyto přístupy patří užívání probiotik a prebiotik, změna stravování nebo mikrobiální transplantace. Poslední z výše zmiňovaných ukazuje velmi slibné výsledky při léčbě infekce bakterií Clostridium difficile u lidí a je navrhovaná jako léčebný postup v případě idiopatických střevních zánětů a metabolických poruch [39].
Ačkoliv pozorujeme velký pokrok v oblasti výzkumu mikrobiomu, což dokazuje i rostoucí počet publikací na toto téma, stále existuje mnoho nezodpovězených otázek souvisejících s vlivem mikrobiomu na rozvoj CRC. V tomto směru zatím chybějí studie, které se na mikrobiom dívají ze širší perspektivy, vč. nádorového mikroprostředí a jeho klinické i molekulární heterogenity, a poskytly by tak komplexní náhled na vlivy vedoucí ke vzniku a vývoji CRC.
Tato práce byla podpořena Ministerstvem zdravotnictví České republiky (projekt AZV 16-31966A), projektem Ministerstva školství, mládeže a tělovýchovy České republiky – NPU I – LO1413 a Ministerstva zdravotnictví České republiky – RVO (MOÚ, 00209805).
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
Mgr. Eva Budinská, Ph.D.
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: eva.budinska@mou.cz
Obdrženo: 15. 4. 2019
Přijato: 17. 6. 2019
Zdroje
1. Siegel RL, Miller KD, Jemal A. Cancer Statistics, 2018. CA Cancer J Clin 2018; 68 (1): 7–30. doi: 10.3322/caac.21442.
2. Novotvary 2015. Ústav zdravotnických informací a statistik ČR. [online]. Dostupné z: https: //www.uzis.cz/publikace/novotvary-2015.
3. Arnold M, Sierra MS, Laversanne M et al. Global patterns and trends in colorectal cancer incidence and mortality. Gut 2017; 66 (4): 683–691. doi: 10.1136/gutjnl-2015-310912.
4. Tan C, Du X. KRAS mutation testing in metastatic colorectal cancer. World J Gastroenterol 2012; 18 (37): 5171–5180. doi: 10.3748/wjg.v18.i37.5171.
5. Renfro LA, Zhang N, Lopatin M et al. Prospective evaluation of a 12-gene assay on patient treatment decisions and physician confidence in mismatch repair proficient stage IIA colon cancer. Clin Colorectal Cancer 2017; 16 (1): 23–30. doi: 10.1016/j.clcc.2016.07.016.
6. Kopetz S, Tabernero J, Rosenberg R et al. Genomic classifier ColoPrint predicts recurrence in stage II colorectal cancer patients more accurately than clinical factors. Oncologist 2015; 20 (2): 127–133. doi: 10.1634/theoncologist.2014-0325.
7. Sharif S, O’Connell MJ. Gene signatures in stage II colon cancer: a clinical review. Curr Colorectal Cancer Rep 2012; 8 (3): 225–231. doi: 10.1007/s11888-012-0132-7.
8. Galon J, Pagès F, Marincola FM et al. Cancer classification using the immunoscore: a worldwide task force. J Transl Med 2012; 10 : 205. doi: 10.1186/1479-5876-10-205.
9. Budinska E, Popovici V, Tejpar S et al. Gene expression patterns unveil a new level of molecular heterogeneity in colorectal cancer. J Pathol 2013; 231 (1): 63–76. doi: 10.1002/path.4212.
10. Dienstmann R, Guinney J, Delorenzi M et al. Colorectal cancer subtyping consortium (CRCSC) identification of a consensus of molecular subtypes. J Clin Oncol 2014; 32 (Suppl 15): 3511–3511.
11. Marisa L, Reyniès A de, Duval A et al. Gene expression classification of colon cancer into molecular subtypes: characterization, validation, and prognostic value. PLoS Med 2013; 10 (5): e1001453. doi: 10.1371/journal.pmed.1001453.
12. Sadanandam A, Lyssiotis CA, Homicsko K et al. A colorectal cancer classification system that associates cellular phenotype and responses to therapy. Nat Med 2013; 19 (5): 619–625. doi: 10.1038/nm.3175.
13. Guinney J, Dienstmann R, Wang X et al. The consensus molecular subtypes of colorectal cancer. Nat Med 2015; 21 (11): 1350–1356. doi: 10.1038/nm.3967.
14. Isella C, Brundu F, Bellomo SE et al. Selective analysis of cancer-cell intrinsic transcriptional traits defines novel clinically relevant subtypes of colorectal cancer. Nat Commun 2017; 8 : 15107. doi: 10.1038/ncomms15 107.
15. Li K, Bihan M, Yooseph S et al. Analyses of the microbial diversity across the human microbiome. PloS One 2012; 7 (6): e32118. doi: 10.1371/journal.pone.0032118.
16. Sender R, Fuchs S, Milo R. Revised estimates for the number of human and bacteria cells in the body. PLoS Biol 2016; 14 (8): e1002533. doi: 10.1371/journal.pbio. 1002533.
17. Sender R, Fuchs S, Milo R. Are we really vastly outnumbered? Revisiting the ratio of bacterial to host cells in humans. Cell 2016; 164 (3): 337–340. doi: 10.1016/j.cell.2016.01.013.
18. Turnbaugh PJ, Ley RE, Hamady M et al. The human microbiome project: exploring the microbial part of ourselves in a changing world. Nature 2007; 449 (7164): 804–810. doi: 10.1038/nature06244.
19. Jandhyala SM, Talukdar R, Subramanyam C et al. Role of the normal gut microbiota. World J Gastroenterol 2015; 21 (29): 8787–8803. doi: 10.3748/wjg.v21.i29.8787.
20. Koeth RA, Wang Z, Levison BS et al. Intestinal microbiota metabolism of L-carnitine, a nutrient in red meat, promotes atherosclerosis. Nat Med 2013; 19 (5): 576–585. doi: 10.1038/nm.3145.
21. LeBlanc JG, Chain F, Martín R et al. Beneficial effects on host energy metabolism of short-chain fatty acids and vitamins produced by commensal and probiotic bacteria. Microb Cell Factories 2017; 16 (1): 79. doi: 10.1186/s12934-017-0691-z.
22. Sommer F, Bäckhed F. The gut microbiota – masters of host development and physiology. Nat Rev Microbiol 2013; 11 (4): 227–238. doi: 10.1038/nrmicro2974.
23. Ho JT, Chan GC, Li JC. Systemic effects of gut microbiota and its relationship with disease and modulation. BMC Immunol 2015; 16 : 21. doi: 10.1186/s12865-015-0083-2.
24. Wu HJ, Wu E. The role of gut microbiota in immune homeostasis and autoimmunity. Gut Microbes 2012; 3 (1): 4–14. doi: 10.4161/gmic.19320.
25. Hill CJ, Lynch DB, Murphy K et al. Evolution of gut microbiota composition from birth to 24 weeks in the INFANTMET cohort. Microbiome 2017; 5 (1): 4. doi: 10.1186/s40168-016-0213-y.
26. Jurjus A, Eid A, Al Kattar S et al. Inflammatory bowel disease, colorectal cancer and type 2 diabetes mellitus: the links. BBA Clin 2015; 5 : 16–24. doi: 10.1016/j.bbacli.2015.11.002.
27. Di Bella JM, Bao Y, Gloor GB et al. High throughput sequencing methods and analysis for microbiome research. J Microbiol Methods 2013; 95 (3): 401–414. doi: 10.1016/j.mimet.2013.08.011.
28. Eckburg PB. Diversity of the human intestinal microbial flora. Science 2005; 308 (5728): 1635–1638. doi: 10.1126/science.1110591.
29. Galperin MY. Genome diversity of spore-forming firmicutes. Microbiol Spectr 2013; 1 (2). doi: 10.1128/microbiolspectrum.TBS-0015-2012.
30. Qin J, Li R, Raes J et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature 2010; 464 (7285): 59–65. doi: 10.1038/nature08821.
31. Kang M, Martin A. Microbiome and colorectal cancer: unraveling host-microbiota interactions in colitis-associated colorectal cancer development. Semin Immunol 2017; 32 : 3–13. doi: 10.1016/j.smim.2017.04.003.
32. Serban DE. Gastrointestinal cancers: influence of gut microbiota, probiotics and prebiotics. Cancer Lett 2014; 345 (2): 258–270. doi: 10.1016/j.canlet.2013.08.013.
33. Ehrlich SD. MetaHIT: The European Union project on metagenomics of the human intestinal tract. In: Nelson KE (ed). Metagenomics of the human body. New York: Springer New York 2011 : 307–316.
34. Arumugam M, Raes J, Pelletier E et al. Enterotypes of the human gut microbiome. Nature 2011; 473 (7346): 174–180. doi: 10.1038/nature09944.
35. Costea PI, Hildebrand F, Arumugam M et al. Enterotypes in the landscape of gut microbial community composition. Nat Microbiol 2018; 3 (1): 8–16. doi: 10.1038/s41564-017-0072-8.
36. Chen J, Pitmon E, Wang K. Microbiome, inflammation and colorectal cancer. Semin Immunol 2017; 32 : 43–53. doi: 10.1016/j.smim.2017.09.006.
37. Dulal S, Keku TO. Gut microbiome and colorectal adenomas. Cancer J 2014; 20 (3): 225–231. doi: 10.1097/PPO.0000000000000050.
38. Goldszmid RS, Dzutsev A, Viaud S et al. Microbiota modulation of myeloid cells in cancer therapy. Cancer Immunol Res 2015; 3 (2): 103–109. doi: 10.1158/2326-6066.CIR-14-0225.
39. Dzutsev A, Goldszmid RS, Viaud S et al. The role of the microbiota in inflammation, carcinogenesis, and cancer therapy. Eur J Immunol 2015; 45 (1): 17–3. doi: 10.1002/eji.201444972.
40. Lax AJ. Opinion: Bacterial toxins and cancer – a case to answer? Nat Rev Microbiol 2005; 3 (4): 343–349. doi: 10.1038/nrmicro1130.
41. Song M, Garrett WS, Chan AT. Nutrients, foods, and colorectal cancer prevention. Gastroenterology 2015; 148 (6): 1244–1260. doi: 10.1053/j.gastro.2014.12.035.
42. Bonnet M, Buc E, Sauvanet P et al. Colonization of the human gut by E. coli and colorectal cancer risk. Clin Cancer Res 2014; 20 (4): 859–867. doi: 10.1158/1078-0432.CCR-13-1343.
43. Arthur JC, Perez-Chanona E, Mühlbauer M et al. Intestinal inflammation targets cance – inducing activity of the microbiota. Science 2012; 338 (6103): 120–123. doi: 10.1126/science.1224820.
44. Grivennikov SI, Greten FR, Karin M. Immunity, inflammation, and cancer. Cell 2010; 140 (6): 883–899. doi: 10.1016/j.cell.2010.01.025.
45. Sun J, Kato I. Gut microbiota, inflammation and colorectal cancer. Genes Dis 2016; 3 (2): 130–143. doi: 10.1016/j.gendis.2016.03.004.
46. Kim M, Ashida H, Ogawa M et al. Bacterial interactions with the host epithelium. Cell Host Microbe 2010; 8 (1): 20–35. doi: 10.1016/j.chom.2010.06.006.
47. De Rycke J, Oswald E. Cytolethal distending toxin (CDT): a bacterial weapon to control host cell proliferation? FEMS Microbiol Lett 2001; 203 (2): 141–148. doi: 10.1111/j.1574-6968.2001.tb10832.x.
48. Wu S, Lim KC, Huang J et al. Bacteroides fragilis enterotoxin cleaves the zonula adherens protein, E-cadherin. Proc Natl Acad Sci USA 1998; 95 (25): 14979–14984. doi: 10.1073/pnas.95.25.14979.
49. Wu S, Morin PJ, Maouyo D et al. Bacteroides fragilis enterotoxin induces c-Myc expression and cellular proliferation. Gastroenterology 2003; 124 (2): 392–400. doi: 10.1053/gast.2003.50047.
50. Sears CL, Geis AL, Housseau F. Bacteroides fragilis subverts mucosal biology: from symbiont to colon carcinogenesis. J Clin Invest 2014; 124 (10): 4166–4172. doi: 10.1172/JCI72334.
51. Maddocks ODK, Short AJ, Donnenberg MS et al. Attaching and effacing Escherichia coli downregulate DNA mismatch repair protein in vitro and are associated with colorectal adenocarcinomas in humans. PLoS One 2009; 4 (5): e5517. doi: 10.1371/journal.pone.0005517.
52. Cougnoux A, Dalmasso G, Martinez R et al. Bacterial genotoxin colibactin promotes colon tumour growth by inducing a senescence-associated secretory phenotype. Gut 2014; 63 (12): 1932–1942. doi: 10.1136/gutjnl-2013-305257.
53. Wang X, Huycke MM. Extracellular superoxide production by Enterococcus faecalis promotes chromosomal instability in mammalian cells. Gastroenterology 2007; 132 (2): 551–561. doi: 10.1053/j.gastro.2006.11. 040.
54. Wang X, Allen TD, May RJ et al. Enterococcus faecalis induces aneuploidy and tetraploidy in colonic epithelial cells through a bystander effect. Cancer Res 2008; 68 (23): 9909–9917. doi: 10.1158/0008-5472.CAN-08-1551.
55. Attene-Ramos MS, Wagner ED, Plewa MJ et al. Evidence that hydrogen sulfide is a genotoxic agent. Mol Cancer Res 2006; 4 (1): 9–14. doi: 10.1158/1541-7786.MCR-05-0126.
56. Dejea CM, Fathi P, Craig JM et al. Patients with familial adenomatous polyposis harbor colonic biofilms containing tumorigenic bacteria. Science 2018; 359 (6375): 592–597. doi: 10.1126/science.aah3648.
57. Dickson I. Colorectal cancer: Bacterial biofilms and toxins prompt a perfect storm for colon cancer. Nat Rev Gastroenterol Hepatol 2018; 15 (3): 129. doi: 10.1038/nrgastro.2018.16.
58. Kaplan CW, Lux R, Haake SK et al. The Fusobacterium nucleatum outer membrane protein RadD is an arginine-inhibitable adhesin required for inter-species adherence and the structured architecture of multispecies biofilm. Mol Microbiol 2009; 71 (1): 35–47. doi: 10.1111/j.1365-2958.2008.06503.x.
59. Li S, Konstantinov SR, Smits R et al. Bacterial biofilms in colorectal cancer initiation and progression. Trends Mol Med 2017; 23 (1): 18–30. doi: 10.1016/j.molmed.2016.11.004.
60. Johnson CH, Dejea CM, Edler D et al. Metabolism links bacterial biofilms and colon carcinogenesis. Cell Metab 2015; 21 (6): 891–897. doi: 10.1016/j.cmet.2015.04.011.
61. Fearon ER, Vogelstein B. A genetic model for colorectal tumorigenesis. Cell 1990; 61 (5): 759–767. doi: 10.1016/0092-8674 (90) 90186-i.
62. Vogelstein B, Kinzler KW. The multistep nature of cancer. Trends Genet 1993; 9 (4): 138–141.
63. Tjalsma H, Boleij A, Marchesi JR et al. A bacterial driver-passenger model for colorectal cancer: beyond the usual suspects. Nat Rev Microbiol 2012; 10 (8): 575–582. doi: 10.1038/nrmicro2819.
64. Zackular JP, Rogers MA, Ruffin MT et al. The human gut microbiome as a screening tool for colorectal cancer. Cancer Prev Res 2014; 7 (11): 1112–1121. doi: 10.1158/1940-6207.CAPR-14-0129.
65. Zeller G, Tap J, Voigt AY et al. Potential of fecal microbiota for early-stage detection of colorectal cancer. Mol Syst Biol 2014; 10 : 766. doi: 10.15252/msb.20145645.
66. Richman S, Adlard J. Left and right sided large bowel cancer. BMJ 2002; 324 (7343): 931–932. doi: 10.1136/bmj. 324.7343.931.
67. Hagland HR, Søreide K. Cellular metabolism in colorectal carcinogenesis: influence of lifestyle, gut microbiome and metabolic pathways. Cancer Lett 2015; 356 (2PtA): 273–280. doi: 10.1016/j.canlet.2014.02.026.
68. Gao Z, Guo B, Gao R et al. Microbiota disbiosis is associated with colorectal cancer. Front Microbiol 2015; 6 : 20. doi: 10.3389/fmicb.2015.00020.
69. Richard ML, Liguori G, Lamas B et al. Mucosa-associated microbiota dysbiosis in colitis associated cancer. Gut Microbes 2018; 9 (2): 131–142. doi: 10.1080/19490976.2017.1379637.
70. Gagnière J, Bonnin V, Jarrousse AS et al. Interactions between microsatellite instability and human gut colonization by Escherichia coli in colorectal cancer. Clin Sci (Lond) 2017; 131 (6): 471–485. doi: 10.1042/CS20160876.
71. Viljoen KS, Dakshinamurthy A, Goldberg P et al. Quantitative profiling of colorectal cancer-associated bacteria reveals associations between fusobacterium spp., enterotoxigenic bacteroides fragilis (ETBF) and clinicopathological features of colorectal cancer. PLoS One 2015; 10 (3): e0119462. doi: 10.1371/journal.pone.0119462.
72. Mima K, Nishihara R, Qian ZR et al. Fusobacterium nucleatum in colorectal carcinoma tissue and patient prognosis. Gut 2016; 65 (12): 1973–1980. doi: 10.1136/gutjnl-2015-310101.
73. Purcell RV, Visnovska M, Biggs PJ et al. Distinct gut microbiome patterns associate with consensus molecular subtypes of colorectal cancer. Sci Rep 2017; 7 (1): 11590. doi: 10.1038/s41598-017-11237-6.
74. Grenier D. Demonstration of a bimodal coaggregation reaction between Porphyromonas gingivalis and Treponema denticola. Oral Microbiol Immunol 1992; 7 (5): 280–284.
75. Meuric V, Martin B, Guyodo H et al. Treponema denticola improves adhesive capacities of Porphyromonas gingivalis. Mol Oral Microbiol 2013; 28 (1): 40–53. doi: 10.1111/omi.12004.
76. Han YW. Fusobacterium nucleatum: a commensal-turned pathogen. Curr Opin Microbiol 2015; 0 : 141–147. doi: 10.1016/j.mib.2014.11.013.
77. Rubinstein MR, Wang X, Liu W et al. Fusobacterium nucleatum promotes colorectal carcinogenesis by modulating E-cadherin/b-catenin signaling via its FadA adhesin. Cell Host Microbe 2013; 14 (2): 195–206. doi: 10.1016/j.chom.2013.07.012.
78. Allen-Vercoe E, Strauss J, Chadee K. Fusobacterium nucleatum: an emerging gut pathogen? Gut Microbes 2011; 2 (5): 294–298. doi: 10.4161/gmic.2.5.18603.
79. Krisanaprakornkit S, Kimball JR, Weinberg A et al. Inducible expression of human beta-defensin 2 by Fusobacterium nucleatum in oral epithelial cells: multiple signaling pathways and role of commensal bacteria in innate immunity and the epithelial barrier. Infect Immun 2000; 68 (5): 2907–2915. doi: 10.1128/iai.68.5.2907-2915.2000.
80. Moore RA, Warren RL, Freeman JD et al. The sensitivity of massively parallel sequencing for detecting candidate infectious agents associated with human tissue. PLoS One 2011; 6 (5): e19838. doi: 10.1371/journal.pone.0019838.
81. Strauss J, Kaplan GG, Beck PL et al. Invasive potential of gut mucosa-derived Fusobacterium nucleatum positively correlates with IBD status of the host. Inflamm Bowel Dis 2011; 17 (9): 1971–1978. doi: 10.1002/ibd.21606.
82. Mima K, Sukawa Y, Nishihara R et al. Fusobacterium nucleatum and T cells in colorectal carcinoma. JAMA Oncol 2015; 1 (5): 653–661. doi: 10.1001/jamaoncol.2015. 1377.
83. Anand S, Kaur H, Mande SS. Comparative in silico analysis of butyrate production pathways in gut commensals and pathogens. Front Microbiol 2016; 7 : 1945. doi: 10.3389/fmicb.2016.01945.
84. Xiao Y, Freeman GJ. The microsatellite instable subset of colorectal cancer is a particularly good candidate for checkpoint blockade immunotherapy. Cancer Discov 2015; 5 (1): 16–18. doi: 10.1158/2159-8290.CD-14-1397.
85. Bullman S, Pedamallu CS, Sicinska E et al. Analysis of Fusobacterium persistence and antibiotic response in colorectal cancer. Science 2017; 358 (6369): 1443–1448. doi: 10.1126/science.aal5240.
86. Cho E, Park SN, Lim YK et al. Fusobacterium hwasookii sp. nov., isolated from a human periodontitis lesion. Curr Microbiol 2015; 70 (2): 169–175. doi: 10.1007/s00284-014-0692-7.
87. Zwielehner J, Lassl C, Hippe B et al. Changes in human fecal microbiota due to chemotherapy analyzed by TaqMan-PCR, 454 sequencing and PCR-DGGE fingerprinting. PloS One 2011; 6 (12): e28654. doi: 10.1371/journal.pone.0028654.
88. Guarner F, Malagelada JR. Gut flora in health and disease. The Lancet 2003; 361 (9356): 512–519. doi: 10.1016/S0140-6736 (03) 12489-0.
89. van Vliet MJ, Tissing WJ, Dun CA et al. Chemotherapy treatment in pediatric patients with acute myeloid leukemia receiving antimicrobial prophylaxis leads to a relative increase of colonization with potentially pathogenic bacteria in the gut. Clin Infect Dis 2009; 49 (2): 262–270. doi: 10.1086/599346.
90. Siddik ZH. Cisplatin: mode of cytotoxic action and molecular basis of resistance. Oncogene 2003; 22 (47): 7265–7279. doi: 10.1038/sj.onc.1206933.
91. Kroemer G, Galluzzi L, Kepp O et al. Immunogenic cell death in cancer therapy. Annu Rev Immunol 2013; 31 : 51–72. doi: 10.1146/annurev-immunol-032712-100008.
92. Tedjo DI, Jonkers DM, Savelkoul PH et al. The effect of sampling and storage on the fecal microbiota composition in healthy and diseased subjects. PloS One 2015; 10 (5): e0126685. doi: 10.1371/journal.pone.0126685.
93. Mathay C, Hamot G, Henry E et al. Method optimization for fecal sample collection and fecal DNA extraction. Biopreservation Biobanking 2015; 13 (2): 79–93. doi: 10.1089/bio.2014.0031.
94. Panek M, Čipčić Paljetak H, Barešić A et al. Methodology challenges in studying human gut microbiota – effects of collection, storage, DNA extraction and next generation sequencing technologies. Sci Rep 2018; 8 (1): 5143. doi: 10.1038/s41598-018-23296-4.
95. Balamurugan R, Rajendiran E, George S et al. Real-time polymerase chain reaction quantification of specific butyrate-producing bacteria, Desulfovibrio and Enterococcus faecalis in the feces of patients with colorectal cancer. J Gastroenterol Hepatol 2008; 23 (8 Pt 1): 1298–1303. doi: 10.1111/j.1440-1746.2008.05490.x.
96. Sobhani I, Tap J, Roudot-Thoraval F et al. Microbial dysbiosis in colorectal cancer (CRC) patients. PloS One 2011; 6 (1): e16393. doi: 10.1371/journal.pone.0016393.
97. Wang T, Cai G, Qiu Y et al. Structural segregation of gut microbiota between colorectal cancer patients and healthy volunteers. ISME J 2012; 6 (2): 320–329. doi: 10.1038/ismej.2011.109.
98. Chen W, Liu F, Ling Z et al. Human intestinal lumen and mucosa-associated microbiota in patients with colorectal cancer. PloS One 2012; 7 (6): e39743. doi: 10.1371/journal.pone.0039743.
99. Ahn J, Sinha R, Pei Z et al. Human gut microbiome and risk for colorectal cancer. J Natl Cancer Inst 2013; 105 (24): 1907–1911. doi: 10.1093/jnci/djt300.
100. Wu N, Yang X, Zhang R et al. Dysbiosis signature of fecal microbiota in colorectal cancer patients. Microb Ecol 2013; 66 (2): 462–470. doi: 10.1007/s00248-013-0245-9.
101. Feng Q, Liang S, Jia H et al. Gut microbiome development along the colorectal adenoma-carcinoma sequence. Nat Commun 2015; 6 : 6528. doi: 10.1038/ncomms7528.
102. Yu J, Feng Q, Wong SH et al. Metagenomic analysis of faecal microbiome as a tool towards targeted non-invasive biomarkers for colorectal cancer. Gut 2017; 66 (1): 70–78. doi: 10.1136/gutjnl-2015-309800.
103. Marchesi JR, Dutilh BE, Hall N et al. Towards the human colorectal cancer microbiome. PloS One 2011; 6 (5): e20447. doi: 10.1371/journal.pone.0020447.
104. Nakatsu G, Li X, Zhou H et al. Gut mucosal microbiome across stages of colorectal carcinogenesis. Nat Commun 2015; 6 : 8727. doi: 10.1038/ncomms9727.
105. Lu Y, Chen J, Zheng J et al. Mucosal adherent bacterial dysbiosis in patients with colorectal adenomas. Sci Rep 2016; 6 : 26337. doi: 10.1038/srep26337.
Štítky
Detská onkológia Chirurgia všeobecná Onkológia
Článok vyšiel v časopiseKlinická onkologie
Najčítanejšie tento týždeň
2019 Číslo 4- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Kombinace metamizol/paracetamol v léčbě pooperační bolesti u zákroků v rámci jednodenní chirurgie
- Antidepresivní efekt kombinovaného analgetika tramadolu s paracetamolem
- Fixní kombinace paracetamol/kodein nabízí synergické analgetické účinky
-
Všetky články tohto čísla
- Farmakoekonomické studie a procesy HTA při hodnocení nákladů a benefitů nákladné inovativní léčby u nás i ve světě
- Současný pohled na HPV asociované karcinomy orofaryngu a roli p16 jako surogátního markeru high-risk HPV
- Vliv mikrobiomu na vznik a vývoj kolorektálního karcinomu
- Metody klasické a molekulární cytogenetiky vhodné pro biodozimetrii osob s profesionální expozicí karcinogenům
- Exprese ncRNA spojené s P21 aktivovaná poškozením DNA u karcinomu močového měchýře
- BMI a pravděpodobnost vzniku endometriálního adenokarcinomu u českých žen – případová studie
- The Pharmacoeconomic Analysis of Cetuximab and Panitumumab in the 1st Line Treatment of mCRC in Real Clinical Practice in the Czech Republic
- Incidencia a rizikové faktory vzniku vzdialených metastáz u pacientov s karcinómom hlavy a krku
- Úloha radioterapie při metastázách lebky u folikulárního karcinomu štítné žlázy
- Onkologická kazuistika – kdy je vhodný čas na integraci paliativní péče?
- Aplikovat jeden cyklus nebo jednu sérii chemoterapie?
- Urgentní chirurgická léčba GIST ezofago-gastrické junkce u pacienta s obrovskou hiátovou kýlou
- Zemřel prof. MUDr. Josef Koutecký, DrSc., zakladatel dětské onkologie
- Proposed Strategies for Improving Adherence to Tyrosine Kinase Inhibitors in Patients with Chronic Myeloid Leukaemia
- Onkologie v obrazech Kožní toxicita při cílené léčbě generalizovaného melanomu
- Klinická onkologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Vliv mikrobiomu na vznik a vývoj kolorektálního karcinomu
- Současný pohled na HPV asociované karcinomy orofaryngu a roli p16 jako surogátního markeru high-risk HPV
- Onkologická kazuistika – kdy je vhodný čas na integraci paliativní péče?
- Incidencia a rizikové faktory vzniku vzdialených metastáz u pacientov s karcinómom hlavy a krku
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



