-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Dlouhé nekódující RNA – současné možnosti detekce a využití v klinické praxi
Long Non-Coding RNAs – Current Methods of Detection and Clinical Applications
Background: Long non-coding RNAs (lncRNA) are more than 200-nucleotide-long RNA molecules that affect multiple physiologic phenomena and have important regulatory functions in cells. Their levels are often altered in various malignancies, thus they represent a potential biomarker for the diagnostics, prognosis or recurrence of cancer. Their importance has recently led to an enormous increase in a number of publications on the subject. The most frequently studied lncRNAs are HOTAIR, MALAT1 and PCA3.
Aim: Numerous methods are currently being developed for the analysis or detection of lncRNA. They are mostly based on optical methods used for the detection of messenger RNAs, including polymerase chain reaction with reverse transcription, fluorescence in situ hybridisation or next-generation sequencing, but caution must be taken due to their structural differences. Here, we describe not only standard but also novel techniques for lncRNA detection, including chemiluminescent and electrochemical techniques.
Conclusion: Despite the great progress and plethora of papers on this topic, there is only one single approved lncRNA-based diagnostic test, a PCA3 test for the diagnosis of prostate cancer from the patient’s urine. All other tests are only in their research phase and need to be validated. Nevertheless, lncRNA diagnostics offer enormous potential and thus it is highly probable that other diagnostic tests on different lncRNA types will soon appear.
Keywords:
Long non-coding RNA – tumor biomarkers – carcinogenesis – biosensing techniques
Autori: L. Moráňová; M. Bartošík
Pôsobisko autorov: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
Vyšlo v časopise: Klin Onkol 2019; 32(Supplementum 3): 65-71
Kategória: Přehled
doi: https://doi.org/10.14735/amko20193S65Súhrn
Východiska: Dlouhé nekódující RNA (long non-coding RNA – lncRNA) jsou RNA molekuly o délce větší než 200 nukleotidů, které ovlivňují řadu fyziologických funkcí a mají významnou regulační roli v buňkách. Jejich hladiny jsou často změněny u různých malignit, a představují tak slibný biomarker pro diagnostiku, prognózu nebo rekurenci nádorových onemocnění. Díky důležitosti těchto molekul výrazně roste i počet publikací na toto téma. Mezi nejčastěji studované lncRNA patří např. HOTAIR, MALAT1 a PCA3.
Cíl: V současné době jsou vyvíjeny různé metody pro analýzu či detekci lncRNA, obvykle založené na optických metodách pro detekci mediátorové RNA (mRNA), např. polymerázová řetězová reakce s reverzní transkripcí, fluorescenční in situ hybridizace nebo sekvenování nové generace. Je však potřeba dbát na rozdíly ve struktuře mRNA a lncRNA. V této práci popisujeme nejenom standardní metody, ale i nové přístupy pro detekci lncRNA zahrnující např. chemiluminescenční a elektrochemické techniky.
Závěr: I navzdory pokrokům a velkému množství publikovaných prací existuje pouze jeden schválený diagnostický test založený na detekci lncRNA, a to PCA3 test pro diagnostiku karcinomu prostaty analýzou moči. Ostatní jsou v současnosti pouze ve fázi vývoje a bude potřeba je validovat. Diagnostika založená na lncRNA i tak skýtá obrovský potenciál, a je proto velmi pravděpodobné, že se v blízké době objeví další diagnostické testy cílící na jiné typy lncRNA.
Klíčová slova:
dlouhá nekódující RNA – nádorové biomarkery – karcinogeneze – biosenzory
Úvod
I když pouze 2 % lidského genomu tvoří geny kódující proteiny, velká většina genomu (> 70 %) je transkripčně aktivní, dávající vznik různým formám tzv. nekódujících RNA s významnými regulačními funkcemi [1,2]. Mezi ně patří krátké nekódující RNA o délce menší než 200 nukleotidů (nt, obvykle kolem 20–30 nt), zejména mikroRNA, malé interferující RNA nebo Piwi-interagující RNA a též rozsáhlá skupina tzv. dlouhých nekódujících RNA (long non-coding RNA – lncRNA) o délce větší než 200 nt (často až 100 000 nt). Dle NONCODE databáze [3] se v lidském genomu nachází více než 96 000 genů pro lncRNA a až 172 000 transkriptů těchto genů; naopak LNCipedia [4] udává střízlivější odhad, zaokrouhlený na cca 55 000 genů [5]. Tyto geny se nacházejí v různých oblastech genomu, a to jak v nekódujících regionech, tak i v intronech genů kódujících proteiny. Navíc mohou být přepisovány nejenom ze stejného řetězce jako protein-kódující geny (tzv. „sense“ transkripty), ale i z řetězce opačného („antisense“ transkripty), či dokonce mohou sdílet promotor s protein-kódujícím genem v opačném řetězci (divergentní lncRNA). Transkripty lncRNA jsou obvykle generovány RNA polymerázou II, obdobně jako mRNA mají většinou na 5’-konci metylguanozinovou čepičku, na 3’-konci poly (A) konec a podstupují sestřih. Na rozdíl od mRNA ovšem postrádají otevřený čtecí rámec, takže nemohou být překládány do proteinů.
Geny kódující lncRNA jsou často pojmenovány dle sousedících protein-kódujících genů [6]. Jejich klasifikace je relativně složitá, dají se dělit např. dle umístění v genomu, způsobu aktivace nebo dle mechanizmu účinku. Hlavní třídy lncRNA, detailněji popsané v přehledném článku Šány et al [7], jsou mj. mezigenové lncRNA (umístěné mezi dvěma geny), intronové lncRNA (umístěné uvnitř intronů genů), lncRNA asociované s telomerami nebo pseudogeny (kopie genů neschopné kódovat proteiny). I když funkce většiny lncRNA transkriptů nejsou známy, lze obecně říci, že jejich úloha spočívá v regulaci genové exprese, modulaci proteinové aktivity či remodelaci chromatinu, příp. slouží jako prekurzory krátkých nekódujících RNA nebo jsou součástí systému jejich umlčení [8]. Obr. 1 naznačuje, že mechanizmus účinku lncRNA je závislý na vazbě na DNA, RNA nebo protein. Vzhledem k jejich roli při regulaci genové exprese není překvapivé, že při patogenních stavech jsou pozorovány změny jejich hladin, které mohou být detekovatelné dříve než viditelné morfologické změny. Tato skutečnost dělá z lncRNA ideální kandidáty nejen na biomarkery nádorových onemocnění [1,7,9–11], ale např. i kardiovaskulárních chorob [12] nebo Alzheimerovy choroby [13]. Díky důležitosti těchto molekul tak roste i počet publikací na toto téma; jenom v roce 2018 jich bylo přes 4 000 (graf 1). Bližší informace o funkci, klasifikaci nebo struktuře několika hlavních skupin lncRNA je možné najít v článcích publikovaných v dřívějších ročnících Klinické onkologie [7,9]; v tomto článku se chceme zaměřit především na nejnovější poznatky v oblasti metod detekce a jejich aplikace do klinické praxe.
Obr. 1. Tři různé mechanizmy působení lncRNA. Jedná se o interakce s: A) chromatinem (DNA), kdy dochází k remodelaci chromatinu po vazbě lncRNA; B) různými proteiny, kde lncRNA může modulovat protein-proteinové interakce; nebo C) mRNA, kde dochází k narušení translace. 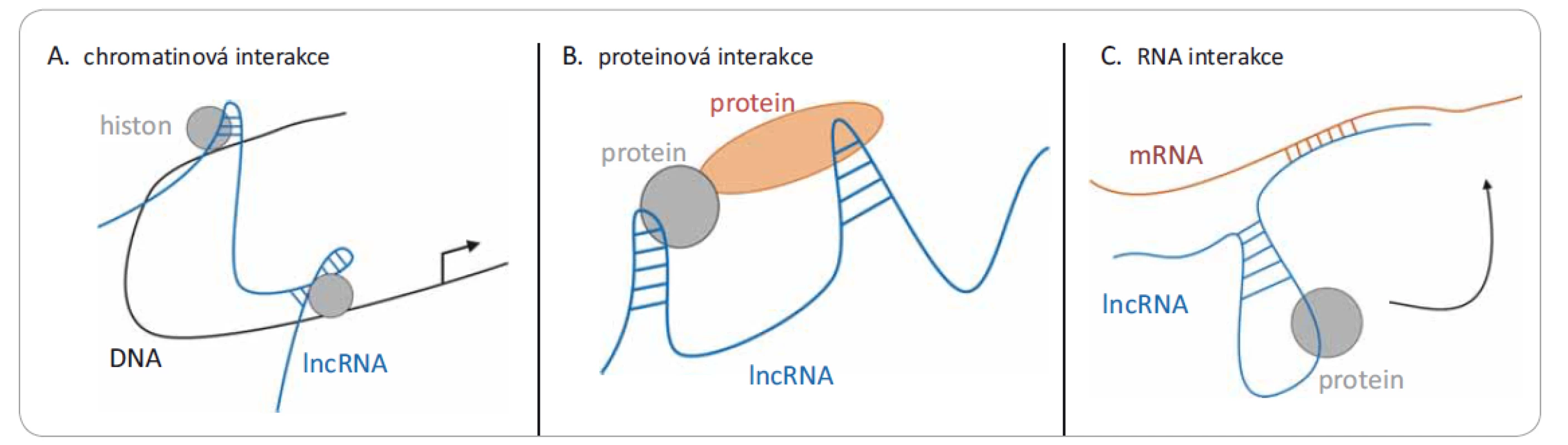
lncRNA – dlouhé nekódující RNA, mRNA – mediátorová RNA Graf 1. Nárůst publikační aktivity zabývající se dlouhými nekódujícími RNA od roku 2000 do roku 2018, dle Web of Science (hledaný výraz: lncRNA OR long noncoding RNA). 
Role lncRNA v karcinogenezi a její aplikace do klinické diagnostiky
Dnes už není pochyb o tom, že deregulace lncRNA je implikována v různých patologických stavech a může vést až k maligní transformaci buněk. Role lncRNA přitom byla prokázána v podstatě u všech charakteristických znaků rakoviny (hallmarks of cancer) definovaných ve dvou dnes již legendárních článcích Hanahana et al [14,15]. Některé konkrétní lncRNA a jejich asociace s těmito znaky jsou blíže popsány v tab. 1. Jde např. o aktivaci proliferačních signálních drah, deregulaci nádorových supresorových drah nebo o účast při metastazování [11,16].
Tab. 1. Spojitost vybraných lncRNA a charakteristických znaků rakoviny (hallmarks of cancer). 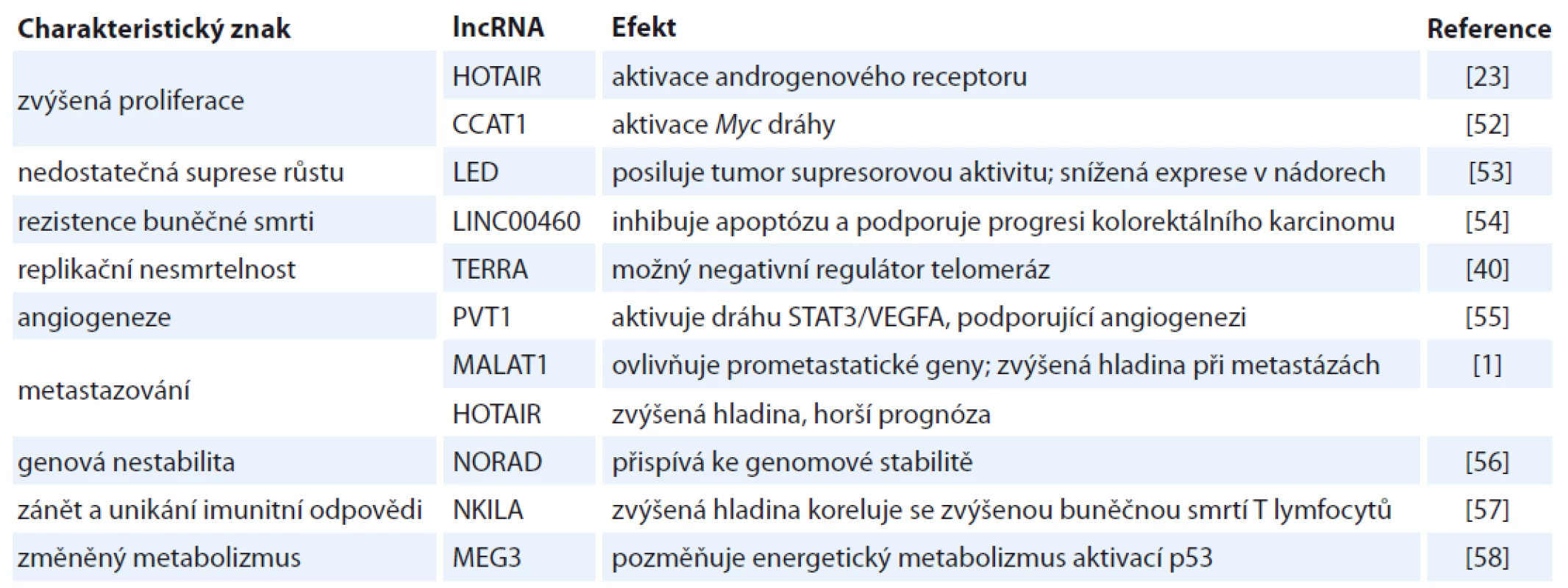
lncRNA – dlouhé nekódující RNA Jednou z nejvíce studovaných lncRNA je HOTAIR (HOX antisense intergenic RNA). Tato lncRNA s délkou cca 2,2 kb reguluje dynamiku chromatinu a ovlivňuje transkripci jiných genů vazbou na histon metylázu PRC2 a histon demetylázu LSD1 [17,18]. Množství studií prokázalo deregulaci HOTAIR v desítkách nádorů, vč. nádorů prsu [19], tlustého střeva [20], slinivky břišní [21], vaječníků [22] nebo prostaty [23]. U nádorů tlustého střeva korelovala zvýšená hladina HOTAIR nejenom s progresí onemocnění, ale zvýšila se i chemorezistence k cytostatikům [24].
Další diagnosticky zajímavou lncRNA je PCA3 (prostate cancer antigen 3), jejíž hladina je zvýšená u více než 90 % mužů s karcinomem prostaty [25]. V roce 2012 americký Úřad pro kontrolu potravin a léčiv (FDA) schválil použití PCA3 diagnostického testu v moči mužů nad 50 let s předchozí negativní biopsií prostaty, který by tak mohl snížit počet opakovaných biopsií a zároveň poskytnout doplňkové informace ke standardnímu testu prostatického specifického antigenu (prostate specific antigen – PSA). Test na PCA3 má sice nižší citlivost než PSA test, na druhé straně vykazuje vyšší specificitu, a může tak predikovat karcinom prostaty u případů, kdy PSA test selhává [26]. Hladina PCA3 byla ve více než 90 % studovaných případů karcinomu prostaty asi 60–100× vyšší než ve zdravé prostatické tkáni [27] a zároveň nedetekovatelná v jiných typech nádorů [28]. PCA3 také může sloužit jako indikátor agresivity rozvíjejícího se nádoru prostaty [29]. Přes značné výhody tohoto biomarkeru se v ČR toto vyšetření zatím takřka neprovádí.
Některé lncRNA mohou dokonce indikovat metastatický potenciál nádoru. Mezi ně patří např. MALAT1 (metastasis-associated lung adenocarcinoma transcript 1), obvykle s vysokou koncentrací v jádře, kde reguluje prometastatické geny [30]. MALAT1 je vysoce konzervovaná lncRNA, jejíž umlčení vede ke snížení proliferace a migrace nádorových buněk [31] a k aktivaci tumor-supresorového genu TP53 [32]. U některých typů nádorů byla pozorována zvýšená hladina MALAT1 korelující s progresí metastáz a celkově horší prognózou, např. u plicního adenokarcinomu, kolorektálního karcinomu, karcinomu slinivky břišní, karcinomu močového měchýře, karcinomu jícnu nebo gliomu [1,33–36]. Souhrnná analýza několika nezávislých studií prokázala, že tato lncRNA je spojená s horší prognózou, kratším přežíváním a rekurencí onemocnění [37]. Nicméně data z nedávné studie provedené na myších modelech naznačila, že v prsních nádorových buňkách má MALAT1 opačný, tj. potlačující účinek na vznik metastáz, a autoři tak navrhují zpřesnění zažitého modelu definující MALAT1 jako prometastatické lncRNA [38].
Další efekt lncRNA je možné pozorovat na úrovni telomer, a to prostřednictvím tzv. TERRA (telomere repeat-containing RNA) molekul, které obsahují telomerové repetice UUAGGG na svém 3’-konci a podílejí se na regulaci telomerové homeostázy [39]. Vzhledem k tomu, že až 85 % lidských nádorů překoná replikační limit daný délkou telomer (tím, že reaktivují expresi enzymu telomeráza), jeví se regulační role TERRA lncRNA jako zásadní [7]. Sampl et al zjistili, že expresní hladina TERRA negativně koreluje s malignitou gliomů [40]. Přesná role TERRA jakožto negativního regulátoru telomeráz in vivo však stále zůstává nejasná [41]. V tab. 2 jsou uvedeny příklady studií využívajících početnější soubory klinických vzorků pro validaci různých druhů lncRNA.
Tab. 2. Vybrané studie pro validaci lncRNA jako biomarkeru. 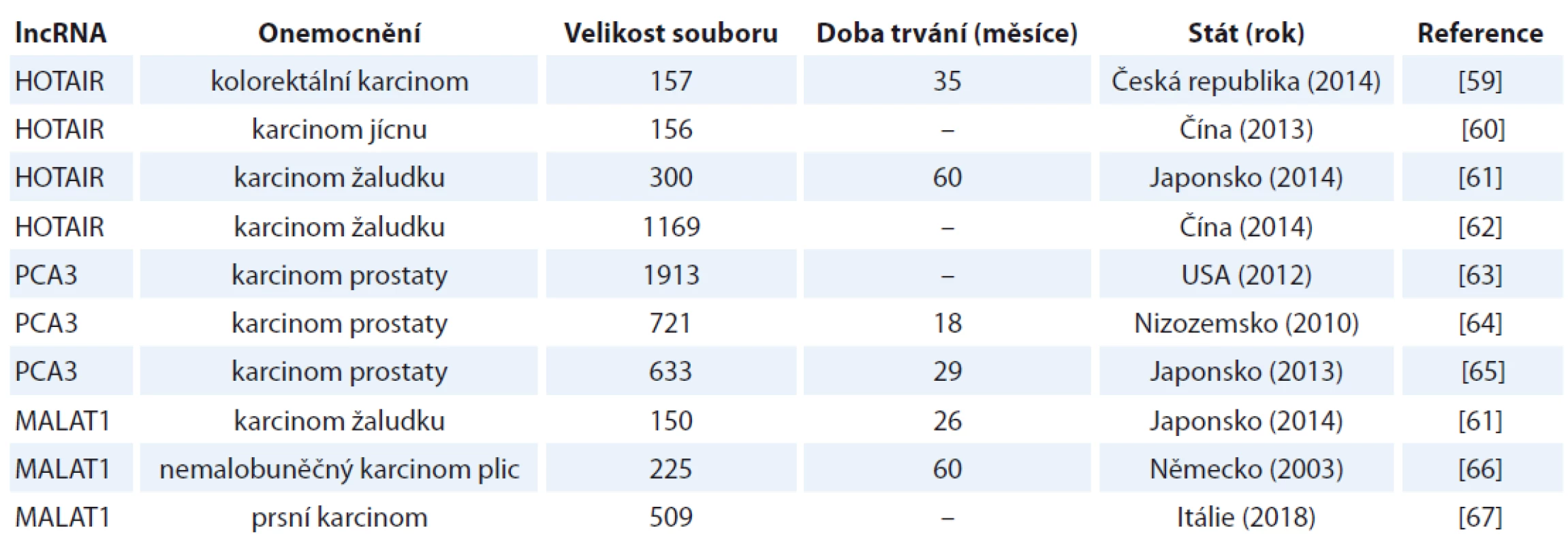
lncRNA – dlouhé nekódující RNA Metody detekce
V současné době existuje pouze jeden lncRNA test schválený pro klinickou diagnostiku, a to výše zmíněný PCA3 test (s názvem Progensa PCA3 Assay) od firmy Hologic, využívaný pro diagnostiku karcinomu prostaty. Detekce probíhá v moči po masáži per rectum, aby došlo k uvolnění buněk prostaty. Moč je následně modifikována přidáním média, které lyzuje uvolněné buňky a stabilizuje RNA. Princip samotného testu spočívá v hybridizaci cílové RNA na povrch magnetických kuliček, které slouží pro separaci cílové RNA. Dále proběhne reverzní transkripce pomocí první sady primerů a RNA amplifikace pomocí další sady primerů. Dochází tedy nejprve k přepisu lncRNA do komplementární DNA (complementary DNA – cDNA) a dále k její amplifikaci pomocí DNA dependentní RNA polymerázy T7. V závěru jsou RNA amplikony detekovány pomocí chemiluminiscenčně značené DNA sondy komplementární k RNA. Kvantifikace se provádí měřením intenzity produkovaného záření a jejím porovnáním s referenčními vzorky, které jsou součástí diagnostické sady. Daný produkt je nabízen v lehce obměněné verzi specificky i pro PSA. Obě laboratorní stanovení se tedy dají provádět současně, a je tak možné PCA3 hodnoty vztáhnout k hladině PSA [42].
PCA3 je možné detekovat také pomocí klasické imunoenzymatické metody (enzyme-linked immuno sorbent assay – ELISA). Tuto možnost komerčně nabízí společnost MyBioSource pod názvem PCA3 ELISA Kit. Ten je ovšem určený pouze pro výzkumné účely a nemá oprávnění pro použití v klinické diagnostice. Princip metody vychází z klasické sendvičové ELISA. PCA3 je zachycena pomocí PCA3-specifické protilátky a poté označena další specifickou protilátkou konjugovanou s křenovou peroxidázou. Na základě enzymatické reakce peroxidázy se substrátem vzniká barevný produkt, který je kvantifikován měřením absorbance při 450 nm [43].
I ostatní vyvíjené metody jsou určené pouze pro výzkumné účely, obvykle se jedná o modifikaci metod pro detekci dlouhých RNA obecně, i když ne všechny jsou použitelné. Některé z nich určené pro izolaci, identifikaci nebo nespecifickou amplifikaci mRNA např. využívají přítomnosti poly (A) konce na 3’-konci mRNA. Vzhledem k tomu, že ne všechny lncRNA nesou poly (A) konec [44], může tak při vysokokapacitních analýzách docházet k zatížení velkými chybami. Technologie sekvenování nové generace přitom často využívá záchyt mRNA pomocí poly (T) oligonukleotidů. Společnost Novogene [45] tak nabízí protokol a reagencie k vytváření knihoven a sekvenací profilů lncRNA a firma Roche pod označením SeqCap lncRNA probes [46] nabízí doplňkové sondy lncRNA pro kompletní profilaci celého transkriptomu. Oproti tomu společnost Illumina v rámci standardního protokolu využívá metodu přípravy sekvenční knihovny z depletované RNA (celkové RNA zbavené ribozomální RNA), a tudíž nedochází ke ztrátě informace o lncRNA bez poly (A) konců. Tento postup tedy umožňuje celkovou analýzu lncRNA profilu bez nutnosti pořízení dodatečných reagencí nebo obměny protokolu [47].
Další metodou z klasického portfolia pro studium mRNA je polymerázová řetězová reakce s reverzní transkripcí. Touto metodou nejprve dochází k přepisu RNA do cDNA a ta je následně amplifikována. V reálném čase je zaznamenáván nárůst fluorescence, který koresponduje s množstvím DNA ve vzorku. Některé společnosti vyvinuly reagencie pro přepis do cDNA pomocí univerzálních poly (T) primerů, které se vážou na poly (A) konce. Tento přístup je pro komplexní analýzu lncRNA nevhodný, dá se ovšem modifikovat jednoduchou kombinací poly (T) primerů a tzv. random hexamers (využívaných v nespecifické reverzní transkripci). Tuto vylepšenou a zjednodušenou variantu pro studium lncRNA nabízí např. společnost BioRad [48].
Klasickou metodou pro kvantifikaci lncRNA in situ je i fluorescenční in situ hybridizace (FISH), která využívá fluorescenční hybridizační sondu komplementární ke studované lncRNA a umožňuje kvantifikaci a lokalizaci pomocí fluorescenčního mikroskopu. Firma LGC Biosearch Technologies nabízí sadu reagencií pod názvem Stelaris® RNA FISH a fluorescenčně značené sondy pro nejčastěji studované lncRNA vč. možnosti značení různými fluorofory pro simultánní detekci více biomolekul současně [49].
Další možnost kvantifikace lncRNA představuje elektrochemie, která nabízí relativně levné a nenáročné přístrojové vybavení, možnost miniaturizace a paralelního měření. Zajímavým příkladem je elektrochemický biosenzor založený na izotermální amplifikaci s dUTP značeným biotinem [50]. Amplikony byly přes vazbu streptavidin-biotin navázány na streptavidinové magnetické kuličky a zároveň označeny peroxidázou konjugovanou s biotinem (obr. 2). Výsledný komplex byl inkubován s tetrametylbenzidinem, jehož oxidovaný produkt vykazuje modré zbarvení pozorovatelné pouhým okem. Autoři kvantifikovali HOTAIR lncRNA spektrofotometricky při 652 nm a paralelně i elektrochemicky pomocí chronoamperometrie. Metoda byla vyvinuta na ovariálních nádorových liniích a validována na vzorcích plazmy od tří pacientek s epiteliálním ovariálním karcinomem a dvou benigních kontrol. Ještě novější je biosenzor pro detekci lncRNA MALAT1 pomocí zlatých nanočástic, polyamid-aminu, detekční sondy a peroxidázy [51]. Autoři zde demonstrovali použití biosenzoru na jaterních nádorových liniích, avšak použili vzorky krevní plazmy pouze pro hledání případných interferencí s detekční metodou přídavkem syntetické molekuly MALAT1 do vzorků plazmy.
Obr. 2. Schéma kolorimetrické a elektrochemické detekce HOTAIR kombinující izotermální amplifi kace lncRNA, vazbu amplikonů na magnetické kuličky a enzymatickou reakci zprostředkovanou HRP. TMB jakožto substrát pro HRP je detekován volným okem nebo citlivěji elektrochemicky. Upraveno z [58], se svolením Royal Society of Chemistry. ![Schéma kolorimetrické a elektrochemické detekce HOTAIR kombinující izotermální amplifi kace lncRNA, vazbu amplikonů na
magnetické kuličky a enzymatickou reakci zprostředkovanou HRP. TMB jakožto substrát pro HRP je detekován volným okem nebo
citlivěji elektrochemicky. Upraveno z [58], se svolením Royal Society of Chemistry.](https://www.prelekara.sk/media/cache/resolve/media_object_image_small/media/image_pdf/9b6a0b86a152e2d0d0f4d21d09e3eae2.png)
HRP – křenová peroxidáza, TMB – tetrametylbenzidin Závěr
Přítomnost, a tudíž snadná detekovatelnost lncRNA nejen v tkáních, ale především v tělních tekutinách nabízí možnost širokého využití lncRNA jako prognostického a prediktivního biomarkeru. Velké množství lncRNA skýtá obrovský potenciál k diagnostickému využití, avšak dobře prostudovaných mechanizmů působení a funkcí lncRNA je doposud objasněno jen několik set. Díky tkáňové specificitě hladin lncRNA lze vybrat vhodné kandidáty pro včasný záchyt onkologických onemocnění i pro doplnění panelu již existujících biomarkerů za účelem zpřesnění diagnostiky. V posledních letech se provádějí zejména meta-analýzy kombinující výsledky jednotlivých studií, hledající na základě chování nádoru vhodný biomarker predikující jeho vlastnosti. I přes důležitost lncRNA bude pro jejich zařazení mezi klinicky relevantní biomarkery důležité vyvinout spolehlivé metody jejich detekce a prokázat, že hladiny studovaných lncRNA skutečně korelují s karcinogenezí, prognózou nebo rekurencí nádorového onemocnění.
Práce byla podpořena projekty MŠMT – NPU I – LO1413 a GAČR 17-08971S.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
Mgr. Martin Bartošík, Ph.D.
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: martin.bartosik@mou.cz
Obdrženo/Submitted: 28. 5. 2019
Přijato/Accepted: 30. 7. 2019
Zdroje
1. Weidle UH, Birzele F, Kollmorgen G et al. Long non-coding RNAs and their role in metastasis. Cancer Genom Proteom 2017; 14 (3): 143–160. doi: 10.21873/cgp.20027.
2. Mattick JS, Rinn JL. Discovery and annotation of long noncoding RNAs. Nat Struct Mol Biol 2015; 22 (1): 5–7. doi: 10.1038/nsmb.2942.
3. noncode.org. An integrated knowledge database dedicated to ncRNAs, especially lncRNAs. [online]. Available from: http: //www.noncode.org/.
4. LNCipedia. A comprehensive compendium of human long non-coding RNAs. [online]. Available from: https: //lncipedia.org/.
5. Volders PJ, Anckaert J, Verheggen K et al. LNCipedia 5: towards a reference set of human long non-coding RNAs. Nucleic Acid Res 2018; 47 (D1): D135–D139. doi: 10.1093/nar/gky1031.
6. Gibbons HR, Shaginurova G, Kim LC et al. Divergent lncRNA GATA3-AS1 regulates GATA3 transcription in T-helper 2 cells. Front Immunol 2018; 9 : 2512. doi: 10.3389/fimmu.2018.02512.
7. Šána J, Faltejsková P, Svoboda M et al. Dlouhé nekódující RNA a jejich význam u nádorových onemocnění. Klin Onkol 2012; 25 (4): 246–254. doi: 10.14735/amko2012246.
8. Dykes IM, Emanueli C. Transcriptional and post-transcriptional gene regulation by long non-coding RNA. Genom Proteom Bioinform 2017; 15 (3): 177–186. doi: 10.1016/j.gpb.2016.12.005.
9. Gromesová B, Kubaczková V, Bollová B et al. Potenciál dlouhých nekódujících molekul RNA v diagnostice nádorových onemocnění. Klin Onkol 2016; 29 (1): 20–28. doi: 10.14735/amko201620.
10. Qi P, Zhou XY, Du X. Circulating long non-coding RNAs in cancer: current status and future perspectives. Mol Cancer 2016; 15 (1): 39. doi: 10.1186/s12943-016-0524-4.
11. Schmitt AM, Chang HY. Long noncoding RNAs in cancer pathways. Cancer Cell 2016; 29 (4): 452–463. doi: 10.1016/j.ccell.2016.03.010.
12. Sallam T, Sandhu J, Tontonoz P. Long noncoding RNA discovery in cardiovascular disease. Circul Res 2018; 122 (1): 155–166. doi: 10.1161/CIRCRESAHA.117.311802.
13. Luo Q, Chen Y. Long noncoding RNAs and Alzheimer’s disease. Clin Interv Aging 2016; 11 : 867–872. doi: 10.2147/CIA.S107037.
14. Hanahan D, Weinberg RA. The hallmarks of cancer. Cell 2000; 100 (1): 57–70. doi: 10.1016/s0092-8674 (00) 81683-9.
15. Hanahan D, Weinberg Robert A. Hallmarks of cancer: the next generation. Cell 2011; 144 (5): 646–674. doi: 10.1016/j.cell.2011.02.013.
16. de Oliveira JC, Oliveira LC, Mathias C et al. Long non-coding RNAs in cancer: Another layer of complexity. J Gene Med 2019; 21 (1): e3065. doi: 10.1002/jgm.3065.
17. Bhan A, Mandal SS. LncRNA HOTAIR: a master regulator of chromatin dynamics and cancer. Biochim Biophys Acta 2015; 1856 (1): 151–164. doi: 10.1016/j.bbcan.2015.07.001.
18. Gupta RA, Shah N, Wang KC et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature 2010; 464 (7291): 1071–1076. doi: 10.1038/nature08975.
19. Avazpour N, Hajjari M, Tahmasebi Birgani M. HOTAIR: a promising long non-coding RNA with potential role in breast invasive carcinoma. Front Genet 2017; 8 : 170. doi: 10.3389/fgene.2017.00170.
20. Luo ZF, Zhao D, Li XQ et al. Clinical significance of HOTAIR expression in colon cancer. World J Gastroenterol 2016; 22 (22): 5254–5259. doi: 10.3748/wjg.v22.i22.5254.
21. Yang SZ, Xu F, Zhou T et al. The long non-coding RNA HOTAIR enhances pancreatic cancer resistance to TNF-related apoptosis-inducing ligand. J Biol Chem 2017; 292 (25): 10390–10397. doi: 10.1074/jbc.M117.786830.
22. Liu S, Lei H, Luo F et al. The effect of lncRNA HOTAIR on chemoresistance of ovarian cancer through regulation of HOXA7. Biol Chem 2018; 399 (5): 485–497. doi: 10.1515/hsz-2017-0274.
23. Zhang A, Zhao JC, Kim J et al. LncRNA HOTAIR enhances the androgen-receptor-mediated transcriptional program and drives castration-resistant prostate cancer. Cell Rep 2015; 13 (1): 209–221. doi: 10.1016/j.celrep.2015.08.069.
24. Xiao Z, Qu Z, Chen Z et al. LncRNA HOTAIR is a prognostic biomarker for the proliferation and chemoresistance of colorectal cancer via miR-203a-3p-mediated wnt/ß-catenin signaling pathway. Cell Physiol Biochem 2018; 46 (3): 1275–1285. doi: 10.1159/000489110.
25. Wright CM. Long noncoding RNAs and cancer. In: Gray SG (ed). Epigenetic Cancer Therapy. Boston: Academic Press 2015 : 91–114.
26. Lu Y, Madu C. Prostate cancer biomarkers. In: Gupta RC (ed). Biomarkers in Toxicology. Boston: Academic Press 2014 : 771–783.
27. de Kok JB, Verhaegh GW, Roelofs RW et al. DD3 (PCA3), a very sensitive and specific marker to detect prostate tumors. Cancer Res 2002; 62 (9): 2695–2698.
28. Bussemakers MJG, van Bokhoven A, Verhaegh GW et al. DD3: a new prostate-specific gene, highly overexpressed in prostate cancer. Cancer Res 1999; 59 (23): 5975–5979.
29. Taille Adl, Irani J, Graefen M et al. Clinical evaluation of the PCA3 assay in guiding initial biopsy decisions. J Urol 2011; 185 (6): 2119–2125. doi: 10.1016/j.juro.2011.01.075.
30. Gutschner T, Hämmerle M, Eissmann M et al. The noncoding RNA MALAT1 is a critical regulator of the metastasis phenotype of lung cancer cells. Cancer Res 2013; 73 (3): 1180–1189. doi: 10.1158/0008-5472.CAN-12-2850.
31. Wang X, Li M, Wang Z et al. Silencing of long noncoding RNA MALAT1 by miR-101 and miR-217 inhibits proliferation, migration, and invasion of esophageal squamous cell carcinoma cells. J Biol Chem 2015; 290 (7): 3925–3935. doi: 10.1074/jbc.M114.596866.
32. Tripathi V, Shen Z, Chakraborty A et al. Long noncoding RNA MALAT1 controls cell cycle progression by regulating the expression of oncogenic transcription factor B-MYB. PLoS Genet 2013; 9 (3): e1003368. doi: 10.1371/journal.pgen.1003368.
33. Ying L, Chen Q, Wang Y et al. Upregulated MALAT-1 contributes to bladder cancer cell migration by inducing epithelial-to-mesenchymal transition. Mol Biosyst 2012; 8 (9): 2289–2294. doi: 10.1039/c2mb25070e.
34. Wu XS, Wang XA, Wu WG et al. MALAT1 promotes the proliferation and metastasis of gallbladder cancer cells by activating the ERK/MAPK pathway. Cancer Biol Ther 2014; 15 (6): 806–814. doi: 10.4161/cbt.28584.
35. Hu L, Wu Y, Tan D et al. Up-regulation of long noncoding RNA MALAT1 contributes to proliferation and metastasis in esophageal squamous cell carcinoma. J Exp Clin Canc Res 2015; 34 (1): 7. doi: 10.1186/s13046-015-0123-z.
36. Ma KX, Wang HJ, Li XR et al. Long noncoding RNA MALAT1 associates with the malignant status and poor prognosis in glioma. Tumour Biol 2015; 36 (5): 3355–3359. doi: 10.1007/s13277-014-2969-7.
37. Tian X, Xu G. Clinical value of lncRNA MALAT1 as a prognostic marker in human cancer: systematic review and meta-analysis. BMJ Open 2015; 5 (9): e008653. doi: 10.1136/bmjopen-2015-008653.
38. Kim J, Piao HL, Kim BJ et al. Long noncoding RNA MALAT1 suppresses breast cancer metastasis. Nat Genet 2018; 50 (12): 1705–1715. doi: 10.1038/s41588-018-0252-3.
39. Oliva-Rico D, Herrera LA. Regulated expression of the lncRNA TERRA and its impact on telomere biology. Mech Age Develop 2017; 167 : 16–23. doi: 10.1016/j.mad.2017.09.001.
40. Sampl S, Pramhas S, Stern C et al. Expression of telomeres in astrocytoma WHO grade 2 to 4: TERRA level correlates with telomere length, telomerase activity, and advanced clinical grade. Transl Oncol 2012; 5 (1): 56–65. doi: 10.1593/tlo.11202.
41. Cusanelli E, Chartrand P. Telomeric repeat-containing RNA TERRA: a noncoding RNA connecting telomere biology to genome integrity. Front Genet 2015; 6 : 143. doi: 10.3389/fgene.2015.00143.
42. Durand X, Moutereau S, Xylinas E et al. Progensa™ PCA3 test for prostate cancer. Exp Rev Mol Diagn 2011; 11 (2): 137–144. doi: 10.1586/erm.10.122.
43. MyBioSource.com. PCA3 elisa kit: human prostate cancer antigen 3 (PCA3) ELISA kit. [online]. Available from: https: //www.mybiosource.com/pca3-human-elisa-kits/prostate-cancer-antigen-3-pca3/109022.
44. Yang Q, Wan Q, Zhang L et al. Analysis of LncRNA expression in cell differentiation. RNA Biol 2018; 15 (3): 413–422. doi: 10.1080/15476286.2018.1441665.
45. Novogene.com. lncRNA SEQUENCING. [online]. Available from: https: //en.novogene.com/next-generation-sequencing-services/gene-regulation/lncrna-sequencing/.
46. sequencing.roche.com. SeqCap lncRNA Probes. [online]. Available from: https: //sequencing.roche.com/en/products-solutions/by-category/target-enrichment/hybridization/seqcap-lncrna.html.
47. illumina.com. rRNA & Globin mRNA removal kit selection guide. [online]. Available from: https: //www.illumina.com/products/selection-tools/rrna-depletion-selection-guide.html.
48. bio-rad.com. lncRNA RT-qPCR workflow. [online]. Available from: http: //www.bio-rad.com/en-cz/category/lncrna-rt-qpcr-workflow?ID=OUJ92R15.
49. Coassin SR, Orjalo AV, Semaan SJ et al. Simultaneous detection of nuclear and cytoplasmic RNA variants utilizing Stellaris® RNA fluorescence in situ hybridization in adherent cells. In: Nielsen BS (ed). In Situ Hybridization Protocols. New York: Springer 2014 : 189–199.
50. Islam MN, Moriam S, Umer M et al. Naked-eye and electrochemical detection of isothermally amplified HOTAIR long non-coding RNA. Analyst 2018; 143 (13): 3021–3028. doi: 10.1039/c7an02109g.
51. Liu F, Li T, Zhang L et al. PAMAM/polyhedral nanogold-modified probes with DNAase catalysis for the amperometric electrochemical detection of metastasis-associated lung adenocarcinoma transcript 1. J Biol Eng 2019; 13 (1): 21. doi: 10.1186/s13036-019-0149-4.
52. Kim T, Cui R, Jeon YJ et al. Long-range interaction and correlation between MYC enhancer and oncogenic long noncoding RNA CARLo-5. Proc Natl Acad Sci USA 2014; 111 (11): 4173–4178. doi: 10.1073/pnas.1400350111.
53. Léveillé N, Melo CA, Rooijers K et al. Genome-wide profiling of p53-regulated enhancer RNAs uncovers a subset of enhancers controlled by a lncRNA. Nat Commun 2015; 6 : 6520. doi: 10.1038/ncomms7520.
54. Lian Y, Yan C, Xu H et al. A novel lncRNA, LINC00460, affects cell proliferation and apoptosis by regulating KLF2 and CUL4A expression in colorectal cancer. Mol Ther Nucl Acid 2018; 12 : 684–697. doi: 10.1016/ j.omtn.2018.06.012.
55. Zhao J, Du P, Cui P et al. LncRNA PVT1 promotes angiogenesis via activating the STAT3/VEGFA axis in gastric cancer. Oncogene 2018; 37 (30): 4094–4109. doi: 10.1038/s41388-018-0250-z.
56. Munschauer M, Nguyen CT, Sirokman K et al. The NORAD lncRNA assembles a topoisomerase complex critical for genome stability. Nature 2018; 561 (7721): 132–136. doi: 10.1038/s41586-018-0453-z.
57. Huang D, Chen J, Yang L et al. NKILA lncRNA promotes tumor immune evasion by sensitizing T cells to activation-induced cell death. Nat Immunol 2018; 19 (10): 1112–1125. doi: 10.1038/s41590-018-0207-y.
58. Zhang X, Rice K, Wang Y et al. Maternally expressed gene 3 (MEG3) noncoding ribonucleic acid: isoform structure, expression, and functions. Endo-crinology 2010; 151 (3): 939–947. doi: 10.1210/en.2009-0657.
59. Svoboda M, Slyskova J, Schneiderova M et al. HOTAIR long non-coding RNA is a negative prognostic factor not only in primary tumors, but also in the blood of colorectal cancer patients. Carcinogenesis 2014; 35 (7): 1510–1515. doi: 10.1093/carcin/bgu055.
60. Li X, Wu Z, Mei Q et al. Long non-coding RNA HOTAIR, a driver of malignancy, predicts negative prognosis and exhibits oncogenic activity in oesophageal squamous cell carcinoma. Br J Cancer 2013; 109 (8): 2266–2278. doi: 10.1038/bjc.2013.548.
61. Okugawa Y, Toiyama Y, Hur K et al. Metastasis-associated long non-coding RNA drives gastric cancer development and promotes peritoneal metastasis. Carcinogenesis 2014; 35 (12): 2731–2739. doi: 10.1093/carcin/bgu200.
62. Guo W, Dong Z, Bai Y et al. Associations between polymorphisms of HOTAIR and risk of gastric cardia adenocarcinoma in a population of north China. Tumor Biol 2015; 36 (4): 2845–2854. doi: 10.1007/s13277-014-2912-y.
63. Crawford ED, Rove KO, Trabulsi EJ et al. Diagnostic performance of PCA3 to detect prostate cancer in men with increased prostate specific antigen: a prospective study of 1,962 cases. J Urol 2012; 188 (5): 1726–1731. doi: 10.1016/j.juro.2012.07.023.
64. Roobol MJ, Schröder FH, van Leeuwen P et al. Performance of the prostate cancer antigen 3 (PCA3) gene and prostate-specific antigen in prescreened men: exploring the value of PCA3 for a first-line diagnostic test. Eur Urol 2010; 58 (4): 475–481. doi: 10.1016/j.eururo.2010.06. 039.
65. Ochiai A, Okihara K, Kamoi K et al. Clinical utility of the prostate cancer gene 3 (PCA3) urine assay in Japanese men undergoing prostate biopsy. BJU Int 2013; 111 (6): 928–933. doi: 10.1111/j.1464-410X.2012.11683.x.
66. Ji P, Diederichs S, Wang W et al. MALAT-1, a novel noncoding RNA, and thymosin β4 predict metastasis and survival in early-stage non-small cell lung cancer. Oncogene 2003; 22 (39): 8031–8041. doi: 10.1038/sj.onc.1206928.
67. Wang Z, Katsaros D, Biglia N et al. High expression of long non-coding RNA MALAT1 in breast cancer is associated with poor relapse-free survival. Breast Cancer Res Treat 2018; 171 (2): 261–271. doi: 10.1007/s10549-018-4839-2.
Štítky
Detská onkológia Chirurgia všeobecná Onkológia
Článek Editorial 2019
Článok vyšiel v časopiseKlinická onkologie
Najčítanejšie tento týždeň
2019 Číslo Supplementum 3- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Kombinace metamizol/paracetamol v léčbě pooperační bolesti u zákroků v rámci jednodenní chirurgie
- Antidepresivní efekt kombinovaného analgetika tramadolu s paracetamolem
- Fixní kombinace paracetamol/kodein nabízí synergické analgetické účinky
-
Všetky články tohto čísla
- CRISPR-Cas9 jako nástroj v terapii nádorových onemocnění
- Syntetická letalita – její současné využití a potenciál v onkologické léčbě
- Pokroky ve využití organokovových sloučenin při vývoji protinádorových léčiv
- Editorial 2019
- Souhrn aktuálních poznatků o úloze estrogenového receptoru α v nádorové buněčné signalizaci
- Glykoproteiny v sérech onkologických pacientů
- Glykozylácia ako dôležitý regulátor funkcie protilátok
- Výskum ubikvitinácie proteínov v onkológii
- Méně časté mutace EGFR v kontextu léčby nemalobuněčného karcinomu plic
- Dlouhé nekódující RNA – současné možnosti detekce a využití v klinické praxi
- Interakce onkogenních virových proteinů s proteiny rodiny p53
- Kooperace genomických, transkriptomických a proteomických metod v detekci mutovaných proteinů
- Klinická onkologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Výskum ubikvitinácie proteínov v onkológii
- CRISPR-Cas9 jako nástroj v terapii nádorových onemocnění
- Glykozylácia ako dôležitý regulátor funkcie protilátok
- Méně časté mutace EGFR v kontextu léčby nemalobuněčného karcinomu plic
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



