-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Vznik a význam sekvenčních variant mikroRNA v kancerogenezi
The development and significance of microRNA sequence variants in carcinogenesis
Background: MicroRNA (miRNA) are a class of short non-coding RNAs that regulate gene expression at the posttranscriptional level. They are involved in key cellular processes and development as well as progression of many diseases. Their levels reflect the physiological state of organisms; therefore, the expression profiles of these molecules are analyzed in biomarker studies. Due to their properties, miRNA appear to be promising diagnostic, prognostic and predictive biomarkers of cancer. Recent studies indicate the existence of sequence variants in miRNA, so-called isomiRs, which differ from the annotated miRNAs by altered sequences due to posttranscriptional modifications. These isomiRs may have a higher abundance than canonical miRNA. The characterization of isomiRs reveals their regulated distribution and different biological properties and thus suggest the possible biological significance of the modifications. The presence of isomiRs can also significantly affect the results of biomarker studies. Currently, the research is focused on their possible clinical significance.
Purpose: The aim of this review is to provide an overview of current knowledge about sequence variants in miRNA. The review summarizes the mechanisms of isomiRs biogenesis and describes the effects of sequence heterogeneity on miRNA stability, function and analysis. Subsequently, the role of isomiRs in biomarker studies is discussed.
Keywords:
isomiRs – mikroRNA – sequence variants – diagnostics – biomarkers
Autori: Mgr. Pifková Lucie 1; Mgr. Veselá Petra 1; prof. RNDr. Slabý Ondřej, Ph.D. 1,2
Pôsobisko autorov: Středoevropský technologický institut, MU Brno 1; MOÚ Brno 2
Vyšlo v časopise: Klin Onkol 2021; 34(1): 20-25
Kategória: Přehled
doi: https://doi.org/10.48095/ccko202120Súhrn
Východiska: MikroRNA (miRNA) jsou jednou ze skupin krátkých nekódujících RNA, které na posttranskripční úrovni regulují genovou expresi. Jsou zapojeny do klíčových buněčných procesů a podílejí se na vzniku a progresi mnohých onemocnění. Hladiny miRNA mohou odrážet fyziologický stav organizmů, a proto jsou v rámci biomarkerových studií analyzovány jejich expresní profily. Díky svým vlastnostem se jeví jako slibné diagnostické, prognostické a prediktivní biomarkery nádorových onemocnění. Nedávné studie poukazují na existenci sekvenčních variant miRNA, tzv. izomiRs, které se od anotovaných miRNA liší pozměněnou sekvencí v důsledku posttranskripčních modifikací. Tyto izomiRs mohou vykazovat vyšší zastoupení než některé kanonické miRNA. Bližší studium izomiRs odhaluje jejich nenáhodnou distribuci i odlišné biologické vlastnosti a poukazuje tak na možný biologický význam modifikací sekvence. Přítomnost izomiRs může také zásadně ovlivňovat výsledky biomarkerových studií. V současné době je pozornost zaměřena na jejich možný klinický význam.
Cíl: Cílem této práce je poskytnout přehled současných poznatků o sekvenčních variantách miRNA. V práci jsou popsány mechanizmy vzniku izomiRs a vliv heterogenity v sekvenci na stabilitu, funkci a analýzu miRNA. Prostor je věnován také roli izomiRs v biomarkerových studiích.
Klíčová slova:
izomiRs – mikroRNA – sekvenční varianty – diagnostika – biomarkery
Úvod
MikroRNA (miRNA) jsou skupinou krátkých nekódujících RNA, které se podílejí na posttranskripční regulaci genové exprese. Tuto funkci zprostředkovávají díky komplementaritě k nepřekládaným oblastem na 3’ konci mRNA, ke kterým navádějí efektorový komplex zvaný miRISC. Samotný komplex miRISC tvoří kromě miRNA také různé proteiny, z nichž nejvýznamnější jsou argonautové (Ago) proteiny. Regulace se v závislosti na stupni komplementarity může uskutečňovat dvěma mechanizmy, kterými jsou degradace mRNA nebo represe translace. MikroRNA se uplatňují v signálních drahách asociovaných s významnými buněčnými procesy, jakými jsou proliferace, diferenciace či apoptóza [1]. Doposud bylo popsáno 2 654 lidských maturovaných miRNA a sekvence jejich kanonických forem jsou anotovány v databázi miRBase [2]. Deregulace hladin specifických miRNA jsou asociovány s konkrétními onemocněními, a proto jsou expresní profily miRNA v rámci biomarkerových studií analyzovány za účelem bližší charakterizace a molekulární klasifikace nádorů [3], určení prognózy onemocnění [4] nebo predikce odpovědi na konkrétní léčbu [5]. Pokrok v oblasti analýzy miRNA, zejména implementace metod sekvenování nové generace, vedly ke zjištění, že příslušné miRNA nejsou zastoupeny pouze kanonickou formou, ale také dalšími sekvenčními variantami. Tyto sekvenční varianty zvané izomiRs se od kanonických miRNA liší v primární struktuře a vznikají posttranskripčními úpravami nebo nepřesným štěpením v průběhu maturace [6]. Jejich variabilita spočívá obvykle v adici či deleci 1–2 nukleotidů [7]. O významu izomiRs vypovídá jejich relativně vysoké zastoupení, které se liší v závislosti na typu tkáně a vývojovém [8] či fyziologickém stavu dané tkáně [9].
Sekvenční varianty miRNA (izomiRs)
Klasifikace izomiRs zatím není oficiálně sjednocena, avšak autoři prací zabývajících se touto problematikou se shodují v jejich rozdělení na templátové a netemplátové izomiRs. Zatímco u templátových izomiRs dochází ke změně pouze v délce sekvence, v případě netemplátových izomiRs nemusí sekvence dané varianty plně odpovídat sekvenci kódované v genomu. Pro charakterizaci izomiRs je dále zásadní stanovení, zda se variabilní nukleotidy nacházejí na 3’ konci, 5’ konci nebo uvnitř sekvence. Častější je výskyt 3’ variant [10], což je pravděpodobně důsledkem odlišných tlaků na konzervovanost 3’ a 5’ konce. Zachování sekvence 5’ konce souvisí se specifitou dané miRNA, protože se v této oblasti nachází tzv. seed sekvence, která je klíčová pro komplementaritu ke specifické mRNA [11]. Dalším aspektem podílejícím se na vyšší konzervovanosti 5’ konce může být uspořádání a vazba domén Ago proteinů. Zatímco 5’ konec je účinněji chráněn před působením exonukleáz díky pevné vazbě MID domény Ago proteinu, 3’ konec s navázanou PAZ doménou podléhá štěpení exonukleázami snáze [10,12]. Samotné Ago proteiny na 5’ konci navíc preferují konkrétní báze nukleotidů a změna v sekvenci 5’ konce se tak projeví na efektivitě vazby k Ago proteinům a začlenění do miRISC komplexu [12].
Mechanizmy vzniku izomiRs
Pro vznik izomiRs bylo popsáno několik mechanizmů, které probíhají na posttranskripční úrovni. Templátové izomiRs vznikají především nepřesným štěpením prekurzorů miRNA endonukleázami Drosha a Dicer [13,14], které se při kanonické biogenezi přirozeně podílejí na maturaci miRNA [15,16]. Heterogenita konců miRNA má častěji původ v nepřesném štěpení Dicerem [17]. Štěpení Dicerem je přesné, pouze pokud se ve vzdálenosti dvou nukleotidů od místa štěpení nachází jednořetězcová sekvence tvořící strukturu vlásenky či výdutě. V opačném případě dochází následkem nepřesného štěpení ke vzniku 5’ variant [18]. Heterogenita sekvencí je vysvětlována také přítomností asymetrických strukturních motivů, které obvykle zvětšují rozsah štěpení miRNA [14]. Významnou roli hraje dále dsRNA vazebný protein TRBP, který může měnit místo štěpení Dicerem [19]. IzomiRs vznikají i v průběhu alternativní biogeneze, která se vyznačuje maturací miRNA nezávisle na štěpení Droshou nebo Dicerem. V tomto případě dochází k nepřesnému zkrácení prostřednictvím neznámé 5’-3’ exonukleázy a RNA exozomu [20] nebo Ago2 proteinu s ribonukleázovou aktivitou [21]. Na vzniku templátových izomiRs se dále podílejí exonukleázy, které štěpí fosfodiesterové vazby nukleových kyselin a zkracují především 3’ konec miRNA [22]. Za vznikem netemplátových izomiRs stojí především nukleotidyltransferázy, které ke 3’ konci připojují nukleotidy s adenylovými či uracilovými bázemi. Doposud byla schopnost modifikovat miRNA popsána u sedmi lidských nukleotidyltransferáz [23]. Další skupinou enzymů podílejících se na vzniku netemplátových izomiRs jsou RNA editační enzymy ADAR a CDAR. Zajišťují především záměnu adeninu za guanin a cytosinu za uracil u prekurzorových i maturovaných miRNA a dávají tak vzniknout tzv. substitučním izomiRs [6,10]. Jednotlivé mechanizmy vzniku izomiRs v průběhu maturace shrnuje obr. 1.
Obr. 1. Mechanizmy vzniku izomiRs v průběhu maturace. 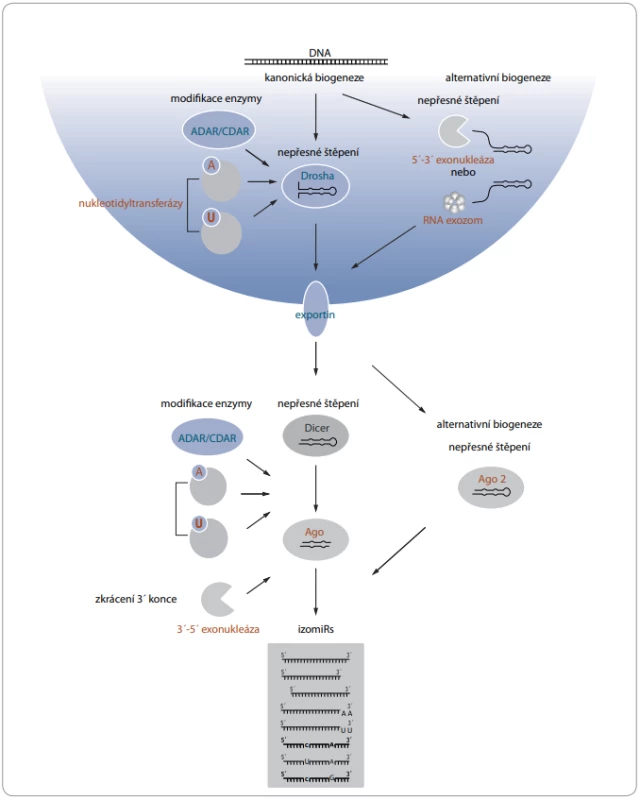
A – adenin, ADAR/CDAR – editační enzymy, AGO/AGO 2 – argonautové proteiny, Dicer/Drosha – endonukleázy, izomiRs – sekvenční varianty miRNA, U – uracil Vliv modifikací na biologické vlastnosti miRNA
Konkrétní modifikace v sekvenci miRNA se projevují na jejich biologických vlastnostech a ovlivňují i samotný průběh maturace. Záměna nukleotidů způsobená působením editačních enzymů vede ke změně v párovacích vlastnostech a změně struktury prekurzorových miRNA, což může snížit efektivitu zpracování endonukleázami Drosha a Dicer [24,25]. Nukleotidyltransferázy prostřednictvím uridylace mění velikost přesahu 3’ konce, který je klíčový pro rozeznání prekurzoru Dicerem. V závislosti na velikosti uridylovaného přesahu dochází důsledkem této modifikace ke zvýšení či snížení efektivity zpracování prekurzoru [26,27]. Heterogenita 5’ konce může vést ke změně termodynamické stability miRNA duplexu a tím ovlivnit výběr řetězce, který bude začleněn do RISC komplexu [13]. Modifikace se projevují také na distribuci miRNA. Bylo pozorováno, že adenylované miRNA zůstávají uvnitř buňky, a naopak uridylované miRNA jsou ve vyšší míře sekretovány, obvykle formou exozomů [28]. Bylo také pozorováno, že se jednotlivé izoformy dané miRNA liší ve stabilitě. Vysvětlením je skutečnost, že konkrétní sekvenční motivy jednotlivých miRNA jsou jedním z faktorů určujících stabilitu miRNA a jakákoliv sekvenční změna v těchto motivech může ovlivnit jejich stabilitu [29,30]. Významný je pozitivní vliv adenylace, která chrání 3’ konec variant před působením exonukleáz [31]. Uridylace 3’ konců naopak působí jako degradační signál [32]. Heterogenita v sekvenci se projevuje také na funkci miRNA, a to zejména nachází-li se variabilní nukleotid v oblasti 5’ konce a seed sekvence. Závažnost změny v repertoáru cílových mRNA se u 5’ variant liší v závislosti na tom, do jaké míry se u konkrétní interakce s mRNA uplatňuje centrální párování [33], které probíhá mezi 4. a 15. nukleotidem a nahrazuje funkci seed sekvence [34]. Byly pozorovány případy, kdy na změnu v repertoáru měly vliv i modifikace 3’ konce [35]. Přestože izomiRs cílí na odlišné mRNA, často regulují stejné signální dráhy jako jejich kanonické formy [36]. Změny v sekvencích dále ovlivňují volbu vazby k jednomu ze čtyř typů Ago proteinů [37,38], přičemž jednotlivé typy se mohou lišit s ohledem na schopnost ribonukleázové aktivity [39], zastoupení v jednotlivých tkáních [40] a jak studie naznačují, tak i specifitou regulace signálních drah [41].
Analýza izomiRs
Pro studium izomiRs se obvykle využívají stejné přístupy, jaké se uplatňují při analýze kanonických miRNA. Mezi běžně používané metody patří kvantitativní PCR (qPCR) a dále vysokokapacitní přístupy, jako microarrays a PCR arrays. Všechny zmíněné metody jsou založeny na hybridizaci, vyžadují tedy předchozí znalost cílové sekvence. Použití těchto metod, zejména qPCR, se při analýze izomiRs vyznačuje určitými limitacemi, které jsou dány vysokou vzájemnou podobností sekvencí [42,43]. Pro předejití nepřesným výsledkům zapříčiněným přítomností variant jsou v posledních letech běžně používané metody modifikovány za účelem zvýšení specificity. Např. použití nově vyvíjené modifikace PCR, tzv. dumbbelPCR (dbPCR), slibuje detekci specifických izomiRs. Metoda dbPCR vyžaduje 3’ i 5’ komplementaritu primerů a tím dosahuje vyšší přesnosti a lepšího rozlišení jednotlivých izomiRs než běžné qPCR platformy [44]. Odlišným přístupem je sekvenování nové generace, které umožňuje analyzovat nukleové kyseliny bez předchozí znalosti sekvence. Tento přístup umožňuje detekci všech izoforem a podává tak nejpřesnější informace o zastoupení jednotlivých izomiRs [45].
Pokud je existence izomiRs opomíjena, může vést k chybné interpretaci výsledků studií zaměřených na charakterizaci expresních profilů miRNA [46]. Při bioinformatickém zpracování sekvenačních dat bývají často jednotlivé izoformy přiřazeny pod název jejich kanonické formy, čímž dochází ke ztrátě informací o zastoupení izomiRs [47]. Důsledkem mohou být neúspěšné validace získaných dat [48] a rozdílné výsledky nezávislých studií [42]. Pro validaci sekvenačních dat se obvykle využívá analýza pomocí qPCR. Podobně jako ostatní hybridizační metody, qPCR však bývá navržena pro detekci pouze kanonických miRNA. V těchto případech sice dochází i k detekci sekvenčních variant, avšak s odlišnou efektivitou, a pokud sekvenční změny neumožňují účinnou hybridizaci navržených oligonukleotidů, jsou jejich hladiny podhodnoceny [42,46,49]. Je-li kanonická varianta zastoupena jen minoritně, může dojít až k selhání detekce dané miRNA a v důsledku toho i k neúspěšné validaci [50]. Získání přesnějších informací o zastoupení miRNA již při bioinformatickém zpracování sekvenačních dat nabízí některé z bioinformatických nástrojů, které klasifikaci izomiRs umožňují [51–55], problémem je však chybějící konsenzus týkající se parametrů těchto nástrojů [56].
Klinický význam izomiRs
Díky vysoké četnosti výskytu některých izomiRs se začíná obracet pozornost na jejich zastoupení při identifikaci a validaci potenciálních biomarkerů. Recentní studie ukazují, že analýza specifických izomiRs může o fyziologickém stavu poskytovat přesnější informace než jejich kanonické varianty. Hinton et al při využití NGS přístupu detekovali vedle kanonických variant také izomiRs, které umožňovaly přesnější charakterizaci diferenciace buněk [57]. V případě nádorových onemocnění bylo pozorováno, že v nádorové tkáni může převažovat jiná izomiR, než je tomu u tkáně nenádorové. Toto pozorování vedlo k závěru, že specifické izomiRs mohou představovat vhodnější biomarkery než jejich kanonické formy, a to zejména v případech, kdy jsou kanonické miRNA zastoupeny minoritně [48]. Jednou z příčin rozdílného zastoupení jednotlivých izoforem miRNA mezi tkáněmi může být přítomnost miRNA modifikujících enzymů v těchto tkáních. Popsána byla např. změna hladin nukleotidyltransferáz ve tkáni nádorů prostaty [23] a RNA editačních enzymů u melanomů [58]. Studie Watahikiho et al z roku 2011 byla jednou z prvních prací naznačujících diagnostický potenciál izomiRs, jejichž exprese se lišila mezi tkání metastáz a primárních nádorů prostaty na xenograftním myším modelu [59]. Koppers Lalic et al docílili pomocí izomiRs vyšší specifity při neinvazivní detekci karcinomu prostaty z moči. Zatímco validace kanonických variant miR‑21, miR‑375 a miR‑204 pomocí qPCR nepotvrdila diagnostický potenciál těchto miRNA, při detekci konkrétních 3’ izomiRs bylo možné rozlišit pacienty od kontrolní skupiny se senzitivitou 72,9 %, specificitou 88 % a AUC 0,866 [48]. Zhang et al identifikovali na základě sekvenačních dat dostupných z databáze TCGA izomiRs, jejichž hladiny byly významně deregulované u invazivního karcinomu prsu. Škála těchto sekvenčních variant příslušné miRNA umožňovala rozlišit nádorovou tkáň od nenádorové za vyšší hodnoty AUC než při využití samostatných kanonických miRNA [60].
Prognostický význam izomiRs je demonstrován na sekvenčních variantách miR-200b. Editovaná varianta miR-200b se liší od kanonické formy schopností podporovat buněčnou invazi a migraci díky snížené schopnosti tlumit expresi genů ZEB1 a ZEB2. Na rozdíl od kanonické miR-200b tato varianta také negativně reguluje nádorový supresor LIFR [61]. Zatímco kanonická forma miR-200b je asociována s delším celkovým přežitím pacientů [62], vyšší hladiny editované miR-200b indikují kratší celkové přežití [61].
Na využití izomiRs při klasifikaci nádorů se zaměřuje práce Telonise et al z roku 2017. Ze studie vyplývá, že na základě přítomnosti či nepřítomnosti 7 466 izomiRs a miRNA (tzv. binárních profilů) v dané tkáni lze efektivně klasifikovat nádory do 32 tříd dle TCGA (The Cancer Genome Atlas) s průměrnou senzitivitou 90 % a mírou falešných objevů (false discovery rate – FDR) 3 %. Autoři dále pozorovali, že vybrané miRNA generovaly sekvenční varianty jen u určitých typů nádorových onemocnění, např. miR-9 byla výrazně zastoupena sekvenčními variantami pouze u low-grade gliomů [63].
Závěr
Prací zaměřujících se na sekvenční varianty miRNA v současné době významně přibývá a ukazuje se, že analýza izomiRs představuje více informativní přístup jak v biomarkerových studiích, tak v pracích zaměřených na studium role miRNA v kancerogenezi. Komplikací stále zůstává volba platformy, která by umožňovala detekci jednotlivých variant se stejnou efektivitou. V této oblasti byl však zaznamenán pokrok a dochází k vývoji nových či modifikovaných metod detekce miRNA a bioinformatických nástrojů. Pro srovnatelnost výsledků studií však bude nutné parametry pro detekci a klasifikaci izomiRs sjednotit.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
The authors declare they have no potential confl icts of interest concerning drugs, products, or services used in the study.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
The Editorial Board declares that the manuscript met the ICMJE recommendation for biomedical papers.
prof. RNDr. Ondřej Slabý, Ph.D.
Masarykův onkologický ústav
Žlutý kopec 7
656 Brno
e-mail: on.slaby@gmail.com
Obdrženo/Submitted: 13. 9. 2020
Přijato/Accepted: 18. 9. 2020
Zdroje
1. Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 2004; 116 (2): 281–297. doi: 10.1016/s0092-8674 (04) 00045-5.
2. Kozomara A, Birgaoanu M, Griffiths-Jones S. miRBase: from microRNA sequences to function. Nucleic Acids Res 2019; 47 : 155–162. doi: 10.1093/nar/gky1141.
3. Lu J, Getz G, Miska EA et al. MicroRNA expression profiles classify human cancers. Nature 2005; 435 (7043): 834–838. doi: 10.1038/nature03702.
4. Calin GA, Liu CG, Sevignani C et al. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci USA 2004; 101 (32): 11755–11760. doi: 10.1073/pnas.0404432101.
5. Blower PE, Verducci, JS, Lin S et al. MicroRNA expression profiles for the NCI-60 cancer cell panel. Mol Cancer Ther 2007; 6 (5): 1483–1491. doi: 10.1158/1535-7163.MCT-07-0009.
6. Morin RD, O‘Connor MD, Griffith M et al. Application of massively parallel sequencing to microRNA profiling and discovery in human embryonic stem cells. Genome Res 2008; 18 (4): 610–621. doi: 10.1101/gr.7179508.
7. Guo L, Chen F. A challenge for miRNA: multiple isomiRs in miRNAomics. Gene 2014; 544 (1): 1–7. doi: 10.1016/j.gene.2014.04.039.
8. Fernandez-Valverde SL, Taft RJ, Mattick JS. Dynamic isomiR regulation in Drosophila development. RNA 2010; 16 (10): 1881–1888. doi: 10.1261/rna.2379610.
9. Salem O, Erdem N, Jung J et al. The highly expressed 5‘ isomiR of hsa-miR-140-3p contributes to the tumor--suppressive effects of miR-140 by reducing breast cancer proliferation and migration. BMC Genomics 2016; 17 : 566. doi: 10.1186/s12864-016-2869-x.
10. Neilsen CT, Goodall GJ, Bracken CP. IsomiRs – the overlooked repertoire in the dynamic microRNAome. Trends Genet 2012; 28 (11): 544–549. doi: 10.1016/j.tig.2012.07.005.
11. Lewis BP, Shih IH, Jones-Rhoades MW et al. Prediction of mammalian microRNA targets. Cell 2003; 115 (7): 787–798. doi: 10.1016/s0092-8674 (03) 01018-3.
12. Frank F, Sonenberg N, Nagar B. Structural basis for 5‘-nucleotide base-specific recognition of guide RNA by human AGO2. Nature 2010; 465 (7299): 818–822. doi: 10.1038/nature09039.
13. Wu H, Ye C, Ramirez D et al. Alternative processing of primary microRNA transcripts by Drosha generates 5‘ end variation of mature microRNA. PLoS One 2009; 4 (10): e7566. doi: 10.1371/journal.pone.0007566.
14. Starega-Roslan J, Krol J, Koscianska E et al. Structural basis of microRNA length variety. Nucleic Acids Res 2011; 39 (1): 257–268. doi: 10.1093/nar/gkq727.
15. Lee Y, Ahn C, Han J et al. The nuclear RNase III Drosha initiates microRNA processing. Nature 2003; 425 (6956): 415–419. doi: 10.1038/nature01957.
16. Hutvágner G, McLachlan J, Pasquinelli AE et al. A cellular function for the RNA-interference enzyme Dicer in the maturation of the let-7 small temporal RNA. Science 2001; 293 (5531): 834–838. doi: 10.1126/science.1062961.
17. Starega-Roslan J, Koscianska E, Kozlowski P et al. The role of the precursor structure in the biogenesis of microRNA. Cell Mol Life Sci 2011; 68 (17): 2859–2871. doi: 10.1007/s00018-011-0726-2.
18. Gu S, Jin L, Zhang Y et al. The loop position of shRNAs and pre-miRNAs is critical for the accuracy of dicer processing in vivo. Cell 2012; 151 (4): 900–911. doi: 10.1016/j.cell.2012.09.042.
19. Fukunaga R, Han BW, Hung JH et al. Dicer partner proteins tune the length of mature miRNAs in flies and mammals. Cell 2012; 151 (4): 912. doi: 10.1016/j.cell.2012; 10.029.
20. Ameres SL, Zamore PD. Diversifying microRNA sequence and function. Nat Rev Mol Cell Biol 2013; 14 (8): 475–488. doi: 10.1038/nrm3611.
21. Liang T, Yu J, Liu C et al. IsomiR expression patterns in canonical and Dicer-independent microRNAs. Mol Med Rep 2017; 15 (3): 1071–1078. doi: 10.3892/mmr.2017.6117.
22. Han BW, Hung JH, Weng Z et al. The 3‘-to-5‘ exoribonuclease Nibbler shapes the 3‘ ends of microRNAs bound to Drosophila Argonaute1. Curr Biol 2011; 21 (22): 1878–1887. doi: 10.1016/j.cub.2011.09.034.
23. Wyman SK, Knouf EC, Parkin RK et al. Post-transcriptional generation of miRNA variants by multiple nucleotidyl transferases contributes to miRNA transcriptome complexity. Genome Res 2011; 21 (9): 1450–1461. doi: 10.1101/gr.118059.110.
24. Yang W, Chendrimada TP, Wang Q et al. Modulation of microRNA processing and expression through RNA editing by ADAR deaminases. Nat Struct Mol Biol 2006; 13 (1): 13–21. doi: 10.1038/nsmb1041.
25. Kawahara Y, Zinshteyn B, Chendrimada TP et al. RNA editing of the microRNA-151 precursor blocks cleavage by the Dicer-TRBP complex. EMBO Rep 2007; 8 (8): 763–769. doi: 10.1038/sj.embor.7401011.
26. Heo I, Joo C, Kim YK et al. TUT4 in concert with Lin28 suppresses microRNA biogenesis through premicroRNA uridylation. Cell 2009; 138 (4): 696–708. doi: 10.1016/j.cell.2009.08.002.
27. Heo I, Ha M, Lim J et al. Mono-uridylation of pre-microRNA as a key step in the biogenesis of group II let-7 microRNAs. Cell 2012; 151 (3): 521–532. doi: 10.1016/j.cell.2012.09.022.
28. Koppers-Lalic D, Hackenberg M, Bijnsdorp IV et al. Nontemplated nucleotide additions distinguish the small RNA composition in cells from exosomes. Cell Rep 2014; 8 (6): 1649–1658. doi: 10.1016/j.celrep.2014.08.027.
29. Bail S, Swerdel M, Liu H et al. Differential regulation of microRNA stability. RNA 2010; 16 (5): 1032–1039. doi: 10.1261/rna.1851510.
30. Rissland OS, Hong SJ, Bartel DP. MicroRNA destabilization enables dynamic regulation of the miR-16 family in response to cell-cycle changes. Mol Cell 2011; 43 (6): 993–1004. doi: 10.1016/j.molcel.2011.08.021.
31. Katoh T, Sakaguchi Y, Miyauchi K et al. Selective stabilization of mammalian microRNAs by 3‘ adenylation mediated by the cytoplasmic poly (A) polymerase GLD-2. Genes Dev 2009; 23 (4): 433–438. doi: 10.1101/gad.1761509.
32. Jones MR, Quinton LJ, Blahna MT et al. Zcchc11-dependent uridylation of microRNA directs cytokine expression. Nat Cell Biol 2009; 11 (9): 1157–1163. doi: 10.1038/ncb1931.
33. Martin HC, Wani S, Steptoe AL et al. Imperfect centered miRNA binding sites are common and can mediate repression of target mRNAs. Genome Biol 2014; 15 (3): R51. doi: 10.1186/gb-2014-15-3-r51.
34. Shin C, Nam JW, Farh KK et al. Expanding the microRNA targeting code: functional sites with centered pairing. Mol Cell 2010; 38 (6): 789–802. doi: 10.1016/j.molcel.2010.06.005.
35. Burroughs AM, Ando Y, de Hoon MJ et al. A comprehensive survey of 3‘ animal miRNA modification events and a possible role for 3‘ adenylation in modulating miRNA targeting effectiveness. Genome Res 2010; 20 (10): 1398–1410. doi: 10.1101/gr.106054.110.
36. Cloonan N, Wani S, Xu Q et al. MicroRNAs and their isomiRs function cooperatively to target common biological pathways. Genome Biol 2011; 12 (12): R126. doi: 10.1186/gb-2011-12-12-r126.
37. Azuma-Mukai A, Oguri H, Mituyama T et al. Characterization of endogenous human Argonautes and their miRNA partners in RNA silencing. Proc Natl Acad Sci USA 2008; 105 (23): 7964–7969. doi: 10.1073/pnas.0800334105.
38. Burroughs AM, Ando Y, de Hoon MJ et al. Deep-sequencing of human Argonaute-associated small RNAs provides insight into miRNA sorting and reveals Argonaute association with RNA fragments of diverse origin. RNA Biol 2011; 8 (1): 158–177. doi: 10.4161/rna.8.1.14300.
39. Rand TA, Petersen S, Du F et al. Argonaute2 cleaves the anti-guide strand of siRNA during RISC activation. Cell 2005; 123 (4): 621–629. doi: 10.1016/j.cell.2005.10.020.
40. Petri S, Dueck A, Lehmann G et al. Increased siRNA duplex stability correlates with reduced off-target and elevated on-target effects. RNA 2011; 17 (4): 737–749. doi: 10.1261/rna.2348111.
41. Modzelewski AJ, Holmes RJ, Hilz S et al. AGO4 regulates entry into meiosis and influences silencing of sex chromosomes in the male mouse germline. Dev Cell 2012; 23 (2): 251–264. doi: 10.1016/j.devcel.2012.07.003.
42. Leshkowitz D, Horn-Saban S, Parmet Y et al. Differences in microRNA detection levels are technology and sequence dependent. RNA 2013; 19 (4): 527–538. doi: 10.1261/rna.036475.112.
43. Magee R, Telonis AG, Cherlin T et al. Assessment of isomiR discrimination using commercial qPCR methods. Non-coding RNA 2017; 3 (2): 18. doi: 10.3390/ncrna3020018.
44. Honda S, Kirino Y. Dumbbell-PCR: a method to quantify specific small RNA variants with a single nucleotide resolution at terminal sequences. Nucleic Acids Res 2015; 43 (12): e77. doi: 10.1093/nar/gkv218.
45. Chugh P, Dittmer DP. Potential pitfalls in microRNA profiling. Wiley Interdiscip Rev DNA 2012; 3 (5): 601–616. doi: 10.1002/wrna.1120.
46. Lee LW, Zhang S, Etheridge A et al. Complexity of the microRNA repertoire revealed by next-generation sequencing. RNA 2010; 16 (11): 2170–2180. doi: 10.1261/rna.2225110.
47. Wu CW, Evans JM, Huang S et al. A comprehensive approach to sequence-oriented IsomiR annotation (CASMIR): demonstration with IsomiR profiling in colorectal neoplasia. BMC Genomics 2018; 19 (1): 401. doi: 10.1186/s12864-018-4794-7.
48. Koppers-Lalic D, Hackenberg M, de Menezes R et al. Non-invasive prostate cancer detection by measuring miRNA variants (isomiRs) in urine extracellular vesicles. Oncotarget 2016; 7 (16): 22566–22578. doi: 10.18632/oncotarget.8124.
49. Schamberger A, Orbán TI. 3‘ IsomiR species and DNA contamination influence reliable quantification of microRNAs by stem-loop quantitative PCR. PLoS One 2014; 9 (8): e106315. doi: 10.1371/journal.pone.0106315.
50. Nassirpour R, Mathur S, Gosink MM et al. Identification of tubular injury microRNA biomarkers in urine: comparison of next-generation sequencing and qPCR-based profiling platforms. BMC Genomics 2014; 15 (1): 485. doi: 10.1186/1471-2164-15-485.
51. Pantano L, Estivill X, Martí E. SeqBuster, a bioinformatic tool for the processing and analysis of small RNAs datasets, reveals ubiquitous miRNA modifications in human embryonic cells. Nucleic Acids Res 2010; 38 (5): e34. doi: 10.1093/nar/gkp1127.
52. Shi J, Dong M, Li L et al. mirPRo-a novel standalone program for differential expression and variation analysis of miRNAs. Sci Rep 2015; 5 : 14617. doi: 10.1038/srep14617.
53. Zhang Y, Zang Q, Zhang H et al. DeAnnIso: a tool for online detection and annotation of isomiRs from small RNA sequencing data. Nucleic Acids Res 2016; 44 (W1): W166–175. doi: 10.1093/nar/gkw427.
54. Urgese G, Paciello G, Acquaviva A et al. isomiR-SEA: an RNA-Seq analysis tool for miRNAs/isomiRs expression level profiling and miRNA-mRNA interaction sites evaluation. BMC Bioinf 2016; 17 : 148. doi: 10.1186/s12859-016-0958-0.
55. De Oliveira LF, Christoff AP, Margis R. isomiRID: a framework to identify microRNA isoforms. Bioinformatics 2013; 29 (20): 2521–2523. doi: 10.1093/bioinformatics/btt424.
56. Desvignes T, Loher P, Eilbeck K et al. Unification of miRNA and isomiR research: the mirGFF3 format and the mirtop API. Bioinformatics 2020; 36 (3): 698–703. doi: 10.1093/bioinformatics/btz675.
57. Hinton A, Hunter SE, Afrikanova I et al. sRNA-seq analysis of human embryonic stem cells and definitive endoderm reveals differentially expressed microRNAs and novel IsomiRs with distinct targets. Stem cells 2014; 32 (9): 2360–2372. doi: 10.1002/stem.1739.
58. Shoshan E, Mobley AK, Braeuer RR et al. Reduced adenosine-to-inosine miR-455-5p editing promotes melanoma growth and metastasis. Nat Cell Biol 2015; 17 (3): 311–321. doi: 10.1038/ncb3110.
59. Watahiki A, Wang Y, Morris J et al. MicroRNAs associated with metastatic prostate cancer. PLoS One 2011; 6 (9): e24950. doi: 10.1371/journal.pone.0024950.
60. Zhang H, Yang S, Guo L et al. Comparisons of isomiR patterns and classification performance using the rank-based MANOVA and 10-fold crossvalidation. Gene 2015; 569 (1): 21–26. doi: 10.1016/j.gene.2014.11.026.
61. Wang Y, Xu X, Yu S et al. Systematic characterization of A-to-I RNA editing hotspots in microRNAs across human cancers. Genome Res 2017; 27 (7): 1112–1125. doi: 10.1101/gr.219741.116.
62. Hu X, Macdonald DM, Huettner PC et al. A miR-200 microRNA cluster as prognostic marker in advanced ovarian cancer. Gynecol Oncol 2009; 114 (3): 457–464. doi: 10.1016/j.ygyno.2009.05.022.
63. Telonis AG, Magee R, Loher P et al. Knowledge about the presence or absence of miRNA isoforms (isomiRs) can successfully discriminate amongst 32 TCGA cancer types. Nucleic Acids Res 2017; 45 (6): 2973–2985. doi: 10.1093/nar/gkx082.
Štítky
Detská onkológia Chirurgia všeobecná Onkológia
Článek Aktuality z odborného tiskuČlánek Zemřela Mgr. Jana Drexlerová
Článok vyšiel v časopiseKlinická onkologie
Najčítanejšie tento týždeň
2021 Číslo 1- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Kombinace metamizol/paracetamol v léčbě pooperační bolesti u zákroků v rámci jednodenní chirurgie
- Antidepresivní efekt kombinovaného analgetika tramadolu s paracetamolem
- Srovnání analgetické účinnosti metamizolu s ibuprofenem po extrakci třetí stoličky
-
Všetky články tohto čísla
- Patnáct let Národního onkologického programu České republiky a jeho vliv na výsledky u solidních nádorů dospělých v přehledu a číslech
- Rehabilitácia pri rakovine prsníka
- Vznik a význam sekvenčních variant mikroRNA v kancerogenezi
- Zárodečné mutace v genech RAD51C a RAD51D a dědičná predispozice ke vzniku karcinomu ovaria
- Droplet digital PCR as a novel diagnostic tool
- Odkládání návštěvy lékaře při podezření na karcinom prsu u žen v České republice
- Pathological response and clinical outcomes in operable triple-negative breast cancer with cisplatin added to standard neoadjuvant chemotherapy
- Post-treatment persistent alpha-fetoprotein elevation in a patient with testicular cancer
- Pembrolizumab v 1. linii terapie generalizovaného spinocelulárního karcinomu jícnu
- Skvamocelulárny karcinóm rekta u mladej ženy
- Four years of low dose enzalutamide for metastatic castration-resistant prostate cancer
- Selhání adjuvantní léčby maligního melanomu – a co dál?
- Aktuality z odborného tisku
- Král Z, Adam Z et al. Histiocytární neoplazie a další vybrané velmi vzácné krevní nemoci.
- Zemřela Mgr. Jana Drexlerová
- Klinická onkologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Rehabilitácia pri rakovine prsníka
- Zárodečné mutace v genech RAD51C a RAD51D a dědičná predispozice ke vzniku karcinomu ovaria
- Droplet digital PCR as a novel diagnostic tool
- Skvamocelulárny karcinóm rekta u mladej ženy
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



