Využití PCR-HRMA při přímé detekci a identifikaci původců dermatofytóz z klinických vzorků
Use of PCR-HRMA for Direct Detection and Identification of Dermatophytosis Agents from Clinical Samples
The aim of the study was to test the utility of the PCR method in combination with High Resolution Melting Analysis (HRMA) for the detection and identification of dermatophytes directly from clinical skin and adnexa samples. The methodology should reduce the time between sampling and diagnosis, increase the diagnostic sensitivity, and overall improve patient care. In the study, 128 clinical samples from patients with suspected dermatomycosis were analyzed. To isolate fungal DNA from the clinical specimens, a KIT ZR Fungal/Bacterial DNA MiniPrepTM was used. Two sets of primers specific for a wide range of dermatophytes were used to detect ribosomal DNA regions. The real-time PCR High Resolution Melting Analysis (PCR-HRMA) method was used for dermatophyte species identification. The PCR detection success was 74% for both sets of primers, the PCR-HRMA method enabled dermatophyte species identification in all PCR positive cases. In contrast, only 52% of patients with dermatophytosis were culture positive. Microscopy from the sample was positive in 90% of patients with proven dermatophytosis. 90% of dermatophytes were successfully identified using microscopy, culture and PCR-HRMA. The most common dermatophyte species (Trichophyton rubrum, T. interdigitale, T. benhamiae) were reliably detected by this methodology. PCR detection of rDNA directly from clinical material using HRMA increased the number of species identificacions in the diagnosis of dermatophytosis. The combination of classical and molecular biologic examinations appears to be a suitable method for the rapid and reliable diagnosis of dermatophytosis.
Keywords:
Trichophyton benhamiae – dermatophytes – direct identification – ribosomal DNA – Trichophyton rubrum – Trichophyton interdigitale
Authors:
R. Dobiáš 1,2; M. Kantorová 3; P. Jaworská 1; P. Hamal 2; J. Mrázek 3
Authors‘ workplace:
Oddělení bakteriologie a mykologie, Centrum klinických laboratoří, Zdravotní ústav se sídlem v Ostravě, vedoucí oddělení RNDr. Vladislav Holec
1; Ústav mikrobiologie, Lékařská fakulta Univerzity Palackého v Olomouci, přednosta ústavu prof. MUDr. Milan Kolář, Ph. D.
2; Oddělení molekulární biologie, Centrum klinických laboratoří, Zdravotní ústav se sídlem v Ostravě, vedoucí oddělení Mgr. Jakub Mrázek
3
Published in:
Čes-slov Derm, 93, 2018, No. 6, p. 259-265
Category:
Clinical and laboratory Research
Overview
Cílem studie bylo otestovat použitelnost metody PCR v kombinaci s High Resolution Melting Analysis (HRMA) pro detekci a identifikaci dermatofytů přímo z klinických vzorků kůže a jejích adnex. Metodika by měla vést ke zrychlení a zvýšení výtěžnosti diagnostiky dermatomykóz a ke zkvalitnění péče o pacienta. Ve studii bylo analyzováno 128 klinických vzorků od pacientů s podezřením na dermatomykózu. Pro izolaci DNA dermatofytů z klinického materiálu byl použit izolační KIT ZR Fungal/Bacterial DNA MiniPrepTM. Pro detekci DNA oblastí kódujících ribozomální RNA byly využity dvě sady primerů zachycující široké spektrum dermatofytů. Pro druhovou identifikaci dermatofytů byla použita real-time PCR metoda v kombinaci s High Resolution Melting Analysis (PCR-HRMA).
Dermatofytóza byla potvrzena u 42 ze 128 zkoumaných vzorků (pozitivní přímá mikroskopie, kultivace, detekce PCR-HRMA, nebo kombinace metod). Výtěžnost PCR detekce byla 74 % pro obě sady primerů, metoda HRMA umožnila ve všech případech druhovou identifikaci detekovaných dermatofytů pomocí PCR. Naproti tomu kultivace byla pozitivní pouze u 52 % pacientů s dermatofytózou. Mikroskopie ze vzorku byla pozitivní v 90 % pozitivních dermatofytóz. Při současném použití mikroskopie, kultivace a PCR-HRMA bylo úspěšně identifikováno na úroveň druhu 90 % dermatofytů. Nejčastější druhy dermatofytů (Trichophyton rubrum, T. interdigitale, T. benhamiae) byly touto metodikou spolehlivě zachyceny. PCR detekce rDNA přímo z klinického materiálu s využitím HRMA zvýšila výtěžnost diagnostiky dermatofytóz zvláště tím, že přispěla k dosažení druhové identifikace u výrazně vyššího počtu případů v porovnání s konvenčními metodami. Kombinace konvenčních a molekulárně biologických vyšetření se zdá být vhodná pro rychlou a spolehlivou diagnostiku dermatofytóz bez nutnosti výrazného navýšení nákladů.
Klíčová slova:
Trichophyton benhamiae – dermatofyty – přímá identifikace – ribozomální DNA
ÚVOD
Infekce způsobené dermatofyty jsou jedny z nejčastějších kožních infekcí na světě a náklady spojené s jejich diagnostikou a léčbou každoročně přesahují 400 milionů dolarů [2]. Celosvětová prevalence dermatomykóz je 20–25 % [14]. Nejčastěji se vyskytujícím onemocněním způsobeným dermatofyty v rozvinutých zemích je tinea unguium, jejíž prevalence se v evropské populaci odhaduje na 12,4 % [19], zatímco ve vyšších věkových skupinách může dosahovat až 50 % [21]. Podle poslední epidemiologické studie založené na kultivačně pozitivních případech dermatofytóz, jsou nejběžnějšími klinickými jednotkami v České republice tinea unguium (55,5 %), dále tinea corporis (29,2 %), tinea pedis (14,6 %) a tinea capitis (0,7 %). Dominantní druhy dermatofytů ve spektru původců dermatomykóz u nás jsou Trichophyton rubrum (79,2 %), T. benhamiae (7,2 %), T. interdigitale (7,1 %) a Microsporum canis (3,6 %) [16].
Klasické metody pro detekci a identifikaci dermatofytů z klinických vzorků kůže zahrnují přímou mikroskopii klinického materiálu a identifikaci narostlých kultur mikro - a makromorfologicky. Časová náročnost kompletní diagnostiky dermatomykóz je minimálně 10–15 dní a často však až 3–4 týdny. Mikroskopické a kultivační metody mají nízkou senzitivitu, 50–70 % [4], proto je nutné odběr klinického vzorku mnohdy opakovat, aby bylo dosaženo zvýšení záchytu původce dermatomykózy, a tím se čas mezi odběrem vzorku a vlastní diagnózou prodlužuje. Diagnostiku dermatomykózy je možné urychlit a zvýšit její výtěžnost pomocí detekce DNA původce přímo z klinického materiálu metodou PCR [3, 5, 22]. Real-time PCR metoda kombinovaná s následnou analýzou křivek tání s vysokým rozlišením (High Resolution Melting Analysis; PCR-HRMA) se zdá být slibnou alternativou pro současnou diagnostickou praxi [9], a to zejména z hlediska citlivosti metody, finanční efektivity, ale také díky možnosti přímo identifikovat základní druhy původců. Tato metoda se mimo jiné také používá k rychlé typizaci lékařsky významných virů a bakterií, pro identifikaci druhů rodu Candida, zástupců řádu Mucorales apod. [13, 17, 18].
V této studii jsme testovali použitelnost metodiky přímé detekce DNA dermatofytů pomocí PCR v klinických vzorcích v kombinaci s HRMA, umožňující identifikaci běžných patogenů na úroveň druhu. Cílem studie bylo ověření efektivity metody PCR-HRMA při identifikaci dermatofytů a zrychlení diagnostiky dermatofytóz pro rutinní praxi.
MATERIÁL A METODY
Klinický materiál
Do studie byly zahrnuty všechny klinické vzorky (kůže a adnexa) běžně zasílaného pro rutinní diagnostiku suspektních dermatomykóz do Laboratoře klinické mykologie Zdravotního ústavu se sídlem v Ostravě ze spádové oblasti Moravskoslezského kraje v období 20. 3. 2017 až 20. 7. 2017. Na uvedeném pracovišti byly klinické vzorky zpracovány a podrobeny rutinní detekci a identifikaci dermatofytů zahrnující přímou mikroskopii klinického materiálu a identifikaci narostlých kultur mikromorfologicky a makromorfologicky. Vyšetřovaný materiál zahrnoval nehtové šupiny a podnehtovou drť, kožní šupiny a vlasy.
Molekulárně biologické metody
Ve studii bylo analyzováno 128 vzorků od pacientů s podezřením na dermatomykózu. Pro izolaci rDNA mikromycetů z klinického materiálu byl použit izolační KIT ZR Fungal/Bacterial DNA MiniPrepTM (Zymo Research, Californie, USA). Izolace probíhala podle pokynů výrobce. Prvním krokem bylo rozrušení vzorků klinického materiálu pomocí zirkoniových kuliček. Následovala izolace DNA pomocí pufrů a izolačních kolonek. Takto získaná DNA byla použita pro real-time PCR.
K detekci rDNA byly využity dvě PCR metody využívající dvojice primerů zachycující široké spektrum dermatofytů. První metoda byla zacílena na amplifikaci ITS1-5,8S oblasti, kdežto druhá amplifikovala celou oblast ITS rDNA (ITS1-5,8S-ITS2). Sekvence použitých primerů a sond jsou uvedeny v tabulce 1. Obě analýzy byly provedeny na přístroji LightCycler 96 (Roche, Basel, Switzerland).

Amplifikace úseku ITS1-5,8S byla provedena při následujících podmínkách: PCR reakce byla provedena při finálním objemu 20 µl, směs obsahovala 5 µl DNA z klinického vzorku, 15 µl reakčního mixu, který obsahoval 10 µl TaqMan® Fast Advanced Master Mix (Thermo Fisher Scientific, Waltham, USA), 10 pmol každého z dvojice primerů, a 4 pmol TaqMan sondy; teplotní profil amplifikace zahrnoval aktivaci uracil-DNA-glykosylázy (50°C/2 min), úvodní denaturaci 95 °C/20 s a 40 amplifikačních cyklů sestávajících z denaturace 95 °C/3 s, annealingu včetně elongace a měření signálu při 60 °C/30 s. Hodnocení reakce bylo kvalitativní, pozitivní izoláty byly dále postoupeny druhové identifikaci metodou real-time PCR-HRMA.
Pro druhovou identifikaci nejvýznamnějších druhů byla použita real-time PCR-HRMA oblasti ITS1-5,8S-ITS2. Amplifikace úseku ITS1-5,8S-ITS2 byla provedena při následujících podmínkách: PCR reakce byla provedena při finálním objemu 20 µl, směs obsahovala 5 µl DNA z klinického vzorku, 15 µl reakčního mixu, který obsahoval 12,5 µl Type-it HRM PCR Master Mix 2× (Qiagen, Hilden, Germany) s fluorescenčním markerem EvaGreen, 17,5 pmol každého z dvojice primerů; teplotní profil amplifikace zahrnoval úvodní denaturaci 95 °C/5 min a 45 amplifikačních cyklů sestávajících z denaturace 95 °C/10 s, annealingu 55° C/30 s a elongace 72 °C/10 s. Následná HRMA analýza probíhala rychlostí 0,05 °C/s v rozsahu 65–95 °C a kontinuálním čtením fluorescence (20 čtení/s). Hodnocení reakce je umožněno porovnáním křivek tání se standardy pro druhy Trichophyton rubrum, T. benhamiae, T. interdigitale a T. tonsurans. Vzorky, které byly pozitivní, ale nevykazovaly pík shodný s použitými standardy, byly dále identifikovány pomocí sekvenační analýzy [5, 9].
VÝSLEDKY
Ve 42 případech (33 %) ze 128 odběrů vzorků klinicky suspektních na mykotickou etiologii byla diagnóza potvrzena buď konvenčními metodami (přímá mikroskopie, kultivace), metodou PCR-HRMA, nebo jejich kombinací. Data o prokázaných případech dermatofytóz jsou shrnuta v tabulkách 2 a 3. Ze všech 42 potvrzených dermatofytóz byla zastoupena tinea unguium ve 28 případech (67 %), tinea corporis v 8 (19 %), tinea pedis v 5 (12 %) a tinea faciei v 1 případě (2 %).
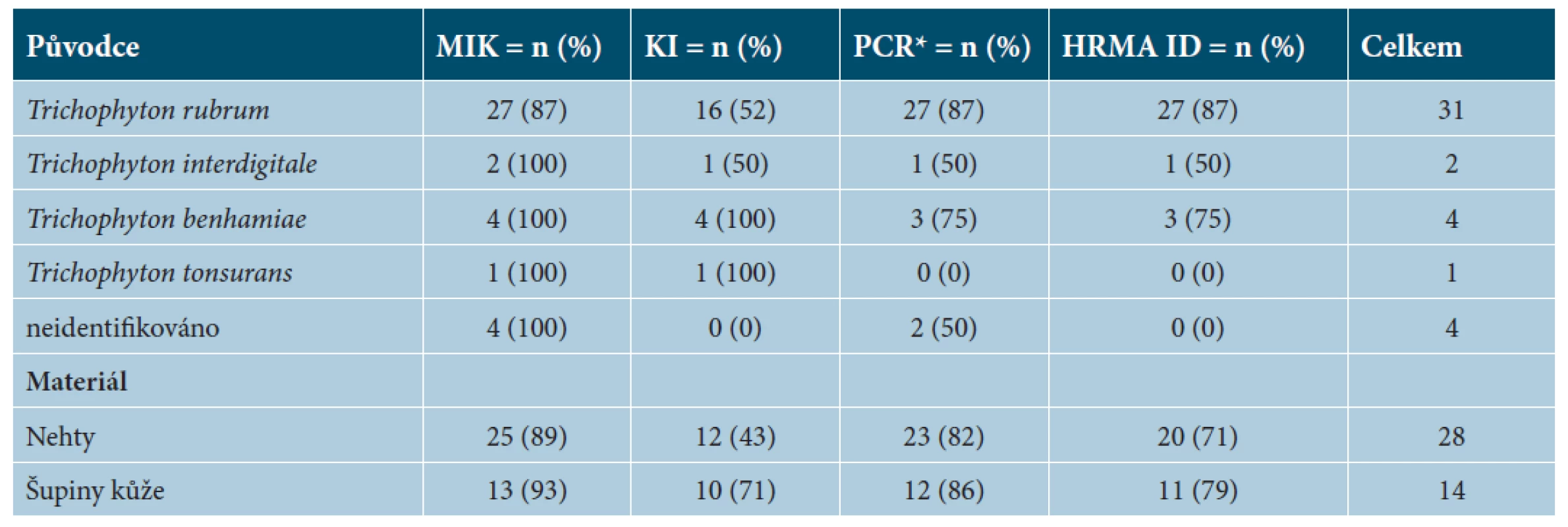
Pozitivní nález v přímé mikroskopii byl přítomen u 38 ze 42 (90 %) potvrzených případů dermatofytózy. Běžně používanou kombinací metod přímé mikroskopie a následné kultivace bylo identifikováno pouze 22 ze 42 (52 %) původců dermatofytózy. Žádné mikroskopicky negativní klinické vzorky nebyly kultivačně pozitivní (viz tab. 2 a 3). PCR detekce byla úspěšná ve 31 případech ze 42 (74 %) pro obě použité metody, metoda HRMA umožnila ve všech případech druhovou identifikaci PCR pozitivních nálezů. V našem souboru 128 pacientů zvýšilo současné použití mikroskopie, kultivace a PCR-HRMA úspěšnost diagnostiky dermatofytóz z 38 na celkový počet 42 případů. Druhové identifikace bylo dosaženo u 38 ze 42 případů (90 %), oproti 22 případům (52 %) za použití kultivace a následné fenotypové identifikace.
S využitím přímé detekce dermatofytů z klinického materiálu metodou PCR-HRMA se zvýšila úspěšnost identifikace původců o 22 % oproti klasické identifikaci (kultivace, makro - a mikromorfologie). Výsledek je patrný z grafu 1. Nejčastější původci v našem souboru vzorků, T. rubrum, a T. benhamiae, byly touto metodikou spolehlivě zachyceny a identifikovány pomocí typických křivek tání vynesených v časovém diagramu – graf 2. Trichophyton interdigitale byl detekován pomocí PCR-HRMA v jednom ze dvou případů zastoupených v našem souboru (viz tab. 2); typická křivka tání dovede tento druh jednoznačně odlišit od obou jmenovaných druhů.


DISKUSE A ZÁVĚR
Ačkoliv identifikace dermatofytů pomocí makro - a mi-kromorfologických znaků je považována za zlatý standard, nemusí být jednoduchá a spolehlivá [6, 8, 11, 15]. V diagnostice dermatomykóz proto někteří autoři dokonce od tohoto „zlatého standardu“ ustupují [20]. Správná identifikace druhů dermatofytů je důležitá pro zahájení včasné a cílené terapie dermatomykózy, a také pro detekci zdroje infekce [1, 16]. Vzhledem k existující možnosti terapie azolovými preparáty a značně variabilní citlivosti antropofilních, zoofilních a geofilních dermatofytů k těmto antimykotikům je včasná identifikace původců tohoto onemocnění žádoucí [12]. V nedávných studiích byly jako alternativní metody pro identifikaci dermatofytů použity různé soupravy založené na amplifikaci nukleových kyselin, jako je sekvenování DNA, PCR-RFLP, Nested-PCR, PCR-ELISA a mikročipy (microarray). Tyto metody mají několik výhod oproti klasické přímé mikroskopii a kultivaci klinického materiálu, včetně vyšší citlivosti, rychlosti, a objektivity [7, 10, 22]. Nicméně hlavní nevýhodou těchto metod jsou často jejich vysoké náklady. Vzhledem k tomu, že v praxi je potřeba analyzovat množství vzorků, jsou zapotřebí rychlé a ekonomicky nenáročné metody, mezi které patří i zde studovaná metoda PCR-HRMA. Podobně jako ve studii Spiliopoulou et al. [22], bylo i v této práci pomocí alternativní metody přímé detekce dermatofytů z klinického materiálu dosaženo zvýšení výtěžnosti v detekci a identifikaci dermatomykóz o 22 %, a při současném použití klasických metod a PCR-HRMA bylo úspěšně identifikováno 90 % původců dermatomykózy.
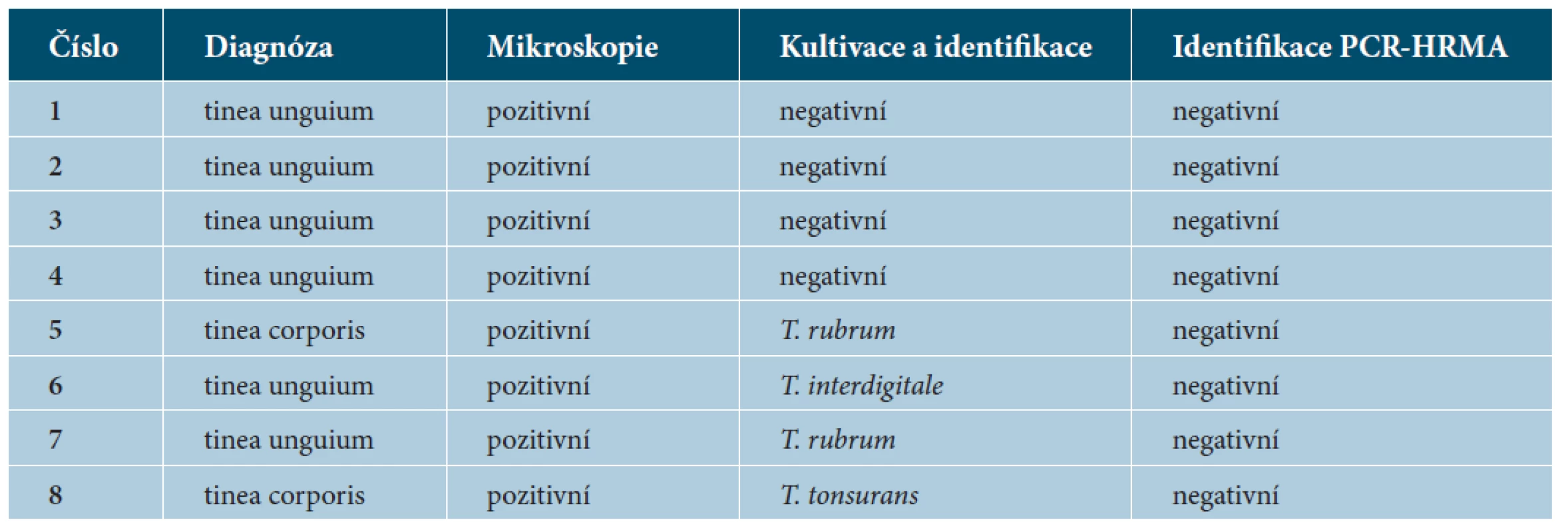
Metoda PCR-HRMA se zdá být dobře využitelná pro přímou detekci a identifikaci dermatofytů z klinického materiálu v rutinní praxi. Kombinace klasických a molekulárně biologických vyšetření se jeví jako vhodná metodika pro rychlou a spolehlivou diagnostiku dermatomykóz. Ve čtyřech případech nedošlo k úspěšné kultivaci dermatofytů, ani detekci metodou HRMA, přestože přímá mikroskopie klinického materiálu byla pozitivní. V sedmi případech se detekce a identifikace patogena metodou PCR-HRMA nezdařila, přestože přímá mikroskopie i kultivace byla pozitivní. Tato situace mohla vzniknout následkem nedostatečné homogenity vzorku. Poloviční efektivita je patrná i v případě identifikace T. interdigitale (viz tab. 2), přestože typická křivka tání dovede tento druh jednoznačně odlišit od jiných druhů (viz graf 2). Tento problém proto rovněž přikládáme nedostatečné homogenitě klinického vzorku použitého pro kultivaci a PCR detekci. Řešením by mělo být vypracování metodiky pro lepší homogenizaci vzorku před samotnou izolací DNA, která by měla zásadně zvýšit výtěžnost této metody. Je proto nutno dále rozvíjet metodiku přípravy vzorků z tohoto náročného klinického materiálu, jakým je kůže a deriváty, zejména nehty. Nezbytné je zdůraznit, že diverzita dermatofytických hub není vzhledem k menšímu počtu použitých klinických vzorků v krátkém sledovaném období příliš rozmanitá, nicméně kopíruje epidemiologickou situaci v České republice [16]. Vzhledem k tomu, že prospektivní studie dále pokračuje, bude zdokonalení tohoto postupu a rozšíření souboru sledovaných vzorků publikováno v budoucnu.
Poděkování
Studie byla podpořena projekty Ministerstva zdravotnictví České republiky (AZV 17-31269A) a Interní grantovou agenturou Univerzity Palackého v Olomouci (IGA_LF_2017_031).
Do redakce došlo dne 13. 4. 2018
Adresa pro korespondenci:
Mgr. Radim Dobiáš
Oddělení bakteriologie a mykologie
Centrum klinických laboratoří
Zdravotní ústav se sídlem v Ostravě
Partyzánské náměstí 7
702 00 Ostrava
e-mail: radim.dobias@zuova.cz
Sources
1. AGHAMIRIAN, M. R., GHIASIAN, S. A. Dermatophytoses in outpatients attending the Dermatology Center of Avicenna Hospital in Qazvin, Iran. Mycoses, 2008, 51, p. 155–160
2. AMEEN, M. Epidemiology of superficial fungal infections. Clin Dermatol, 2010, 28, p. 197–201.
3. BERGMAN, A., HEIMER, D., KONDORI, N., ENROTH, H. Fast and specific dermatophyte detection by automated DNA extraction and real-time PCR. Clin Microbiol Infect, 2013, 19, p. 205–211.
4. BERGMANS, A. M., SCHOULS, L. M., VAN DER ENT, M., KLAASSEN, A., BOHM, N., WINTERMANS, R. G. Validation of PCR-reverse line blot, a method for rapid detection and identification of nine dermatophyte species in nail, skin and hair samples. Clin Microbiol Infect, 2008, 14, p. 778–788.
5. BERGMANS, A. M., VAN DER ENT, M., KLAASSEN, A., BOHM, N., ANDRIESSE, G. I., WINTERMANS, R. G. Evaluation of a single-tube real-time PCR for detection and identification of 11 dermatophyte species in clinical material. Clin Microbiol Infect, 2010, 16, p. 704–710.
6. ČMOKOVÁ, A., HAMAL, P., SVOBODOVÁ, L., HUBKA, V. Detekce, identifikace a typizace dermatofytů molekulárně genetickými metodami. Čes-slov Derm, 2014, 89, p. 175–186.
7. DE BAERE, T., SUMMERBELL, R., THEELEN, B., BOEKHOUT, T., VANEECHOUTTE, M. Evaluation of internal transcribed spacer 2-RFLP analysis for the identification of dermatophytes. J Med Microbiol, 2010, 59, p. 48–54.
8. DE HOOG, G. S., DUKIK, K., MONOD, M. et al. Toward a Novel Multilocus Phylogenetic Taxonomy for the Dermatophytes. Mycopathologia, 2017, 182, p. 5–31.
9. DIDEHDAR, M., KHANSARINEJAD, B., AMIR-RAJAB, N., SHOKOHI, T. Development of a high--resolution melting analysis assay for rapid and high-throughput identification of clinically important dermatophyte species. Mycoses, 2016, 59, p. 442–449.
10. ELAVARASHI, E., KINDO, A. J., KALYANI, J. Optimization of PCR-RFLP Directly from the Skin and Nails in Cases of Dermatophytosis. Targeting the ITS and the 18S Ribosomal DNA Regions. J Clin Diagn Res, 2013, 7, p. 646–651.
11. FAGGI, E., PINI, G., CAMPISI, E., BERTELLINI, C., DIFONZO, E., MANCIANTI, F. Application of PCR to distinguish common species of dermatophytes. J Clin Microbiol, 2001, 39, p. 3382–3385.
12. FAVRE, B., HOFBAUER, B., HILDERING, K. S., RYDER, N. S. Comparison of in vitro activities of 17 antifungal drugs against a panel of 20 dermatophytes by using a microdilution assay. J Clin Microbiol, 2003, 41, p. 4817–4819.
13. FORTINI, D., CIAMMARUCONI, A., DE SANTIS, R., FASANELLA, A., BATTISTI, A., D’AMELIO, R., LISTA, F., CASSONE, A., CARATTOLI, A. Optimization of high-resolution melting analysis for low-cost and rapid screening of allelic variants of Bacillus anthracis by multiple-locus variable-number tandem repeat analysis. Clin Chem, 2007, 53, p. 1377–1380.
14. HAVLICKOVA, B., CZAIKA, V. A., FRIEDRICH, M. Epidemiological trends in skin mycoses worldwide. Mycoses, 2008, 51, p. 2–15.
15. HUBKA, V., ČMOKOVÁ, A., SKOŘEPOVÁ, M. et al. Současný vývoj v taxonomii dermatofytů a doporučení pro pojmenovávání klinicky významných druhů. Čes-slov Derm, 2014, 89, p. 151–165.
16. HUBKA, V., VĚTROVSKÝ, T., DOBIÁŠOVÁ, S.et al. Molekulární epidemiologie dermatofytóz v České republice–výsledky dvouleté studie. Čes-slov Derm, 2014, 89, p. 167–174.
17. LENGEROVA, M., RACIL, Z., HRNCIROVA, K. et al. Rapid detection and identification of mucormycetes in bronchoalveolar lavage samples from immunocompromised patients with pulmonary infiltrates by use of high-resolution melt analysis. J Clin Microbiol, 2014, 52, p. 2824–2828.
18. LIN, J. H., TSENG, C. P., CHEN, Y. J. Rapid differentiation of influenza A virus subtypes and genetic screening for virus variants by high-resolution melting analysis. J Clin Microbiol, 2008, 46, p. 1090–1097.
19. MORIARTY, B., HAY, R., MORRIS-JONES, R. The diagnosis and management of tinea. BMJ, 2012, 345, p. 4380.
20. OHST, T., KUPSCH, C., GRASER, Y. Detection of common dermatophytes in clinical specimens using a simple quantitative real-time TaqMan polymerase chain reaction assay. Br J Dermatol, 2016, 174, p. 602–609.
21. SALGO, P., DANIEL, C., GUPTA, A., MOZENA, J., JOSEPH, S. Onychomycosis disease management. Medical Crossfire: debates, peer exchange and insights in medicine, 2003, 4, p. 1–17.
22. SPILIOPOULOU, A., BARTZAVALI, C., JELASTOPULU, E., ANASTASSIOU, E. D., CHRISTOFIDOU, M. Evaluation of a commercial PCR test for the diagnosis of dermatophyte nail infections. J Med Microbiol, 2015, 64, p. 25–31.
Labels
Dermatology & STDs Paediatric dermatology & STDsArticle was published in
Czech-Slovak Dermatology

2018 Issue 6
-
All articles in this issue
- Efektivní léčebná modulace poškozujícího zánětu u nemocných s psoriázou guselkumabem cílícím na specifickou podjednotku p19 IL-23 regulační osy IL-23/Th17
- Identifikace dermatofytů pomocí MALDI-TOF hmotnostní spektrometrie
- Využití PCR-HRMA při přímé detekci a identifikaci původců dermatofytóz z klinických vzorků
- Možnosti terapie onychomykózy nízkoteplotním plazmatem
- Zoonotické dermatofytózy: klinický obraz, diagnostika, etiologie, léčba, epidemiologická situace u nás
- Pět případů lidských dermatofytóz vyvolaných zoofilním druhem Trichophyton erinacei přeneseným z ježků
- Czech-Slovak Dermatology
- Journal archive
- Current issue
- About the journal
Most read in this issue
- Zoonotické dermatofytózy: klinický obraz, diagnostika, etiologie, léčba, epidemiologická situace u nás
- Pět případů lidských dermatofytóz vyvolaných zoofilním druhem Trichophyton erinacei přeneseným z ježků
- Identifikace dermatofytů pomocí MALDI-TOF hmotnostní spektrometrie
- Možnosti terapie onychomykózy nízkoteplotním plazmatem

