Minimální reziduální nemoc – možnosti detekce u hematologických i nehematologických malignit
Minimal residual disease – detection possibilities in haematological and non-haematological malignancies
Minimal residual disease (MRD) monitoring is of a great importance for the patients and often determines their future therapy. At present, MRD detection in patients suffering from hematologic malignancies is included in most of treatment protocols. However, it appears that the MRD detection, sometimes described as circulating tumor cells or minimal disseminated disease, is important in patients with solid tumors as well. Molecular MRD monitoring principles include detection of specific DNA, RNA or protein markers of tumour cells, which are not present in bone marrow and/or peripheral blood cells. High specificity and sensitivity of this specific molecular tumour marker is necessary. Quantitative MRD monitoring by means of molecular methods can determine the decrease or increase of MRD level.
In this review, we describe different molecular methods used, an overview on their advantages, limitations, and the sample quality and processing requirements. A summary of molecular markers employed in hematological and non-hematological diseases is also presented.
Keywords:
minimal residual disease – minimal disseminated disease – circulating tumor cells – PCR – quantitative PCR – Ig/TCR rearrangement
Authors:
Markéta Kalinová
Authors‘ workplace:
Ústav patologie a molekulární medicíny, 2. LF UK a FN Motol, Praha
Published in:
Čes.-slov. Patol., 49, 2013, No. 4, p. 131-138
Category:
Review articles
Overview
Sledování hladiny minimální reziduální nemoci (MRN) má obrovský význam pro pacienta a často rozhoduje o jeho dalším osudu. U pacientů s hematologickými malignitami je sledování MRN již součástí léčebných protokolů, nicméně se ukazuje, že sledování MRN, někdy vyjadřované také jako cirkulující nádorové buňky nebo minimální diseminovaná nemoc, má význam i u pacientů se solidními nádory. Principem molekulárního sledování MRN je najít na nádorové buňce marker na DNA, RNA nebo proteinové úrovni, kterým se bude lišit od normálních buněk kostní dřeně a/nebo periferní krve a který bude možno detekovat s vysokou specifitou a senzitivitou pomocí molekulárních metod. Rozvoj kvantitativních metod detekce MRN nám umožňuje i vyjádřit se k případnému úbytku či nárůstu MRN. V přehledu je pojednáno o jednotlivých molekulárních metodikách detekce MRN, jejich úskalích, výhodách, limitacích a nárocích na vstupní materiál a zpracování. Také je uveden přehled molekulárních markerů hematologických i nehematologických onemocnění.
Klíčová slova:
minimální reziduální nemoc – minimální diseminovaná nemoc – cirkulující nádorové buňky – PCR – kvantitativní PCR – Ig/TCR přestavby
První zmínky o minimální reziduální nemoci (MRN) pochází již ze sedmdesátých let minulého století (1), kdy byla studována MRN u pacientů s akutními leukémiemi. MRN v hematologické problematice, kde se s tímto pojmem setkáváme nejčastěji, je definována jako subklinická úroveň nemoci, kdy v průběhu terapie již maligní buňky nejsou běžnými cytologickými metodami detekovatelné ve vzorku kostní dřeně a/nebo periferní krve pacienta. V této situaci hovoříme o kompletní klinické remisi, přestože může mít pacient v těle až 1010 maligních buněk (2). Toto množství je nadále vysoké a právě tyto přežívající nádorové buňky mohou způsobit relaps onemocnění.
V literatuře se můžeme setkat i s pojmem minimální diseminovaná nemoc (MDN), která je popisována jako MRN u pacientů se solidními nádory, např. s lymfomy, rabdomyosarkomem (RMS), neuroblastomem či Ewingovým sarkomem (3,4). Někdy je také jako MDN označována detekce nádorových buněk v kostní dřeni během diagnózy solidního nádorového onemocnění a MRN jako detekce nádorových buněk v kostní dřeni během léčby onemocnění (5). U pacientů s karcinomy a solidními nádory se přítomnost nádorových buněk v kostní dřeni může také označovat jako mikrometastáza nebo cirkulující nádorové buňky (6,7).
Zjednodušeně lze tedy říci, že MRN je definováno jako přítomnost reziduálních nádorových buněk v kostní dřeni (periferní krvi) u pacientů v morfologické remisi. MRN a její detekce se velmi rychle rozšířila i mimo hematologickou problematiku, kdy např. u pacientů s nádory žaludku, plic nebo mléčné žlázy bývá hlavním důvodem relapsu onemocnění po odstranění primárního nádoru právě přítomnost diseminovaných (cirkulujících) nádorových buněk v kostní dřeni a periferní krvi (8,9). Od počátku 90. let jsou ve větší míře rozvíjeny techniky, které umožňují detekovat tuto submikroskopickou hladinu nádorových buněk.
Detekce MRN má také prognostický význam u mnoha hematologických malignit (ALL, AML, CML, NHL, MM) i solidních nádorů (např. RMS, neuroblastom, Ewingův sarkom, karcinom mléčné žlázy, karcinom prostaty).
Hematologické malignity
Rychlost, s jakou klesá hladina MRN po zahájení léčby, je důležitým znakem, který určuje průběh léčby a riziko vzniku relapsu onemocnění. Některé studie ukazují, že velký význam má sledování kinetiky MRN u leukémií ve více časových bodech, případně před transplantací kostní dřeně (10-17). Během posledních let se značně zvýšil počet studií zabývajících se detekcí MRN v určitých časových bodech léčby, což je důležité pro následná klinická rozhodnutí ohledně zařazení pacientů do specifických léčebných protokolů s určitou intenzitou léčby, případně pro využití nových léčebných modalit (18-21).
Problémem detekce MRN může být variabilita distribuce nízkého počtu maligních buněk v těle pacienta, takže buněčný vzorek nemusí být vždy reprezentativní (22).
Nehematologické malignity
U pacientů se solidními nádory (mimo NHL) se metodika detekce MRN opírá o detekcí cirkulujících nádorových buněk. Někdy se využívá v prvním kroku tzv. obohacení vzorku kostní dřeně nebo periferní krve z důvodu zvýšení senzitivity následných vyšetření. Nejčastěji se používá izolace buněk pomocí hustotního gradientu, kdy dojde k separaci cirkulujících nádorových buněk a mononukleárních buněk ze vzorku. Další možností je izolace buněk dle velikosti, případně imunomagnetická izolace s monoklonálními protilátkami (23,24). Ukazuje se, že detekce cirkulujících nádorových buněk v čase diagnózy a/nebo po odstranění tumoru případně v průběhu terapie je spojena s horší prognózou onemocnění a s metastatickým potenciálem nádorových buněk (3,4,25-28).
Kostní dřeň vs. periferní krev
Pro hodnocení MRN se převážně vyžívají aspiráty kostní dřeně odebrané do EDTA, což je dáno dřeňovým umístěním onemocnění u akutních leukémií a prostředím kostní dřeně, které je vhodné pro osidlování maligními buňkami. Vzorky periferní krve by se však daly odebírat s větší frekvencí, bez traumatizace pacienta odběrem kostní dřeně, kdy navíc vlastní odběr kostní dřeně může být znehodnocen ředěním krve během aspirace. Je mnoho studií zabývajících se porovnáváním hladiny MRN v kostní dřeni a v periferní krvi. Odběr kostní dřeně je pro pacienta mnohem náročnější než odběr periferní krve, avšak ukazuje se, že sledování hladiny MRN u hematologických malignit má větší vypovídající hodnotu v kostní dřeni než v periferní krvi. U pacientů s B-ALL byla MRN detekována častěji v kostní dřeni, než v periferní krvi. Navíc, úroveň MRN v krvi byla obecně nižší, než u simultánně vyšetřeného vzorku kostní dřeně. U pacientů s T-ALL byla ve většině případů MRN v kostní dřeni a periferní krvi srovnatelná. Proto se zdá, že lze MRN u T-ALL sledovat v periferní krvi (29). U pacientů se solidními nádory (např. lymfomy, RMS, Ewingův sarkom, karcinom mléčné žlázy) se MRN (cirkulující nádorové buňky) sleduje ve vzorcích periferní krve a kostní dřeně (3, 4, 25, 30). Zde však může být právě problém s nerovnoměrnou distribucí nádorových buněk osidlujících kostní dřeň.
METODY DETEKCE MRN
Principem detekce MRN je najít u nádorové buňky takovou genetickou změnu, která bude specifická pouze pro nádorovou populaci a tím ji bude odlišovat od buněk nemaligních. K detekci nádorových buněk byly původně využívány kvalitativní a semikvantitativní metody založené na klasické polymerázové řetězové reakci (PCR) (12). Kvalitativní detekce MRN může být vysoce signifikantní (31), dává však pouze limitovanou informaci typu „ano či ne“ a nevypovídá o případné kinetice nádorové zátěže. Rozvoj kvantitativních molekulárních metod, především kvantitativní PCR v reálném čase (RQ-PCR a RT-RQ-PCR) umožnil, aby sledování MRN poskytovalo i informace o dynamice onemocnění.
Kvantitativní detekce MRN může být provedena třemi základními metodami (32):
- pomocí průtokové cytometrie využívající detekci nádorově specifického imunofenotypu
- pomocí PCR techniky využívající detekci nádorově specifické DNA
- pomocí RT-PCR techniky využívající detekci nádorově specifické RNA.
Průtoková cytometrie
Metoda průtokové cytometrie využívá nejčastěji několikabarevnou kombinaci značení antigenů, kdy jsou nádorové buňky detekovány na základě přítomnosti aberantních antigenů, asynchronní exprese nebo atypického množství exprese antigenů či jejich kombinací (2,33,34). Problémem detekce MRN však může být posun v imunofenotypu během léčby onemocnění nebo při relapsu (35,36) a nedostatečná specifita v časových bodech léčby s velkou regenerací kostní dřeně (37). Citlivost imunofenotypizace se pohybuje v řádu 10-3-10-4 což odpovídá detekci jedné nádorové buňky mezi 1 000 - 10 000 zdravými buňkami. Jako výchozí materiál pro vyšetření průtokovou cytometrií jsou používány nativní buňky, případně vitální buňky zamražené v dimethylsulfoxidu (DMSO). Metodice průtokové cytometrie je věnován jiný článek této série.
PCR techniky využívající nádorově specifickou DNA
V případě hematologických malignit vycházejících z B a T lymfocytů je zatím uznávána jako nejspolehlivější metoda kvantifikace přestaveb genů pro imunoglobuliny (Ig) a T-buněčné receptory (TCR), specifických pro daný leukemický/lymfomový klon, pomocí RQ-PCR za využití pacient-specifických primerů. Obrovskou výhodou oproti průtokové cytometrii je stabilita přestaveb na DNA úrovni i v případě měnícího se fenotypu v průběhu terapie a vyšší senzitivita. Tato metoda se stává ve stále větší míře součástí léčebných protokolů, a to i přes svou značnou časovou, finanční i laboratorní náročnost. Z těchto důvodů byla založena skupina ESG-MRD-ALL (European Study Group on Minimal Residual Disease in Acute Lymphoblastic Leukema), nyní EuroMRD (společně ALL a maligní lymfomy), která sdružuje 30 evropských a mimoevropských laboratoří zabývajících se touto problematikou. Pracovní skupina pořádá dvakrát ročně externí kontrolu kvality a vydává metodická doporučení, pravidla a kritéria pro interpretaci výsledků (viz níže). Součásti pracovní skupiny EuroMRD je i laboratoř našeho pracoviště.
VÝBĚR CÍLŮ A URČENÍ KLONALITY
V roce 1999 (BIOMED-1) a následně v roce 2003 (BIOMED-2) byla publikována standardizovaná metodika screeningu přestaveb imunoreceptorových genů. Jedná se o detekci kompletních V-(D)-J a nekompletních D-J přestaveb těžkých a lehkých řetězců Ig (IgH, IgK, IgL) a TCR (TCRβ, TCRα a TCRδ). Protokol BIOMED-2 navíc rozšířil standardizovanou metodiku o detekci i dvou nejčastějších translokací u pacientů s maligními lymfomy: detekce translokace t(11;14) pro pacienty s lymfomem z buněk pláště (MCL) a t(14;18) pro pacienty s folikulárním lymfomem (FL). Dále se sreening Ig/TCR přestaveb rozšířil o „multiplex reakce“ k zachycení detekce klonality i u lymfomů s vysokým zastoupením somatických hypermutací (FL, difúzní velkobuněčný B lymfom - DLBCL) (38,39).
V prvním kroku je tedy třeba analyzovat DNA z diagnostického vzorku s vysokým podílem nádorových buněk a detekovat specifickou Ig/TCR přestavbu daného klonu konkrétního pacienta. U leukémií a lymfomů to bývá infiltrovaná kostní dřeň a/nebo periferní krev, u lymfomů, kde není při diagnóze přítomna infiltrace periferní krve a/nebo kostní dřeně, se používá DNA z primárního nádoru (nejčastěji z lymfatické uzliny postižené lymfomem). Je nutné mít dostatek kvalitní DNA z diagnostického vzorku, proto se doporučuje u suspektních lymfoproliferací odběr nativní tkáně, případně fixace tkáně v 40% alkoholu, neboť fixace v kyselém formaldehydu vede k degradaci nukleových kyselin bez možnosti detekovat molekulární marker onemocnění, což by pacienta eliminovalo z možnosti sledování MRN. Izolace DNA ze vzorku trepanobiopsie je omezena, neboť je nutné po fixaci vzorku ve formaldehydu materiál ještě demineralizovat. Proto je téměř nemožné získat kvalitní a nedegradovanou DNA.
Po proběhnutí „screeningové“ PCR reakce je potřeba rozlišit klony s identickými přechodovými oblastmi od polyklonálních lymfoidních buněk, k čemuž se využívá metoda analýzy heteroduplexů, případně „GeneScanning“ (pro popis viz obr. 1). Po detekci monoklonálního produktu se daná přestavba sekvenuje pro určení přesné sekvence přechodových úseků V-(D)-J oblastí. Získané sekvence lze vyhodnotit pomocí volně přístupných databází na internetu (např. http://ncbi.nlm.nih.gov/igblast, http://imgt.cines.fr).
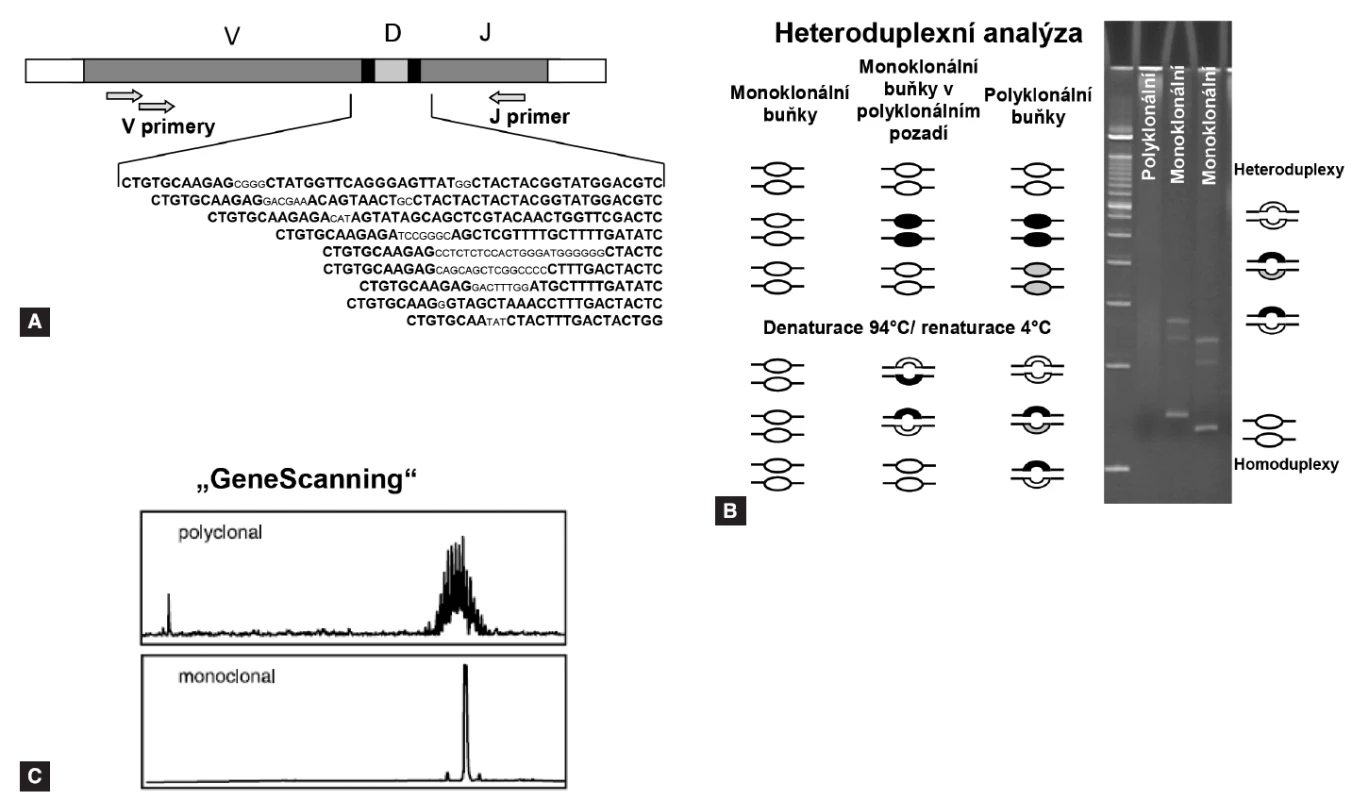
NÁVRH PACIENT-SPECIFICKÝCH PRIMERŮ A JEJICH OPTIMALIZACE
Popis sekvencí a návrh klonálně specifických primerů je vhodné provádět např. v programu VECTOR NTI (Invitrogen). Návrh klonálně specifických primerů (alelově specifické oligonukleotidy - ASO) musí splňovat obecná doporučení a existují pravidla pro jejich sekvenční složení. Je výhodné je navrhovat tak, aby bylo možné využít publikovanou RQ-PCR hydrolyzační sondu a reverzní primer umístěné v konsensus oblasti daného rekombinovaného J, případně D segmentu (40-44). Navržený primer by neměl tvořit vlásenky a dimery sám se sebou ani s reverzním primerem a sondou. Je vhodné pro každou přestavbu navrhovat minimálně dva primery, protože rozdíly v dosažené senzitivitě i specifitě RQ-PCR mohou být velké.
Problémem této metodiky může být detekce oligoklonality při diagnóze. Stanovení MRN je pak problematické, protože klony mohou být zastoupeny v různém poměru a následný relaps onemocnění může nastat z kteréhokoli klonu. Proto se u oligoklonálních onemocnění doporučuje sledovat maximální možné množství klonů, což bývá metodicky velmi obtížné. Zvláštním jevem u leukémií je tzv. liniová promiskuita, která způsobuje přestavování genů pro TCR u B-ALL (TCRα, TCRβ a nekompletní TCRδ) a naopak výskyt nekompletních přestaveb IgH u T-ALL (45). Tento jev umožňuje u leukémií rozšíření spektra sledovaných přestaveb. Podle pravidel ESG-MRD-ALL (Euro-MRD) je požadován pro každého pacienta nejméně jeden systém s minimální citlivostí 10-4 (jedna nádorová buňka mezi 10000 zdravými buňkami). Je vždy lepší sledovat MRN pomocí dvou nezávislých cílů.
OPTIMALIZACE SYSTÉMU A DETEKCE MRN
Pro optimalizaci navrženého primeru a celého systému je nutné sestrojit standardní kalibrační křivku pro přesnou kvantifikaci nádorových buněk v reziduálních vzorcích, neboť se všechny vzorky vztahují a odečítají z této kalibrační křivky specifické pro každého pacienta. Pro tvorbu standardní křivky se používá DNA izolovaná z diagnostického vzorku pacienta se známým počtem nádorových elementů určených např. pomocí průtokové cytometrie nebo klasické histologie ve spojení s imunohistochemií. Tato DNA se logaritmicky ředí do DNA získané z periferní krve zdravých dárců. Doporučuje se používat DNA získanou z periferní krve od minimálně 5 - 10 zdravých dárců, abychom zajistili dostatečně polyklonální pozadí. Standardní kalibrační křivku je vhodné ředit od 10-1 do 10-5. Pro určení koncentrace DNA diagnostického vzorku a později všech reziduálních vzorků se používá kvantifikace kontrolního genu. Většina laboratoří používá gen pro albumin (46). Optimalizace daného systému probíhá pomocí změn teploty nasedání primerů, poměrem koncentrace primerů a hořečnatých iontů, kdy se snažíme dosáhnout co největší specifity a senzitivity. Skupina ESG-MRD-ALL (EuroMRD) nastavila pravidla a kritéria pro senzitivitu, rozmezí kvantifikace a specifiku (viz Příloha č. 1 - v eSupplementu k tomuto článku na www.CSpatologie.cz). Všechny vzorky a standardní kalibrační křivku analyzujeme minimálně v duplikátech. V systému RQ-PCR se hodnotí tzv. „crossing-point“ (Ct, někdy Cp), což je PCR cyklus, ve kterém přechází kinetika reakce do exponenciální fáze amplifikace. Hodnota Ct stoupá s klesající koncentrací templátu ve vzorku (obr. 2).
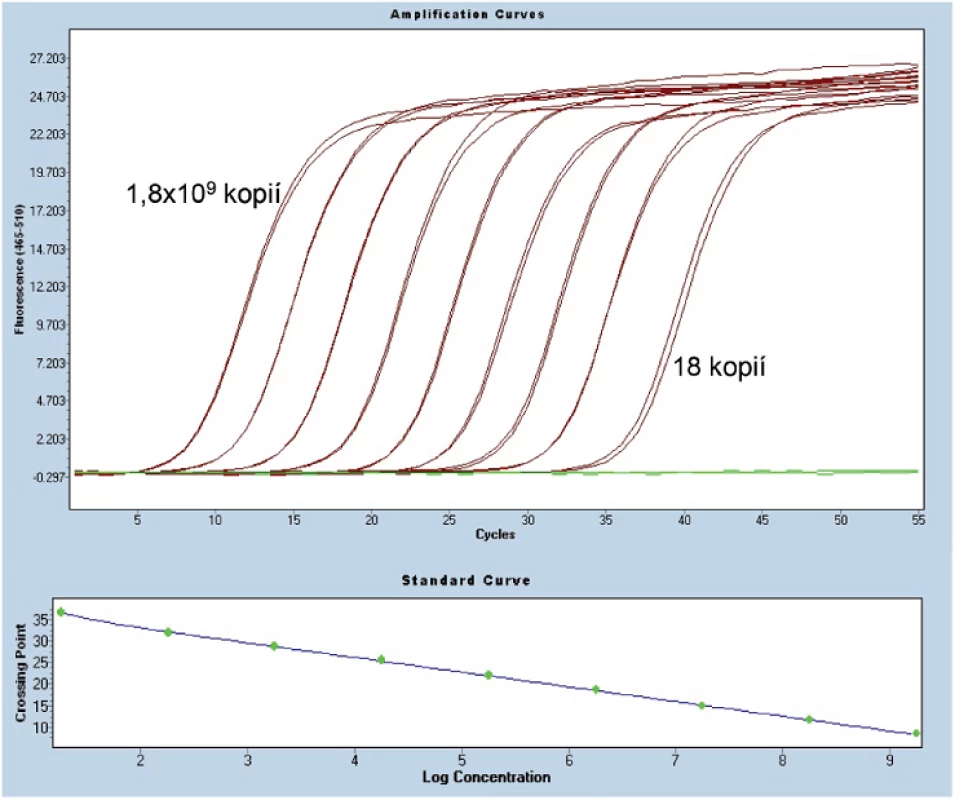
Výsledek sledování MRN pomocí RQ-PCR Ig/TCR je následně předáván klinickému lékaři. Je však nutné, aby byl klinický spolupracovník dobře poučen o správné interpretaci a znal použité termíny, které se objevují na písemně vydávaných protokolech. Vždy je nutné na výstupech uvést rozmezí kvantifikace a senzitivitu, aby bylo možné se vyhnout např. okolnosti, že pokles MRN z kvantifikovatelných hodnot do oblasti pouhé pozitivity mimo rozmezí kvantifikace nemusí znamenat změnu v dynamice MRN, ale pouze horší technickou kvalitu daného RQ-PCR.
Stejný metodický postup platí i v případě RQ-PCR u pacientů s MCL, kdy u více než 90 % dochází k translokaci t(11;14). Touto translokací se gen CCND1, který kóduje protein cyklin D1, dostává pod regulační oblast genu pro IgH, respektive je translokován k J segmentu genu IgH. Překážkou molekulárního stanovení translokace je však charakter zlomů na chromozomu 11, které se mohou vyskytovat v oblasti o velikosti 120 kbp. Genomická PCR dokáže analyzovat pouze zlomy spadající do oblasti tzv. hlavního translokačního místa (major translocation cluster - MTC), což tvoří asi 30 - 50 % případů (47). V případě PCR detekce dané translokace je translokace sekvenována a následně je určena MTC oblast, místo zlomu, přechodová oblast a J segment, kdy se opět přes přechodovou oblast navrhují minimálně dva specifické primery. Další postup, pravidla a kritéria jsou shodná jako v případě detekce MRN pomocí RQ-PCR Ig/TCR.
U pacientů s FL se jako marker MRN používá RQ-PCR detekce translokace t(14;18), kterou nacházíme až u 90 % pacientů. I zde může docházet ke zlomům ve více místech translokujícího se genu Bcl-2, proto jsou v rámci EuroMRD navrhovány a optimalizovány primery pro jednotlivá místa zlomu. V současné době je dostupný komerční kit k RQ-PCR detekci translokace: „t(14;18) quantification kit“, který detekuje pacienty s nejčastějším zlomovým místem MBR – „major breakpoint region“ (ROCHE Diagnostics).
U T-ALL je také možno sledovat MRN pomocí translokace SIL1/TAL1 na úrovni DNA (48).
RT-PCR TECHNIKY VYUŽÍVAJÍCÍ NÁDOROVĚ SPECIFICKOU RNA
Tato metodika využívá mRNA z daného materiálu, je ovlivněna viabilitou analyzovaných buněk a nemonitoruje přímo množství nádorových buněk, ale hladinu exprese genu. Z toho plyne i její omezení a nároky na vstupní materiál. Izolace buněk z kostní dřeně a/nebo periferní krve je nutná do 24 hod od odběru a v případě screeningu dané exprese ve vzorku tkáně je nutný nativní, zmražený materiál, případně materiál fixovaný v 40% alkoholu. Izolace RNA ze vzorku trepanobiopsie je opět limitovaná a získat kvalitní a nedegradovanou RNA téměř nelze. Výhodou metod pracujících s RNA je možnost společné analýzy více vzorků od různých pacientů pomocí jediného zavedeného RT-RQ-PCR systému. Výsledky však mohou být více ovlivněny kvalitou vzorku, dobou mezi odběrem a jeho zpracováním, případně probíhající terapií.
Po izolaci RNA provedeme reverzní transkripci, abychom získali komplementární DNA (cDNA), se kterou následně pracujeme neboť RNA je nestabilní a náchylná k degradaci. Nejčastějšími cíli jsou produkty chromozomálních translokací, fúzní geny nebo aberantně exprimované geny.
Pro kvantifikaci dané exprese se používá buď relativní a/nebo absolutní kvantifikace. U relativní kvantifikace se analyzují změny v genové expresi v porovnání s referenčním vzorkem. Nejprve se vztahuje (normalizuje) exprese kvantifikovaného genu k expresi tzv. „housekeeping genu“, což je gen, který by měl být stabilně exprimován ve všech tkáních. Nejčastěji se používají gen ABL (Abelson), gen pro β-2 mikroglobulin a gen pro β-glukuronidázu (49). Tato normalizovaná exprese se označuje jako Δ Ct (Ct vyšetřovaného genu – Ct „housekeeping genu“ ve vzorku). Dále se stanovuje RFI (relative fold increase/decrease), což je hodnota Δ Ct stanovovaného genu ve vzorku k hodnotě Δ Ct daného genu v referenčním vzorku, např. u zdravých dárců, v neléčeném terénu, případně v kalibrátoru, což je cDNA např. z buněčné linie nebo plazmidová DNA. Tato hodnota je označována jako ΔΔ Ct a hodnota RFI je následně vypočtena jako 2-ΔΔ Ct.
V případě absolutní kvantifikace vztahujeme vyšetřovaný vzorek ke známému množství – standardní kalibrační křivce, kterou sestrojíme buď z buněčné linie, která nese danou genetickou změnu, nebo z plazmidové ředící řady. V případě plazmidové ředící řady je nutné genetickou změnu vložit do plazmidu, pomnožit v bakteriích, následně izolovat a určit počet kopií. Daná exprese se vztahuje opět k expresi „housekeeping genu“, kdy i v tomto případě je nutno sestrojit kalibrační křivku k přesnému určení množství molekul „housekeeping genu“. Tato normalizace slouží k určení koncentrace vstupní cDNA ve vzorku. Zde je normalizovaná hodnota (např. počet kopií) určena jako poměr hodnot vyšetřovaného genu a „housekeeping genu“. I zde musí standardní kalibrační křivky splňovat kritéria a parametry pro reprodukovatelnost a efektivitu amplifikace (ideální sklon - „slope“ 3,33; korelační koeficient 1). Citlivost metodiky RT-RQ-PCR se pohybuje podle daného systému od 10-5-10-6 v případě vztahování k počtu buněk a 10-5 kopií genu v případě využití plazmidové ředící řady.
Pokud sledujeme atypickou expresi genů (např. WT1, cyklin D1) pomocí kvantitativní detekce je nutné pro každý systém nastavit tzv. „cut-off“ limit, který jasně odliší hladinu fyziologické exprese (detekované ve vzorcích zdravých dárců) od atypické exprese.
U pacientů s karcinomy se hledají specificky exprimované molekuly nádorových buněk, např. karcinoembryonální antigen (CEA), různé typy cytokeratinů (CK19, CK20), muciny apod. (6, 7,25). U některých typů nádorů (např. melanom) se doporučuje sledovat expresi více markerů z důvodu falešně negativity dané heterogenitou v expresi např. melanomových antigenů (26).
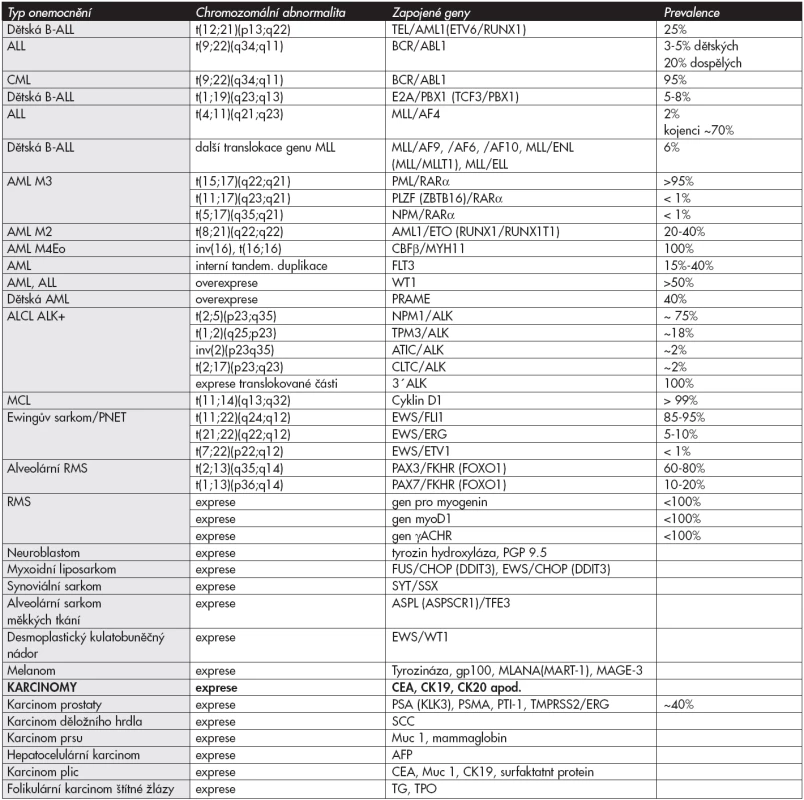
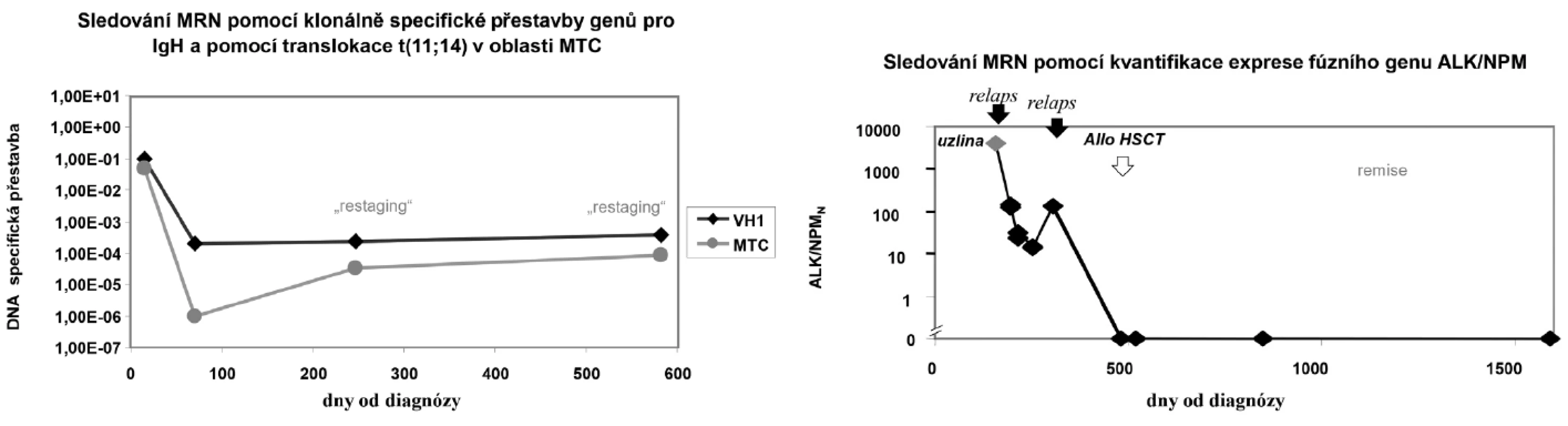
Přehled nejčastějších fúzních a atypicky exprimovaných genů u hematologických i nehematologických onemocnění je uveden v tabulce č.1. Příklady sledování MRN jsou uvedeny na obrázku č.3.
ZÁVĚR
Je zřejmé, že sledování hladiny MRN má obrovský význam pro pacienta a často rozhoduje o jeho dalším osudu. Proto je nutné, aby molekulární metodika detekce MRN byla vždy správně provedená a interpretovaná. Pokud je to možné, je vhodné aplikovat alespoň dvě metodiky sledování a hladiny korelovat. U hematologických malignit je téměř vždy korelováno vyšetření průtokovou cytometrií s molekulární analýzou DNA, případně RNA. Každá z metod má své limity, výhody a nevýhody, náročnost na vstupní materiál a rychlost provedení. Diagnostický vzorek je v první řadě nutno řádně molekulárně typizovat a najít vhodný molekulární marker/markery onemocnění pro následné sledování MRN. Je nutné pamatovat vždy v čase diagnózy na vhodný odběr primárního vzorku, aby pacient nebyl následně vyřazen z možnosti sledovat MRN a to pouze z důvodu chyby při odběru a zpracování materiálu.
PODĚKOVÁNÍ
Podpořeno projektem (Ministerstva zdravotnictví) koncepčního rozvoje výzkumné organizace 00064203 (FN MOTOL) a OPPK CZ.2.16/3.1.00/24022.
SEZNAM ZKRATEK A SLOVNÍČEK
- 3´ ALK – 3´ konec genu “anaplastic lymphoma kinase”
- ABL – gen “v-abl Abelson murine leukemia viral oncogene homolog 1”
- AFP – gen “alpha-fetoprotein”
- ALL – akutní lymfoblastická leukémie
- AML – akutní myeloidní leukémie
- AML1/ETO – fúze genů “acute myoloid leukemia
- (RUNX1/RUNX1T1) – 1/core-binding factor”, nyní “runt-related transcription factor 1/runt-related transcription factor 1; translocated to 1”
- ASPL (ASPSCR1)/TFE3 – fúze genů “alveolar soft part sarcoma chromosome region, candidate 1/transcription factor binding to IGHM enhancer 3”
- ATIC/ALK – fúze genů “5-aminoimidazole-4-carboxamide ribonucleotide formyltransferase/IMP cyclohydrolase/anaplastic lymphoma kinase”
- B-ALL – akutní lymfoblastická leukémie z B buněk
- Bcl2 – gen “B-cell CLL/lymphoma 2”
- BCR/ABL – fúze genů “breakpoint cluster region/c-abl oncogene 1, dříve “v-abl Abelson murine leukemia viral oncogene homolog 1”
- CBFß/MYH11 – fúze genů “core-binding factor/myosin, heavy chain 11
- cDNA – komplementární DNA
- CEA – karcinoembryonální antigen
- CK 19, 20 – cytokeratin 19, 20
- CLTC/ALK – fúze genů “clathrin, heavy chain/anaplastic lymphoma kinase”
- CML – chronická myeloidní leukémie
- Ct, Cp – “crossing point”
- DLBCL – difúzní velkobuněčný B lymfom
- DMSO – dimethylsulfoxid
- DNA – deoxyribonukleová kyselina
- E2A (TCF3)/PBX1 – fúze genů “transcription factor 3 (E2A immunoglobulin enhancer binding factors E12/E47) /pre-B-cell leukemia transcription factor 1
- EDTA – kyselina ethylendiamintetraoctová
- ESG-MRD-ALL – European Study Group on Minimal Residual Disease in Acute Lymphoblastic Leukema
- EWS/ERG – fúze genů “Ewing sarcoma/v-ets erythroblastosis virus E26 oncogene homolog”
- EWS/ETV1 – fúze genů “Ewing sarcoma/ets variant gene 1”
- EWS/FLI1 – fúze genů “Ewing sarcoma/Friend leukemia virus integration 1”
- EWS/CHOP (DDIT3) – fúze genů “Ewing sarcoma/DNA-damage-inducible transcript 3
- EWS/WT1 – fúze genů “Ewing sarcoma/Wolms tumor 1”
- FL – folikulární lymfom
- FLT3 – gen “fms-related tyrosine kinase 3”
- FUS/CHOP (DDIT3) – fúze genů “fused in sarcoma/DNA-damage-inducible transcript 3
- Gp100 – gen pro melanozomální matrix protein
- Ig – imunoglobulíny
- MAGE-3 – gen “melanoma antigen A3 family”
- MBR – “major breakpoint region”
- MCL – lymfom z buněk pláště
- MDN – minimální diseminovaná nemoc
- MLANA (MART-1) – gen “melanoma antigen recognized T cell” nyní “melan A”
- MLL/AF4 – fúze genů “myeloid/lymphoid or mixed-lineage leukemia/ ALL1-fused gene from chromosome 4 (nyní AF4/FMR2 family, member 1)”
- MLL/ELL – fúze genů “myeloid/lymphoid or mixed-lineage leukemia/ elongation factor RNA polymerase II”
- MLL/ENL (MLL/MLLT1) – fúze genů “myeloid/lymphoid or mixed-lineage leukemia/ myeloid/lymphoid or mixed-lineage leukemia translocated to, 1”
- MM – mnohočetný myelom
- MRN – minimální reziduální nemoc
- mRNA – “massanger” RNA
- MTC – “major translocation cluster”
- Muc1 – gen “mucin 1”
- myoD1 – gen “myogenic differentiation 1”
- NHL – non-Hodgkinské lymfomy
- NPM1/ALK – fúze genů “nucleophosmin/anaplastic lymphoma kinase”
- NPM1/RARα – fúze genů “nucleophosmin/retinoic acid receptor, alpha”
- PAX3/FKHR (FOXO1) – fúze genů “paired box 3/forkhead homolog in rhabdomyosarcoma, nyní forkhead box O1”
- PAX7/FKHR (FOXO1) – fúze genů “paired box 7/forkhead homolog in rhabdomyosarcoma, nyní forkhead box O1”
- PCR – polymerázová řetězová reakce
- PGP 9.5 – gen “neuroendocrine protein gene product”
- PLZF (ZBTB16)/RARα – fúze genů “zinc finger protein 145 (Kruppel-like, expressed in promyelocytic leukemia), nyní zinc finger and BTB domain containing 16/retinoic acid receptor, alpha”
- PML/RARα – fúze genů “promyelocytic leukemia/retinoic acid receptor, alpha”
- PNET – primitivní neuroektodermální tumor
- PRAME – gen “preferentially expressed antigen in melanoma”
- PSA (KLK3) – gen “prostate specific antigen”, nyní “kallikrein-related peptidase 3”
- PSMA – gen “prostate-specific membrane antigen”
- PTI-1 – gen “prostate tumour-inducing”
- RFI – “relative fold increase/decrease”
- RMS – rabdomyosarkom
- RNA – ribonukleová kyselina
- RQ-PCR – kvantitativní PCR v reálném čase
- RQ-RT-PCR – reverzně-transkriptázové kvantitativní PCR v reálném čase
- RT-PCR – reverzně-transkriptázová PCR
- SCC – gen “squamous cell carcinoma”
- SIL1/TAL1 – fúze genů “SIL1 homolog, endoplasmic reticulum chaperone/T-cell acute lymphocytic leukemia 1”
- SYT/SSX – fúze genů “synaptotagmin/synovial sarcoma”
- T-ALL – akutní lymfoblastická leukémie z T buněk
- TCR – T buněčné receptory
- TEL/AML1 – fúze genů “TEL oncogene/acute myoloid leukemia 1, nyní ets
(ETV6/RUNX1) – variant 6/runt-related transcription factor 1” - TG – gen “thyroglobulin”
- TMPRSS2/ERG – fúze genů “transmembrane protease, serine 2/v-ets erythroblastosis virus E26 oncogene homolog”
- TPM3/ALK – fúze genů “tropomyosin 3/anaplastic lymphoma kinase”
- TPO – gen “thyroid peroxidase”
- V-(D)-J – variable-diversity-joining
- WT1 – gen “Wilms tumor 1”
- γACHR – gen pro gamma podjednotku acetylcholinového receptoru
Adresa pro korespondenci:
RNDr. Markéta Kalinová, Ph.D.
Ústav patologie a molekulární medicíny 2.LF UK a FN Motol
V Úvalu 84, 150 06 Praha 5
tel.: 224435634
e-mail: marketa.kalinova@lfmotol.cuni.cz
Sources
1. Gutterman JU, Mavligit G, Burgess MA, et al. Immunodiagnosis of acute leukemia: detection of residual disease. J Natl Cancer Inst 1974; 53(2): 389-392.
2. Campana D, Pui CH. Detection of minimal residual disease in acute leukemia: methodologic advances and clinical significance. Blood 1995; 85(6): 1416-1434.
3. Krsková L, Mrhalová M, Hilská I, et al. Detection and clinical significance of bone marrow involvement in patients with rhabdomyosarcoma. Virchows Arch 2010; 456(5): 463-472.
4. Wagner LM, Smolarek TA, Sumegi J, Marmer D. Assessment of minimal residual disease in ewing sarcoma. Sarcoma 2012; 2012 : 780129.
5. Stark B, Avigad S, Luria D, et al. Bone marrow minimal disseminated disease (MDD) and minimal residual disease (MRD) in childhood T-cell lymphoblastic lymphoma stage III, detected by flow cytometry (FC) and real-time quantitative polymerase chain reaction (RQ-PCR). Pediatr Blood Cancer 2009; 52(1): 20-25.
6. Ghossein RA, Bhattacharya S. Molecular detection and characterisation of circulating tumour cells and micrometastases in solid tumours. Eur J Cancer 2000; 36(13 Spec No): 1681-1694.
7. Sun YF, Yang XR, Zhou J, et al. Circulating tumor cells: advances in detection methods, biological issues, and clinical relevance. J Cancer Res Clin Oncol 2011; 137(8): 1151-1173.
8. Cabinaková M, Tesařová P. Disseminated and circulating tumour cells and their role in breast cancer. Folia Biol (Praha) 2012; 58(3): 87-97.
9. Heiss MM, Simon EH, Beyer BC, et al. Minimal residual disease in gastric cancer: evidence of an independent prognostic relevance of urokinase receptor expression by disseminated tumor cells in the bone marrow. J Clin Oncol 2002; 20(8): 2005-2016.
10. Biondi A, Yokota S, Hansen-Hagge TE, et al. Minimal residual disease in childhood acute lymphoblastic leukemia: analysis of patients in continuous complete remission or with consecutive relapse. Leukemia 1992; 6(4): 282-288.
11. Brisco MJ, Condon J, Hughes E, et al. Outcome prediction in childhood acute lymphoblastic leukaemia by molecular quantification of residual disease at the end of induction. Lancet 1994; 343 (8891): 196-200.
12. Goulden NJ, Knechtli CJ, Garland RJ, et al. Minimal residual disease analysis for the prediction of relapse in children with standard-risk acute lymphoblastic leukaemia. Br J Haematol 1998; 100(1): 235-244.
13. Krejci O, van der Velden VH, Bader P, et al. Level of minimal residual disease prior to haematopoietic stem cell transplantation predicts prognosis in paediatric patients with acute lymphoblastic leukaemia: a report of the Pre-BMT MRD Study Group. Bone Marrow Transplant 2003; 32(8): 849-851.
14. Neale GA, Menarguez J, Kitchingman GR, et al. Detection of minimal residual disease in T-cell acute lymphoblastic leukemia using polymerase chain reaction predicts impending relapse. Blood 1991; 78(3): 739-747.
15. Nizet Y, Van Daele S, Lewalle P, et al. Long-term follow-up of residual disease in acute lymphoblastic leukemia patients in complete remission using clonogeneic IgH probes and the polymerase chain reaction. Blood 1993; 82(5): 1618-1625.
16. Wasserman R, Galili N, Ito Y, et al. Residual disease at the end of induction therapy as a predictor of relapse during therapy in childhood B-lineage acute lymphoblastic leukemia. J Clin Oncol 1992; 10(12): 1879-1888.
17. Yokota S, Hansen-Hagge TE, Ludwig WD, et al. Use of polymerase chain reactions to monitor minimal residual disease in acute lymphoblastic leukemia patients. Blood 1991; 77(2): 331-339.
18. Froňková E, Mejstříková E, Avigad S, et al. Minimal residual disease (MRD) analysis in the non-MRD-based ALL IC-BFM 2002 protocol for childhood ALL: is it possible to avoid MRD testing? Leukemia 2008; 22(5): 989-997.
19. Pott C, Schrader C, Gesk S, et al. Quantitative assessment of molecular remission after high-dose therapy with autologous stem cell transplantation predicts long-term remission in mantle cell lymphoma. Blood 2006; 107(6): 2271-2278.
20. Trka J, Kalinová M, Hrušák O, et al. Real-time quantitative PCR detection of WT1 gene expression in children with AML: prognostic significance, correlation with disease status and residual disease detection by flow cytometry. Leukemia 2002; 16(7): 1381-1389.
21. Zaliova M, Froňková E, Krejčíková K, et al. Quantification of fusion transcript reveals a subgroup with distinct biological properties and predicts relapse in BCR/ABL-positive ALL: implications for residual disease monitoring. Leukemia 2009; 23(5): 944-951.
22. Szczepanski T, Orfao A, van der Velden VH, San Miguel JF, van Dongen JJ. Minimal residual disease in leukaemia patients. Lancet Oncol 2001; 2(7): 409-417.
23. Gertler R, Rosenberg R, Fuehrer K, et al. Detection of circulating tumor cells in blood using an optimized density gradient centrifugation. Recent Results Cancer Res 2003; 162 : 149-155.
24. Sleijfer S, Gratama JW, Sieuwerts AM, et al. Circulating tumour cell detection on its way to routine diagnostic implementation? Eur J Cancer 2007; 43(18): 2645-2650.
25. Berois N, Varangot M, Aizen B, et al. Molecular detection of cancer cells in bone marrow and peripheral blood of patients with operable breast cancer. Comparison of CK19, MUC1 and CEA using RT-PCR. Eur J Cancer 2000; 36(6): 717-723.
26. Gkalpakiotis S, Arenberger P, Kremen J, Arenbergerova M. Quantitative detection of melanoma-associated antigens by multimarker real-time RT-PCR for molecular staging: results of a 5 years study. Exp Dermatol 2010; 19(11): 994-999.
27. Hoshino M, Ogose A, Kawashima H, et al. Molecular analyses of cell origin and detection of circulating tumor cells in the peripheral blood in alveolar soft part sarcoma. Cancer Genet Cytogenet 2009; 190(2): 75-80.
28. Beiske K, Ambros PF, Burchill SA, Cheung IY, Swerts K. Detecting minimal residual disease in neuroblastoma patients-the present state of the art. Cancer Lett 2005; 228(1-2): 229-240.
29. van der Velden VH, Jacobs DC, Wijkhuijs AJ, et al. Minimal residual disease levels in bone marrow and peripheral blood are comparable in children with T cell acute lymphoblastic leukemia (ALL), but not in precursor-B-ALL. Leukemia 2002; 16(8): 1432-1436.
30. Kalinová M, Krsková L, Břízová H, et al. Quantitative PCR detection of NPM/ALK fusion gene and CD30 gene expression in patients with anaplastic large cell lymphoma—residual disease monitoring and a correlation with the disease status. Leuk Res 2008; 32(1): 25-32.
31. Grimwade D. The significance of minimal residual disease in patients with t(15;17). Best Pract Res Clin Haematol 2002; 15(1): 137-158.
32. van der Velden VH, Hochhaus A, Cazzaniga G, et al. Detection of minimal residual disease in hematologic malignancies by real-time quantitative PCR: principles, approaches, and laboratory aspects. Leukemia 2003; 17(6): 1013-1034.
33. Hrušák O, Porwit-MacDonald A. Antigen expression patterns reflecting genotype of acute leukemias. Leukemia 2002; 16(7): 1233-1258.
34. Lucio P, Parreira A, van den Beemd MW, et al. Flow cytometric analysis of normal B cell differentiation: a frame of reference for the detection of minimal residual disease in precursor-B-ALL. Leukemia 1999; 13(3): 419-427.
35. Campana D, Coustan-Smith E. Detection of minimal residual disease in acute leukemia by flow cytometry. Cytometry 1999; 38(4): 139-152.
36. van Wering ER, Beishuizen A, Roeffen ET, et al. Immunophenotypic changes between diagnosis and relapse in childhood acute lymphoblastic leukemia. Leukemia 1995; 9(9): 1523-1533.
37. Mejstříková E, Froňková E, Kalina T, et al. Detection of residual B precursor lymphoblastic leukemia by uniform gating flow cytometry. Pediatr Blood Cancer 2010; 54(1): 62-70.
38. van Dongen JJ, Langerak AW, Bruggemann M, et al. Design and standardization of PCR primers and protocols for detection of clonal immunoglobulin and T-cell receptor gene recombinations in suspect lymphoproliferations: report of the BIOMED-2 Concerted Action BMH4-CT98-3936. Leukemia 2003; 17(12): 2257-2317.
39. van Dongen JJ, Macintyre EA, Gabert JA, et al. Standardized RT-PCR analysis of fusion gene transcripts from chromosome aberrations in acute leukemia for detection of minimal residual disease. Report of the BIOMED-1 Concerted Action: investigation of minimal residual disease in acute leukemia. Leukemia 1999; 13(12): 1901-1928.
40. Langerak AW, Wolvers-Tettero IL, van Gastel-Mol EJ, Oud ME, van Dongen JJ. Basic helix-loop-helix proteins E2A and HEB induce immature T-cell receptor rearrangements in nonlymphoid cells. Blood 2001; 98(8): 2456-2465.
41. van der Velden VH, Bruggemann M, Hoogeveen PG, et al. TCRB gene rearrangements in childhood and adult precursor-B-ALL: frequency, applicability as MRD-PCR target, and stability between diagnosis and relapse. Leukemia 2004; 18(12): 1971-1980.
42. van der Velden VH, Wijkhuijs JM, Jacobs DC, van Wering ER, van Dongen JJ. T cell receptor gamma gene rearrangements as targets for detection of minimal residual disease in acute lymphoblastic leukemia by real-time quantitative PCR analysis. Leukemia 2002; 16(7): 1372-1380.
43. van der Velden VH, Willemse MJ, van der Schoot CE, et al. Immunoglobulin kappa deleting element rearrangements in precursor-B acute lymphoblastic leukemia are stable targets for detection of minimal residual disease by real-time quantitative PCR. Leukemia 2002; 16(5): 928-936.
44. Verhagen OJ, Willemse MJ, Breunis WB, et al. Application of germline IGH probes in real-time quantitative PCR for the detection of minimal residual disease in acute lymphoblastic leukemia. Leukemia 2000; 14(8): 1426-1435.
45. Szczepanski T, Beishuizen A, Pongers-Willemse MJ, et al. Cross-lineage T cell receptor gene rearrangements occur in more than ninety percent of childhood precursor-B acute lymphoblastic leukemias: alternative PCR targets for detection of minimal residual disease. Leukemia 1999; 13(2): 196-205.
46. Pongers-Willemse MJ, Verhagen OJ, Tibbe GJ, et al. Real-time quantitative PCR for the detection of minimal residual disease in acute lymphoblastic leukemia using junctional region specific TaqMan probes. Leukemia 1998; 12(12): 2006-2014.
47. Tsujimoto Y, Yunis J, Onorato-Showe L, et al. Molecular cloning of the chromosomal breakpoint of B-cell lymphomas and leukemias with the t(11;14) chromosome translocation. Science 1984; 224(4656): 1403-1406.
48. Pongers-Willemse MJ, Seriu T, Stolz F, et al. Primers and protocols for standardized detection of minimal residual disease in acute lymphoblastic leukemia using immunoglobulin and T cell receptor gene rearrangements and TAL1 deletions as PCR targets: report of the BIOMED-1 CONCERTED ACTION: investigation of minimal residual disease in acute leukemia. Leukemia 1999; 13(1): 110-118.
49. Beillard E, Pallisgaard N, van der Velden VH, et al. Evaluation of candidate control genes for diagnosis and residual disease detection in leukemic patients using ‘real-time’ quantitative reverse-transcriptase polymerase chain reaction (RQ-PCR) - a Europe against cancer program. Leukemia 2003; 17(12): 2474-2486.
50. Froňková E, Trka J. Detekce minimální reziduální nemoci u akutních lymfoblastických leukémií pomocí kvantifikace přestaveb genů pro imunoglobuliny a T-buněčné receptory: jak se vyhnout špatné interpretaci výsledků. Transfuze a hematologie dnes 2005; 11(3): 110-115.
51. van der Velden VH, Cazzaniga G, Schrauder A, et al. Analysis of minimal residual disease by Ig/TCR gene rearrangements: guidelines for interpretation of real-time quantitative PCR data. Leukemia 2007; 21(4): 604-611.
Labels
Anatomical pathology Forensic medical examiner ToxicologyArticle was published in
Czecho-Slovak Pathology

2013 Issue 4
-
All articles in this issue
- Fluorescenční in situ hybridizace na histologických řezech
- Laserová mikrodisekce a její praktické aplikace
- Imunofenotypizace průtokovou cytometrií v patologii
- Minimální reziduální nemoc – možnosti detekce u hematologických i nehematologických malignit
- Jak v praxi zlepšit bioptickou diagnostiku benigních lézí jater (adenom versus fokální nodulární hyperplazie)
- Mucinózny adenokarcinóm (neintestinálneho typu) vzniknutý v zrelom cystickom teratóme vaječníka - kazuistika
- Eozinofilná dysplázia krčka maternice asociovaná s infekciou HPV 6 – kazuistika a prehľad literatúry
- Czecho-Slovak Pathology
- Journal archive
- Current issue
- About the journal
Most read in this issue
- Jak v praxi zlepšit bioptickou diagnostiku benigních lézí jater (adenom versus fokální nodulární hyperplazie)
- Imunofenotypizace průtokovou cytometrií v patologii
- Minimální reziduální nemoc – možnosti detekce u hematologických i nehematologických malignit
- Fluorescenční in situ hybridizace na histologických řezech
