-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Molekulární epidemiologie tuberkulózy v hlavním městě Praha v letech 2013 a 2014
Molecular epidemiology of tuberculosis in the capital of Prague in 2013 and 2014
Aim:
Phylogenetic analysis of Mycobacterium tuberculosis strains by the MIRU-VNTR method and their assignment to lineages and sublineages.Material and methods:
DNA of 132 strains of M. tuberculosis was recovered from patients in the capital of Prague in 2013 and 2014. The MIRU-VNTR method was used. Using the MIRU-VNTRplus website tools, the strains were identified and assigned to genetically related groups.Results:
The highest prevalence of TB was reported in males aged between 45 and 54 years. The isolates of M. tuberculosis show high polymorphism in the number of repetitive sequences. Three global lineages were identified: 1 - Indo-Oceanic, 2 - East-Asian, and 4 - Euro-American, represented by 85.6 % of strains, comprising nine sublineages: Haarlem (40.9 %), H37Rv (25.8 %), S, Cameroon, LAM, X, NEW-1, URAL, and Delhi/CAS. Fully identical repetitive sequences were found in 28.0 % of strains (nine groups) of global lineage 4 - Euro-American, sublineages Haarlem (four groups), H37Rv (three groups), Cameroon (one group), and S (one group). Among the strains analyzed, four multi-drug resistant (MDR) strains were identified without clustering (one in 2013 and three in 2014). Two MDR strains were assigned to Euro-American lineage 4 and two MDR strains to East-Asian lineage 2.Conclusions:
Genotyping of M. tuberculosis strains by the MIRU-VNTR method allows for rapid genetic identification and assignment to lineages and sublineages. The identification of strains of M. tuberculosis, their assignment to lineages, and lineage distribution make it possible to monitor their movement not only within a country but also worldwide. Genotyping data obtained over years make it possible to track the infection source and transmission pathways.Keywords:
lineages – repetitive sequences – phylogenetic analysis
Autoři: E. Tietzeová; M. Pinková; I. Zemanová
Působiště autorů: Národní referenční laboratoř pro mykobakterie, Státní zdravotní ústav Praha
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 67, 2018, č. 2, s. 58-63
Kategorie: Původní práce
Souhrn
Cíl práce:
Fylogenetická analýza kmenů Mycobacterium tuberculosis metodou MIRU-VNTR a jejich zařazení do jednotlivých linií a sublinií.Materiál a metodiky:
DNA 132 kmenů M. tuberculosis získaných od pacientů z hlavního města Praha v roce 2013 a 2014. Byla použita metoda MIRU-VNTR. Kmeny byly webovým softwarem MIRU-VNTRplus identifikovány a zařazeny do geneticky příbuzných skupin.Výsledky:
Nejčastější výskyt onemocnění tuberkulózou v populaci byl prokázán u mužů mezi 45. až 54. rokem. Získané kmeny M. tuberculosis se vyznačují vysokou polymorfií v počtu repetitivních sekvencí. Identifikovány byly tři globální linie: 1 - Indo-Oceánská, 2 - Východo-Asijská a 4 - Euro-Americká, zastoupená 85,6 % kmenů, ke které bylo přiřazeno 9 sublinií: Haarlem (40,9 %), H37Rv (25,8 %), S, Cameroon, LAM, X, NEW-1, URAL a Delhi/CAS. Plná identita repetitivních sekvencí byla prokázána u 28,0 % kmenů (9 skupin) globální linie 4 - Euro-Americké, sublinií Haarlem (4 skupiny), H37Rv (3 skupiny), Cameroon (1 skupina) a S (1 skupina). Mezi analyzovanými kmeny byly čtyři multirezistentní (MDR) kmeny bez klastrování (jeden kmen v roce 2013, tři kmeny v roce 2014). Dva MDR kmeny patřily ke globální linii 4 - Euro-Americké; dva MDR kmeny byly zařazeny ke globální linii 2 - Východo-Asijské.Závěry:
Genotypizace kmenů M. tuberculosis metodou MIRU--VNTR umožňuje rychlou genetickou identifikaci jednotlivých kmenů a jejich zařazení do linií a sublinií. Identifikace kmenů M. tuberculosis, jejich zařazení do linií a rozložení linií dovoluje sledovat jejich pohyb nejen na území jednoho státu, ale i na celém světě. Při víceletém genotypování kmenů lze vysledovat původní zdroj infekce a její přenos.Klíčová slova:
linie Mycobacterium tuberculosis – repetitivní sekvence – fylogenetická analýzaTuberkulóza (TBC) je onemocnění způsobené patogenním agens Mycobacterium tuberculosis komplex (MTBC). Celosvětově se řadí mezi nejčastější infekční onemocnění a příčiny úmrtí. Z hlediska biologické bezpečnosti patří tuberkulóza společně s malárií a AIDS ke třem nejsledovanějším infekčním onemocněním Světovou zdravotnickou organizací (WHO). Celkový počet hlášených případů tuberkulózy v Evropě se značně liší. Nejvyšší výskyt je hlášen z východní Evropy a pobaltských států. V České republice je dlouhodobě zaznamenáván nízký výskyt TBC. V roce 2013 byla incidence onemocnění TBC 4,6/100 000 obyvatel a v roce 2014 4,8/100 000 obyvatel. V Praze byla incidence onemocnění TBC 8,1/100 000 obyvatel v roce 2013 a 7,8/100 000 obyvatel v roce 2014 [19, 20]. Vývoj molekulární biologie posledních let má významný vliv na možnosti diagnostiky tuberkulózy. Identifikací konkrétních alel a jejich řazením do geneticky příbuzných skupin lze prokázat podezření na mikroepidemii [1]. Analýza variabilního počtu opakujících se sekvencí mykobakteriální DNA, MIRU-VNTR (Mycobacterial Interspersed Repetitive Unit--Variable Number of Tandem Repeats), je cenným ukazatelem pro genotypizaci [2]. Velké populační studie prokázaly, že metoda MIRU-VNTR má srovnatelnou diskriminační sílu s metodou RFLP (Restriction fragment length polymorphism) [2, 3]. Optimalizovaný soubor 24 MIRU-VNTR lokusů se stal standardem pro genotypizaci kmenů Mycobacterium tuberculosis po celém světě [2, 3]. Přináší reprodukovatelné výsledky, vyjádřené jako 24místný číselný profil, který umožňuje snadnou výměnu dat na vnitrostátní i mezinárodní úrovni [2, 4]. Na evropské úrovni byla MIRU-VNTR přijata jako nástroj ke sledování přeshraničního přenosu rezistentních kmenů M. tuberculosis.
Cílem předložené práce je fylogenetická analýza kmenů M. tuberculosis metodou MIRU-VNTR a jejich zařazení do jednotlivých linií a sublinií.
MATERIÁL A METODIKY
DNA referenčního kmene M. tuberculosis H37Rv a DNA 132 kmenů M. tuberculosis, získaných od pacientů s trvalým bydlištěm v hlavním městě Praha v letech 2013 a 2014 zaslaných do Národní referenční laboratoře pro mykobakterie laboratořemi ČR k testování citlivosti na antituberkulotika (58 kmenů v roce 2013 a 74 kmenů v roce 2014). Mezi těmito kmeny byly čtyři kmeny multirezistentní (MDR); jeden kmen v roce 2013 a tři kmeny v roce 2014.
Věkové složení souboru pacientů bylo u mužů 22–94 roků (průměr 51,4 let) a u žen 20–87 let (průměr 46,5 roků). Věkové rozložení našeho vzorku populace uvádí graf 1.
Graf 1. Procentuální zastoupení pacientů s TBC podle věku a pohlaví
Chart 1. Age and gender distribution of TB patients
Ze zaslaných kmenů bylo izolačním kitem GenoLyse (HAIN) zhotoveno 132 izolátů DNA. Následně byla provedena genetická analýza kmenů metodou MIRU-VNTR. Panel MIRU-VNTR se skládá z následujících 24 lokusů: 154-424--577-580-802-960-1644-1955-2059-2163b-2165-2347-2401-2461--2531-2687-2996-3007-3171-3192-3690-4052-4156-4348 [5, 6, 7].
Z každého izolátu M. tuberculosis a kontrolního kmene H37Rv bylo metodou rychlého zmnožení (amplifikace) vybraného úseku DNA polymerázovou řetězovou reakcí (PCR) naamplifikováno 24 PCR produktů (tj. 24 sledovaných lokusů). Amplifikované úseky DNA byly ohraničeny 24 páry tzv. primerů, pro každý sledovaný lokus 1 pár (Generi Biotech) [5]. Syntéza vybraného úseku DNA probíhala od přisedlých primerů termostabilními polymerázami HotStar Taq DNA Polymerase kit (QIAGEN) a KAPA2G Robust (Elisabeth Pharmacon). PCR produkty byly nanášeny na 2% agarózový gel (agaróza SIGMA) a vizualizovány interkalačním barvivem SERVA DNA Stain G. K porovnání velikostí fragmentů DNA generovaných v PCR byl použit 50 bp DNA Ladder a 100 bp DNA Ladder (NEW ENGLAND BioLabs).
Fotodokumentace gelů byla pořizována fotoaparátem Canon PowerShot_SX510_HS. Na fotodokumentaci byl podle velikosti jednotlivých PCR produktů kmenů M. tuberculosis a kontrolního kmene H37Rv vizuálně hodnocen počet repetitivních sekvencí podle odečítací tabulky [6]. Každý kmen získal 24místný číselný profil, kde každé číslo znamená počet repetitivních sekvencí v jednotlivých lokusech (například počet repetitivních sekvencí a profil referenčního kmene H37Rv je: 2-2-4-3´-1-3-2-2-2-5-3-4-2-3-6-1-3-3-3-3-5-5-2-2) [7, 8]. Následně byly tyto číselné profily mezi sebou porovnávány a vyhodnocovány. Mykobakteriální kmeny získané od pacientů z pražské aglomerace v letech 2013 a 2014 byly označeny pětimístným kódem (první dvě čísla značí rok, kdy byl materiál odebrán, číslo izolátu představují tři následující čísla). Pod těmito kódy byly kmeny řazeny do fylogeneticky příbuzných skupin. Shluková analýza a identifikace linií a sublinií byla provedena online přístupným programem MIRU-VNTRplus (http://www.miru-vntrplus.org) [7, 8]. Klastrované izoláty byly definovány jako izoláty se shodným počtem repetitivních sekvencí u všech 24 lokusů ve výše uvedeném pořadí.
VÝSLEDKY
Nejčastější výskyt onemocnění tuberkulózou byl prokázán u mužů mezi 45. až 64. rokem; u žen mezi 25. až 44. rokem (viz graf 1).
Kmeny M. tuberculosis získané od 132 pacientů bydlících v Praze v letech 2013–2014 byly zařazeny do geneticky příbuzných skupin. Fylogenetické řazení a identifikace kmenů od pražských pacientů znázorňuje grafický výstup shlukové analýzy na obrázku 1. Mezi identifikovanými kmeny je nejvíce kmenů (113) přiřazeno ke globální linii 4 – Euro-Americké dále se dělící do sublinií: Haarlem 40,9 %; H37Rv 25,8 %; Latinská Amerika-Středomoří (LAM) 5,3 %; Cameroon 3,8 %; S 3,8 %; X 2,3 %; NEW-1 1,5 %;URAL 1,5 % a Delhi/CAS 0,8 %. Z ostatních globálních linií byla v našem souboru identifikována linie 1 – Indo--Oceánská: EAI 7 kmenů a 2 – Východo-Asijská: Beijing 12 kmenů (graf 2).
Obr. 1. Dendrogram kmenů M. tuberculosis v Praze 2013–2014
Fig. 1. Dendrogram of M. tuberculosis strains from Prague, 2013–2014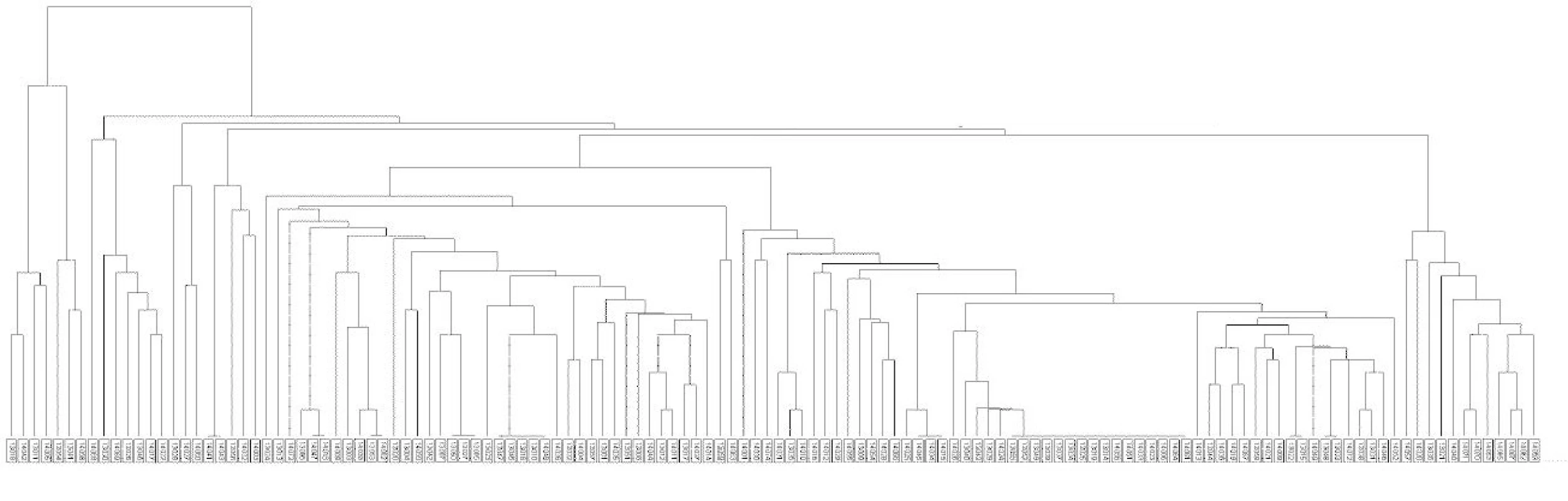
Graf 2. Zastoupení sublinií kmenů M. tuberculosis 2013–2014.
Chart 2. Sublineage distribution of M. tuberculosis strains, 2013–2014
Rozložení opakování repetitivních sekvencí genomu M. tuberculosis u 24 lokusů s vyhodnocením diverzity alel uvádí tabulka 1. Nejvyšší diverzita alel u pražských kmenů byla zjištěna u lokusu 2163b. Kmeny vykazovaly vysokou polymorfii (neklastrovaných kmenů bylo 72 %). Plná identita repetitivních sekvencí 24 lokusů byla u 9 skupin klastrovaných kmenů globální linie 4 - Euro-Americké (28,0 %) zastoupené subliniemi: Haarlem (17,4 %), H37Rv (7,6 %), Cameroon (1,5 %) a S (1,5 %). Počet klastrů a jejich procentuální zastoupení v populaci v průběhu let 2013–2014 uvádí tabulka 2. Čtyři multirezistentní kmeny byly bez klastrování: kmen z roku 2013 byl zařazen do linie 4 – Euro-Americké, jeden kmen z roku v 2014 byl zařazen do linie 4 – Euro-Americké a dva kmeny do linie 2 – Východo-Asijské.
Tab. 1. Tabulka 1. Počet repetitivních sekvencí a diverzita u 24 lokusů genomu M. tuberculosis
Table 1. The number of repetitive sequences and diversity in 24 M. tuberculosis genomic loc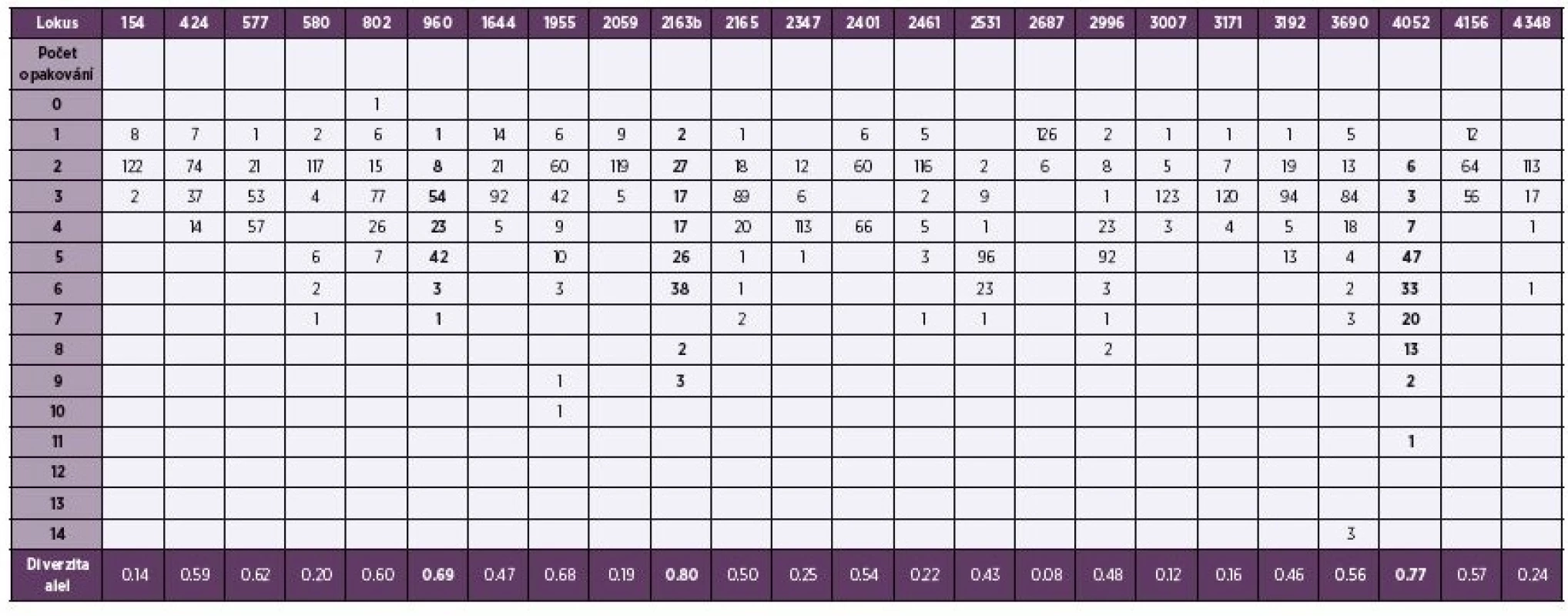
Tab. 2. Tabulka 2. Klastrované kmeny M. tuberculosis linie 4 (Euro-Americké) v Praze 2013–2014
Table 2. Clustered strains of M. tuberculosis of Euro-American lineage 4 from Prague, 2013–2014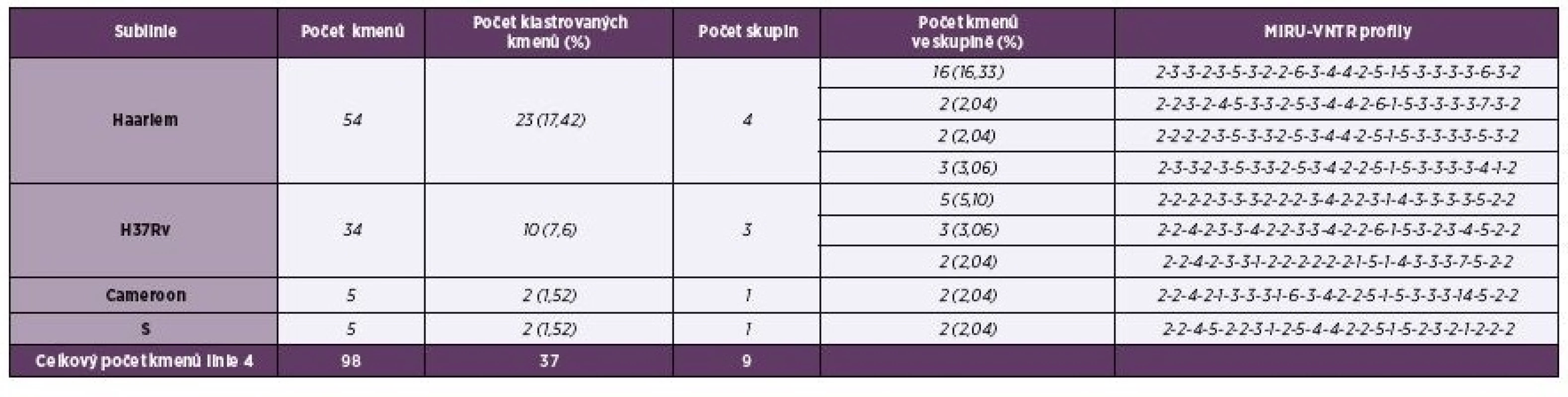
DISKUSE
V Praze je dlouhodobě nejvyšší počet nemocných TBC v celé ČR. Byla proto vybrána jako vhodný reprezentativní vzorek ke genetické analýze kmenů M. tuberculosis metodou MIRU-VNTR. Příčinou zvýšené incidence TBC ve velkoměstských aglomeracích je celý komplex demografických a sociologických faktorů, které souvisejí s přílivem obyvatelstva do měst. S tím souvisí i plošné přelidnění některých velkoměstských částí [10]. Statistické informace celé ČR [19, 20] uvádějí, že nejvíc nemocných TBC je ve vyšších věkových skupinách. U mužů lze sledovat vyšší výskyt také ve střední věkové skupině, zhruba od 45 let. První studie molekulární epidemiologie tuberkulózy v Praze byla provedena v roce 1995 metodou založenou na polymorfismu délky restrikčních fragmentů – RFLP [9]. V této studii se Kubín [9] zabývá souborem 75 kmenů MTB, z nichž 70,7 % bylo neklastrovaných a 29,3 % kmenů klastrovaných. Náš soubor kmenů představoval 72 % kmenů neklastrovaných a 28 % kmenů klastrovaných. Poměr klastrovaných a neklastrovaných kmenů MTB v našem souboru odpovídá poměru zjištěnému v populačním vzorku pacientů v Praze v roce 1995 metodou RFLP.
Dlouho převládal názor, že kmeny M. tuberculosis jsou stejně virulentní. Plošnou genotypizací lze prokázat, že existuje malé procento kmenů, které způsobují neúměrně velký počet případů onemocnění. To znamená, že některé kmeny se šíří účinněji než jiné [1]. MIRU-VNTR je spolehlivá metoda pro rychlou genotypizaci velkého počtu izolátů a jejich fylogenetickou analýzu.
Webová aplikace MIRU-VNTRplus představuje volně přístupnou službu, která umožňuje uživatelům analyzovat údaje získané vyšetřením jejich kmenů. Referenční databáze kmenů reprezentuje kmeny MTBC. Vlastní data lze analyzovat buď jednou, nebo kombinací několika metod genotypizace [7, 8]. Oelemann et al. [12] a Valcheva et al. [17] ukazují možnost zvýšení diskriminační síly shlukové analýzy kombinací použitých PCR metod genotypizace (RFLP, spoligotyping, MIRU-VNTR 12 lokusů, či 15 lokusů). Diskriminační síla MIRU-VNTR 24 lokusů je z dříve používaných PCR metod nejvyšší [18]. O přenosu bakteriálních kmenů způsobujících TBC v Evropě se lze informovat z jednoletých studií [11, 12]. Většina molekulárních studií genotypizace M. tuberculosis však uvádí víceleté výsledky. Příkladem jsou dvouletá studie z Irska [13], tříletá z Belgie [14], čtyřletá z Itálie [15] a také devítiletá z Polska [16]. Víceleté studie umožňují, s ohledem na biologické vlastnosti organismu, vysledování původního zdroje infekce [16]. Ve výše uvedených studiích převažuje globální linie 4 – Euro-Americká [12, 13, 15, 17] zastoupená především subliniemi Latinská Amerika-Středomoří (LAM), Haarlem a T. Sublinie Haarlem má nejvyšší procento zastoupení kmenů ve studii z Německa [12] a z Polska [16].
ZÁVĚRY
Genotypizace kmenů M. tuberculosis metodou MIRU-VNTR umožňuje rychlou genetickou identifikaci jednotlivých kmenů a jejich zařazení do linií a sublinií. Identifikace kmenů MTB, jejich zařazení do linií a rozložení linií dovoluje sledovat jejich pohyb nejen na území jednoho státu, ale i na celém světě. Identifikací konkrétních alel a jejich řazením do geneticky příbuzných skupin lze prokázat podezření na mikroepidemii.
Kmeny s identickým počtem repetitivních sekvencí v genomu mohou být nadále šetřeny ve spolupráci s epidemiologem a klinickými lékaři. Při víceletém genotypování kmenů lze vysledovat původní zdroj infekce a její přenos.
Podpořeno MZ ČR – RVO („Státní zdravotní ústav – SZU, 75010330“).
Do redakce došlo dne 9. 11. 2017.
Adresa pro korespondenci:
Ing. Evženie Tietzeová, CSc.
Národní referenční laboratoř pro mykobakterie
Státní zdravotní ústav Praha
Šrobárova 48
110 00 Praha 10
e-mail: evzenie.tietzova@szu.cz
Zdroje
1. Barnes PF, Cave MD. Molecular Epidemiology of Tuberculosis. N Engl J Med, 2003; 349(12):1149–1156.
2. Supply P, Allix C, Lesjean S, et al. Proposal for standardization of optimized mycobacterial interspersed repetitive unit-variable-number tandem repeat typing of Mycobacterium tuberculosis. J.Clin Microbiol, 2006;44(12):4498–4510.
3. de Beer J, Kremer K, Ködmön C, et al. First worldwide proficiency study on variable-number tandem-repeat typing of Mycobacterium tuberculosis complex strains. J Clin Microbiol, 2012;50(3):662–669.
4. Kanduma E, McHugh TD, Gillespie SH. Molecular methods for Mycobacterium tuberculosis strain typing: a user’s guide. J Appl Microbiol, 2003;94(5):781–791.
5. Supply P. Multilocus Variable Number Tandem Repeat Genotyping of Mycobacterium tuberculosis. Technical Guide. Institut Pasteur de Lille, 2005. Dostupný na www: https://www.researchgate.net/profile/Philip_Supply/publication/265990159_Multilocus_Variable_Number_Tandem_Repeat_Genotyping_of_Mycobacterium_tuberculosis_Technical_Guide/links/5437e5b40cf2027cbb2055e8/Multilocus-Variable-Number-Tandem-Repeat-Genotyping-of-Mycobacterium-tuberculosis-Technical-Guide.pdf
6. de Beer J, van Soolingen D. Protocol, Quality control VNTR typing. 2011. Dostupný na www: http://www.rivm.nl/dsresource?objectid=db266474-cfb8-4d49-8505-5b808842bfe6.
7. Allix-Béguec C, Harmsen D, Weniger T, et al. Evaluation and strategy for use of MIRU-VNTRplus, a multifunctional database for online analysis of genotyping data and phylogenetic identification of Mycobacterium tuberculosis complex isolates. J Clin Microbiol, 2008;46(8):2692–2699.
8. Weniger T, Krawczyk J, Supply P, et al. MIRU-VNTRplus: a web tool for polyphasic genotyping of Mycobacterium tuberculosis complex bacteria. Nucleic Acids Res, 2010;38 Suppl:W326–331.
9. Kubín M. Uplatnění fingerprintingu mykobakteriální DNA v molekulární epidemiologii tuberkulózy. Epidemiol Mikrobiol Imunol,1997;46(3):99–103.
10. Kubín M, Malý M, Jágrová Z. Podíl bezdomovců na incidenci tuberkulózy v Praze. Stud Pneumol Phtiseol, 2016;76(2):67–72.
11. Bidovec-Stojkovic U, Zolnir-Dovc M, Supply P. One year nationwide evaluation of 24 -locus MIRU-VNTR genotyping on Slovenian Mycobacterium tuberculosis isolates. Respir Med, 2011;105(1):67–73.
12. Oelemann MC, Diel R, Vatin V, Haas W, et al. Assessment of an optimized mycobacterial interspersed repetitive-unit-variable number tandem-repeat typing system combined with spoligotyping for population-based molecular epidemiology studies of tuberculosis. J Clin Microbiol, 2007;45(3):691–697.
13. Fitzgibbon MM, Gibbons N, Roycroft E, et al. A snapshot of genetic lineages of Mycobacterium tuberculosis in Ireland over a two-year period, 2010 and 2011. Euro Surveill, 2013;18(3):pii=20367.
14. Allix-Béguec C, Fauville-Dufaux, Supply P. Three-year population-based evaluation of standardized mycobacterial interspersed repetitive-unit-variable-number tandem-repeat typing of Mycobacterium tuberculosis. J Clin Microbiol, 2008;46(4):1398–1406.
15. Garzelli C, Lari N, Cuccu B, et al. Impact of immigration on tuberculosis in a low-incidence area of Italy: a molecular epidemiological approach. Clin Microbiol Infect, 2010;16(11):1691–1697.
16. Augustynowicz-Kopeć E, Jagielski T, Kozińska M, et al. Transmission of tuberculosis within family-households. J Infect, 2012;64(6):596–608.
17. Valcheva V, Mokrousov I, Narvskaya O, et al. Utility of new 24 - locus variable-number tandem-repeat typing for discriminating Mycobacterium tuberculosis clinical isolates collected in Bulgaria. J Clin Microbiol, 2008;46(9):3005–3011.
18. Ojo OO, Sheehan S, Corcoran DG, et al. Molecular epidemiology of Mycobacterium tuberculosis clinical isolates in Southwest Ireland. Infect Genet Evol, 2010;10(7):1110–1116.
19. Zdravotnická statistika. Tuberkulóza a respirační nemoci, 2013. Ústav zdravotnických informací a statistiky ČR. Praha. Dostupný na www: http://www.uzis.cz/publikace/tuberkuloza-respiracni-nemoci-2013.
20. Zdravotnická statistika. Tuberkulóza a respirační nemoci, 2014. Ústav zdravotnických informací a statistiky ČR. Praha. Dostupný na www: http://www.uzis.cz/publikace/tuberkuloza-respiracni-nemoci-2014.
Štítky
Hygiena a epidemiológia Infekčné lekárstvo Mikrobiológia
Článok vyšiel v časopiseEpidemiologie, mikrobiologie, imunologie
Najčítanejšie tento týždeň
2018 Číslo 2- Parazitičtí červi v terapii Crohnovy choroby a dalších zánětlivých autoimunitních onemocnění
- Očkování proti virové hemoragické horečce Ebola experimentální vakcínou rVSVDG-ZEBOV-GP
- Koronavirus hýbe světem: Víte jak se chránit a jak postupovat v případě podezření?
-
Všetky články tohto čísla
- Stav imunitního systému u pacientů se subklinickou formou urogenitální tuberkulózy
- Molekulární epidemiologie tuberkulózy v hlavním městě Praha v letech 2013 a 2014
- Surveillance invazivního meningokokového onemocnění založená na sekvenaci celého genomu (WGS), Česká republika, 2015
- Boj s rezistencí mikroorganismů na antibiotika
- Streptokokový syndrom toxického šoku – život ohrožující stav vyvolaný různými druhy streptokoků
- Bakteriémie vyvolaná Staphylococcus aureus – význam správného přístupu k diagnostice a léčbě
- Aktualizace českých doporučených postupů pro laboratorní diagnostiku infekcí vyvolaných Clostridium difficile
- Epidemiologie, mikrobiologie, imunologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Streptokokový syndrom toxického šoku – život ohrožující stav vyvolaný různými druhy streptokoků
- Bakteriémie vyvolaná Staphylococcus aureus – význam správného přístupu k diagnostice a léčbě
- Boj s rezistencí mikroorganismů na antibiotika
- Aktualizace českých doporučených postupů pro laboratorní diagnostiku infekcí vyvolaných Clostridium difficile
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



