-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Současné metody analýzy mikroRNA
Current Methods of microRNA Analysis
Background:
MicroRNA (miRNA) are a class of short non-coding RNA molecules that regulate gene expression at the post-transcription level by binding to mRNA. By affecting many physiological processes, including cellular proliferation, differentiation, and apoptosis, they have a major impact on the development of cancer as well as other diseases. Hence, miRNAs could serve as potential tumor biomarkers in e.g. early diagnostics, predicting responses to therapy, monitoring relapse, and molecular classification of tumors.
Aim:
miRNA detection requires various sophisticated strategies due to the small size, sequence similarity among family members, and often very low levels of miRNAs in analyzed samples. This review describes standard techniques of miRNA detection, such as the reverse transcriptase polymerase chain reaction, microarrays, and next-generation sequencing, and compares several commercially available detection kits. Major emphasis is given to newly developed technologies and methods, which could make the analysis cheaper and quicker. We present, for instance, alternative amplification techniques (isothermal amplification and the hybridization chain reaction), different types of nanomaterials, special proteins used in miRNA analysis, and a number of biosensors utilizing optical or electrochemical detection.
Conclusion:
The importance of miRNA has led to a huge increase in the number of new methods. Most of them, however, have not been tested on clinical material, and thus it is difficult to assess their potential usefulness in routine practice. Their commercial application strongly depends on strict validation with standard techniques using not only model systems, but also clinical samples.
Key words:
microRNA – gene expression regulation – tumour biomarkers – reverse transcription PCR – biosensors
This work was supported by MEYS – NPS I – LO1413 and GAČR 17-08971S.
The authors declare they have no potential conflicts of interest concerning drugs, products, or services used in the study.
The Editorial Board declares that the manuscript met the ICMJE recommendation for biomedical papers.
Accepted: 9. 7. 2018
Autori: Bartošík Martin; Jiráková Ludmila
Pôsobisko autorov: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
Vyšlo v časopise: Klin Onkol 2018; 31(Supplementum 2): 93-101
Kategória: Přehled
doi: https://doi.org/10.14735/amko20182S93Súhrn
Východiska:
ImikroRNA (miRNA) jsou krátké nekódující molekuly RNA regulující genovou expresi vazbou na mRNA. Ovlivňují řadu fyziologických procesů, vč. buněčné proliferace, diferenciace nebo apoptózy, a mají tak výrazný vliv na vývoj nádorových i jiných onemocnění. Představují proto slibnou skupinu nádorových biomarkerů využitelných např. v časné diagnostice, v predikci odpovědi na léčbu, při záchytu relapsu nebo při klasifikaci nádorů do molekulárních subtypů.
Cíl:
Detekce miRNA vyžaduje různé sofistikované strategie zejména kvůli jejich krátké délce, značné sekvenční podobnosti mezi miRNA patřící do stejné rodiny, i pro jejich často velmi nízkou hladinu ve studovaném vzorku. V této práci jsou zmíněné standardně používané metody, např. reverzní transkripce s následnou polymerázovou řetězovou reakcí, microarrays nebo sekvenování nové generace, a rovněž jsou porovnány různé komerčně dostupné sady pro detekci miRNA. Hlavní důraz je kladen na nově vyvíjené technologie a metody, které by mohly současnou analýzu zlevnit nebo urychlit. Představeny jsou např. alternativní amplifikační techniky (izotermální amplifikace, hybridizační řetězová reakce), různé typy nanomateriálů, speciální proteiny se schopností vázat miRNA, a řada biosenzorů využívající optickou nebo elektrochemickou detekci.
Závěr:
Důležitost miRNA vedla k obrovskému nárůstu počtu nově vyvíjených metod. Většina z nich ovšem nebyla testována na klinickém materiálu, takže je těžké určit jejich eventuální aplikovatelnost do praxe. Pro komerční využití nových metod bude proto nutné provést jejich přísnou validaci pomocí standardních metod nejenom na modelových systémech, ale zejména u klinických vzorků.
Klíčová slova:
mikroRNA – regulace genové exprese – nádorové biomarkery – RT-PCR – biosenzory
Úvod
O důležitosti a významu mikroRNA (miRNA) již dnes není pochyb. Tyto krátké nekódující molekuly RNA s průměrnou délkou 22 bází kontrolují expresi mnoha proteinů, a ovlivňují tak řadu fyziologických procesů, jako je buněčná proliferace, diferenciace, apoptóza, vývoj a růst orgánů, imunita nebo metabolizmus [1]. Od objevu první miRNA v háďátku obecném (Caenorhabditis elegans) uplynulo už čtvrtstoletí [2], během kterého přibylo do celosvětové databáze těchto molekul, známé jako miRBase [3], přes 38 000 nových sekvencí (verze 22, 04/2018), z toho bezmála 2 000 lidských miRNA. Když v roce 2002 skupina profesora Croceho učinila významný objev popisující deregulaci miR-15 a miR-16 u chronické lymfocytární leukemie [4], odstartovala se tím doslova nová éra výzkumu miRNA jakožto potenciálních biomarkerů nádorových onemocnění.
Vznik nové miRNA neboli tzv. biogeneze probíhá v několika krocích: 1. syntéza dlouhých primárních transkriptů (pri-miRNA) z nekódujících oblastí DNA pomocí RNA polymerázy II; 2. jaderné štěpení pri-miRNA pomocí endonukleázy Drosha na cca 60–70 nukleotidů dlouhou vlásenkovou RNA známou jako pre-miRNA; 3. transport pre-miRNA z jádra do cytoplazmy pomocí exportinu-5; 4. štěpení endonukleázou Dicer, která z pre-miRNA vytvoří krátký duplex miRNA-miRNA* o délce cca 22 bází; 5. vazba duplexu na katalyticky aktivní argonautový (AGO) proteinový komplex, čímž dochází k vyloučení a degradaci miRNA* vlákna za vzniku tzv. RISC komplexu (RNA-induced slencing complex). Jedná se o ribonukleoproteinový komplex obsahující vedoucí vlákno miRNA, odpovědný za samotnou post-transkripční regulaci genové exprese.
Již první publikované výsledky naznačily, že miRNA ovlivňuje genovou expresi vazbou na nepřekládanou oblast na 3’ konci mRNA (3’ untranslated region – UTR), i když konkrétní mechanizmus byl dlouho předmětem diskuzí. V současnosti převažuje model rozštěpení cílové mRNA v případě dostatečné komplementarity miRNA a represe translace, pokud je miRNA komplementární jen zčásti [5]. Jelikož jsou vazebná místa krátká a nemusí být plně komplementární, je běžné, že jedna miRNA sekvence reguluje translaci stovek mRNA a že translace jedné mRNA může být regulována několika miRNA současně.
Velikostí, chemickým složením i funkcí jsou miRNA velmi blízké jinému typu krátkých nekódujících RNA molekul, a to siRNA (small interfering RNA). Je mezi nimi ovšem několik významných odlišností. Za prvé, geny kódující miRNA jsou odlišné od ostatních kódujících oblastí a z původně jednořetězcového transkriptu vniká úpravami finální miRNA; oproti tomu siRNA pochází z dlouhé dvouřetězcové RNA, často exogenního původu, z transpozonů či virů [6]. Za druhé, sekvence miRNA jsou vysoce konzervovány napříč příbuznými organizmy, zatímco siRNA tuto vlastnost nevykazují. miRNA dále potlačují expresi často nesouvisejících vzdálených genů, kdežto siRNA obvykle ovlivňuje to místo genomu, ze kterého pochází [6].
Jak už bylo zmíněno, funkce miRNA jsou různorodé. Nejjednodušším způsobem, jak určit funkci dané miRNA, je sledovat změnu fenotypu na nižších organizmech po její deregulaci. Příkladem jsou lin-4 nebo let-7 u hlístic regulující přechod mezi jednotlivými larválními stadii [7]. Pro většinu miRNA však tyto experimenty chybí, a jejich funkci je proto nutno pouze odhadnout. K tomu slouží různé bioinformatické nástroje predikující cílovou mRNA, na kterou se daná miRNA váže [8], a předpovídající možný účinek miRNA, který se až následně ověřuje experimentálně. Mezi nejznámější bezplatné online nástroje patří miRanda [9], TargetScan [10], miRWalk [11] nebo miRDB [12].
Role miRNA v karcinogenezi
Bylo prokázáno, že miRNA mohou regulovat většinu aspektů maligní transformace, jako je produkce růstových signálů, deregulace buněčného cyklu, blokování apoptózy či podpora angiogeneze a metastazování. Exprese miRNA je tkáňově i nádorově specifická, přičemž abnormální hladiny miRNA v nádorech mají zásadní vliv na vývoj onemocnění [13]. Obecně vzato dělíme miRNA do dvou skupin, a to na tumor supresorové a onkogenní (obr. 1). Tumor supresorové miRNA tlumí expresi onkogenů, a oproti zdravé tkáni tak mají v nádorech výrazně sníženou hladinu. Příkladem je rodina miR-34, jejímiž cíli jsou transkripty mnoha onkogenů (např. BCL-2, c-MYC nebo MDM4) a která je sama transkripčně regulována nádorovým supresorovým proteinem p53. Mezi tumor supresorové miRNA dále řadíme např. rodiny let-7 a miR-200, které jsou rovněž častým předmětem studií, např. u nádorů prsu a plic [14].
Obr. 1. Role miRNA v karcinogenezi. 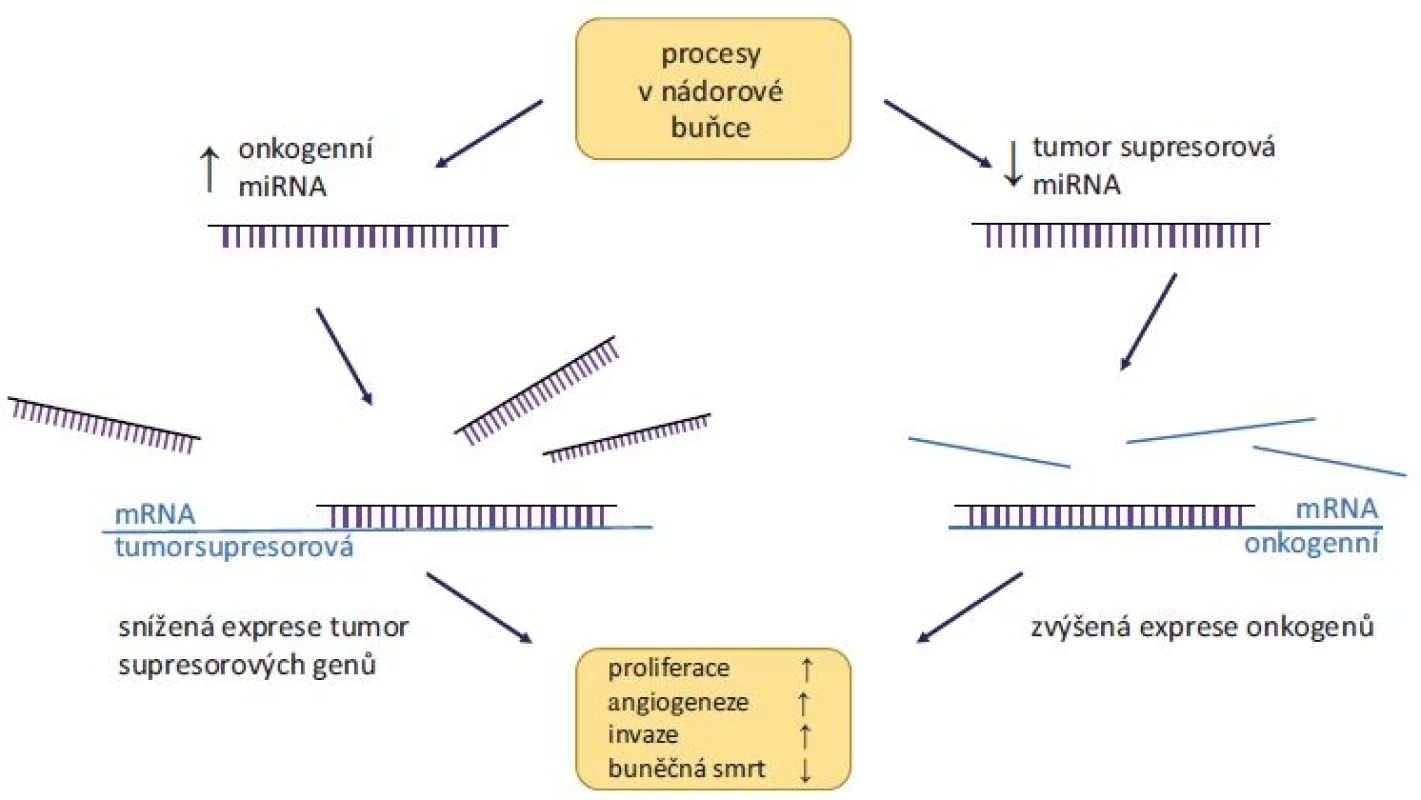
Vlevo – zvýšená hladina onkogenní miRNA vede ke snížené expresi tumor supresorových genů. Vpravo – snížená hladina tumor supresorových miRNA nedostatečně inhibuje expresi onkogenů. Oproti tomu onkogenní miRNA, jejichž funkcí je inhibice tumor supresorových genů, jsou v nádorech nadměrně exprimovány, a jejich hladiny jsou tudíž vyšší v porovnání se zdravou tkání. Asi nejvíce studovanou onkogenní miRNA je miR-21, kódovaná v chromozomální oblasti 17p23.2. Zvýšená amplifikace této oblasti byla pozorována u řady nádorových onemocnění, především pak nádorů prostaty, vaječníků, prsu nebo plic [15]. Experimentálně pak bylo prokázáno, že při indukci miR-21 v nádorových modelech (extrahované buňky plicního karcinomu injektované do myši) došlo ke zvýšení tumorigeneze [16]. Dalšími příklady často studovaných onkogenních miRNA jsou miR-155, miR-210 nebo miR-221 díky jejich zvýšené hladině v nádorech [14].
Z výše uvedeného vyplývá, že miRNA představují velmi slibnou skupinu nádorových biomarkerů, navíc dobře korelujících se známými klinicko-patologickými charakteristikami [13]. Jsou detekovatelné jednak v nádorové tkáni, kde fungují zejména jako prognostické nebo prediktivní biomarkery či zpřesňují molekulární klasifikaci nádorů, tak i v tělních tekutinách (krev, moč, sliny atd.) pro potřeby časné diagnostiky onemocnění nebo záchytu relapsu. Stejně tak se ukazuje důležitost miRNA v patologii solidních nádorů, např. u kolorektálního a renálního karcinomu, nádorů plic a prostaty, melanomu nebo glioblastomu [17], ale i u hematoonkologických malignit [18]. Můžeme se ptát, proč vůbec dochází ke změnám v expresi miRNA v nádorové tkáni. Možností je hned několik – během karcinogeneze může docházet k poškození těch částí chromozomů, které kódují geny pro miRNA [19]; časté jsou i epigenetické mechanizmy, např. metylace promotoru genu tumor supresorové miRNA [20]; v neposlední řadě může docházet ke změnám v expresi proteinů účastnících se biogeneze miRNA, jako jsou endonukleázy Drosha nebo Dicer. Snížená exprese těchto proteinů byla zaznamenána především u nádorů se špatnou odpovědí na léčbu.
miRNA by mohla sloužit nejenom jako biomarker, ale i jako potenciální terapeutický cíl nádorových onemocnění. Existují dva přístupy cílené regulace miRNA. První z nich se zaměřuje na snížení hladiny onkogenní miRNA. Označujeme ho jako inhibiční a jeho principem je aplikování látek, tzv. anti-miRs, se sekvencí komplementární k cílové miRNA. Anti-miRs mají strukturu upravenou tak, aby silněji a snadněji vázaly cílové molekuly (jsou to např. uzamčené nukleové kyseliny (locked nucleic acid – LNA)) a současně odolávaly nukleázám vyskytujícím se běžně v těle (modifikace 2-O metylací). Druhým terapeutickým přístupem je umělé zvyšování hladiny tumor supresorové miRNA, tzv. substituční terapie pomocí miRNA mimics, která má pomoci navodit fyziologický stav. Díky tomu, že se tyto molekuly v těle běžně vyskytují, je pozorováno méně vedlejších účinků než u jiných protinádorových léčiv [21].
Kromě protinádorové léčby se vývoj léčiv cílených na miRNA zaměřuje také na virová a zánětlivá onemocnění, neurologické a kardiovaskulární poruchy a na onemocnění spojená s poruchami metabolizmu. Nejdále je prozatím v testování léčivo Miravirsen (vyvinuto v Santaris Pharma), které potlačuje zvýšenou expresi miR-122 u hepatitidy C a které bylo v roce 2017 testováno v pokročilejší klinické studii fáze II [22].
Standardní metody stanovení miRNA
Při detekci miRNA je potřeba překonat mnohá úskalí. Vzhledem k jejich krátké sekvenci nelze například u polymerázové řetězové reakce (polymerase chain reaction – PCR) použít tradiční primery, neboť jejich sekvence by pokrývaly buď celou miRNA, nebo by v opačném případě byly značně krátké, a tudíž velmi nespecifické. Další problém spočívá ve značné sekvenční podobnosti mezi miRNA, které patří do stejné rodiny (obvykle vykazují i podobné funkce a rozmístění v genomu [23]). V rámci jedné miRNA rodiny se sekvence často liší pouze jediným nukleotidem, a je proto vyžadována velmi vysoká specificita stanovení. Výzvou může být i velmi nízká hladina hledané miRNA, často nedosahující > 0,01 % z celkové RNA izolované z biologického materiálu [24]. Nicméně stanovení miRNA má i výhody, např. zvýšenou odolnost vůči enzymům degradujícím RNA (RNázám) [25].
V počátcích detekce miRNA se navázalo na již zavedené a běžně používané metody pro analýzu molekul RNA, u nichž byl pouze modifikován postup, a podmínky byly přizpůsobeny krátkým strukturám. Tyto metody jsou ověřené praxí a jsou využívány dodnes (tab. 1). Řadí se mezi ně kvantifikace RNA založená na reverzní transkripci a následné PCR (reverse transciption polymerase chain reaction – RT-PCR). Metoda byla zavedena pro běžnou analýzu transkriptomu a spočívá v přepisu RNA templátu do DNA pomocí reverzní transkriptázy a následné kvantifikaci získané DNA pomocí PCR se specificky značenou fluorescenční sondou nebo nespecifickou interkalační barvičkou. Ve výsledku je detekován nárůst fluorescence, z níž je možno při porovnání s hladinou mRNA tzv. housekeeping genu (genu se stabilní transkripcí) vyhodnotit původní množství miRNA ve vzorku. Pro tento typ detekce, stejně jako i pro další níže zmíněné metody, již existují komerčně dostupné sady. Detekci miRNA na principu RT-PCR lze provádět například produkty „miScript miRNA PCR Array“ nebo „miRCURY LNA miRNA PCR Assay“ firmy Qiagen, která poskytuje i bezplatný software pro zpracování dat. Obdobnou variantu nabízí také firma ThermoFisher Scientific, která pod komerčním označením „TaqMan™ MicroRNA Assays“ nabízí sady roztoků na reverzní transkripci, PCR a fluorescenčně značené primery pro konkrétní miRNA. Další volbou může být sada produktů „Luna® Universal“ firmy New England Biolabs. Metoda stanovení miRNA pomocí RT-PCR je běžně využívaná, avšak vyžaduje nadstandardní vybavení, jakým je LightCycler, tedy přístroj měřící intenzitu fluorescence v průběhu amplifikace.
Tab. 1. Porovnání standardních a nově vyvíjených technologií pro detekci mikroRNA. 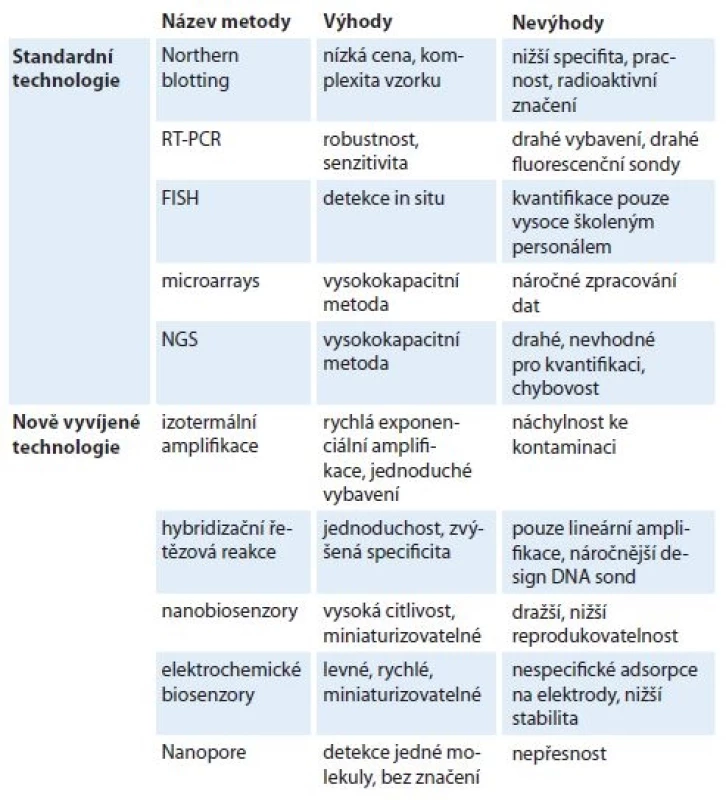
RT-PCR – polymerázová řetězová reakce, FISH – fl uorescenční in situ hybridizace, NGS – sekvenování nové generace Jiným postupem detekce miRNA, často využívaným in situ, je metoda založená na hybridizaci cílové molekuly a následné detekci pomocí fluorescenčně (fluorescent in situ hybridization – FISH) či radiologicky značených sond. Využití techniky v histologických či bioptických preparátech umožňuje sledovat expresi dané miRNA s důrazem na lokalizaci v buňce či tkáni. Při použití tohoto principu in vitro se využívá i přenos separované celkové RNA vzorku z gelu na membránu (Northern blotting) a následná detekce miRNA autoradiograficky pomocí fotografického papíru nebo moderněji kamerou s vysokým rozlišením. Modifikovanou verzi této metody nabízí např. firma Invitrogen pod označením „mirVana™ miRNA Detection Kit“.
Alternativou hybridizační metody jsou tzv. microarrays, do češtiny překládané jako „čipy“. Sonda je v tomto případě imobilizovaná na povrchu čipu a hybridizuje s komplementární miRNA z analyzovaného vzorku, která je před nanesením na čip fluorescenčně značena. Výhodou tohoto uspořádání je značná miniaturizace. Zařazení mezi tzv. vysokokapacitní metody, tedy umožnění analýzy většího počtu vzorků najednou, ji činí vhodnou i pro klinické účely. Nevýhodou je však nutnost pořízení vysokorozlišovací kamery, drahých fluorescenčních značek a taky větší náchylnost k chybám [26].
Nejnovější přístupy využívají moderní technologie, jako např. sekvenování nové generace (next generation sequencing – NGS) [27]. Hlavní nevýhodou této metody při aplikaci do klinické praxe je jistě její finanční náročnost. Při vyšším počtu vzorků charakterizovaných při jedné analýze by sice klesla cena, avšak toto s sebou nutně přináší pokles kvality samotné analýzy. Metoda, ačkoliv je finančně i časově náročná, pomohla objevit sekvence mnoha dříve nepublikovaných miRNA [28]. Sadu reagencií nabízí například firma New England Biolabs (NEBNext®) pro spojení se sekvenátory značky Illumina.
Technologicky nejnáročnější a komerčně nejnověji dostupnou metodu na principu hybridizace nabízí firma NanoString pod označením „nCounter® miRNA Assays“. Výrobce udává vysokou citlivost detekce, a to až jednu stanovovanou molekulu na buňku. Vysoká specificita je zaručena dvěma sondami, z nichž jedna nese kombinaci fluorescenčních značek emitující specifický signál v podobě čárového kódu, což dává možnost paralelního stanovení více různých miRNA sekvencí v jednom vzorku. Komplex miRNA a sond je adsorbován na povrch, který je poté snímán kamerou s vysokým rozlišením (obr. 2). Výraznou nevýhodou je ovšem nutnost pořízení dražší instrumentace (podobně jako v případě sekvenování).
Obr. 2. Princip detekce miRNA pomocí technologie NanoString. miRNA je nejdřív a) spojena ligací s miRtag sekvencí, b) řetězec miRNA-miRtag je hybridizován s vazební a fluorescenční sondou, c) výsledný komplex je pak (A) imobilizován na sklíčku, (B) elektrostaticky přitažen k povrchu, (C) a 5‘ konec fluorescenční sondy hybridizován s další biotinylovanou sondou, která zafi xuje řetězec v požadované orientaci. Pomocí kamery d) se stanoví umístění a počet jednotlivých fluorescenčních značek. 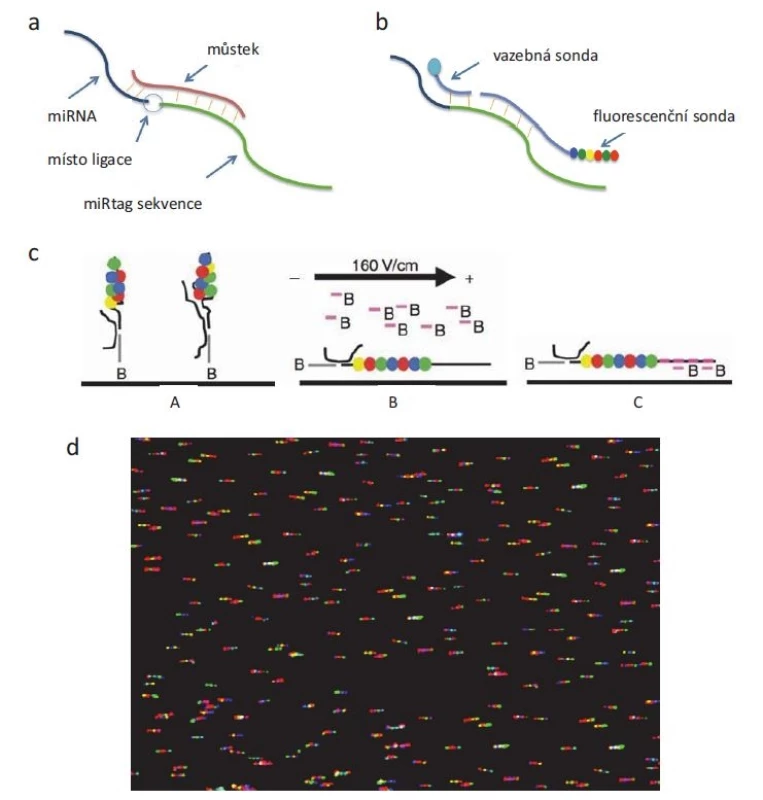
Upraveno podle [29] se souhlasem Macmillan Publishers Limited. Vzhledem k tomu, že jedna miRNA dokáže regulovat desítky genů, je někdy obtížné správně interpretovat změněné hladiny miRNA. Lze rovněž pozorovat značnou nekonzistentnost v publikovaných studiích o hladinách miRNA u různých patologických stavů, a proto je často obtížné vybrat jednoznačně průkazný biomarker predikce onemocnění, jeho progrese či odpovědi na léčbu. Značná variabilita může být rovněž dána použitou metodou analýzy či subjektivní chybou při vyhodnocování dat a vylučováním odlehlých hodnot. Úspěšným příkladem je patentovaný diagnostický test pro detekci hladin miR-31-5p, miR-93-5p a miR-191-5p v močovém supernatantu, kterým lze odlišit pacienty s karcinomem močového měchýře od zdravých jedinců [30].
Současné trendy
Každá z výše zmíněných metod, běžně používaných ve výzkumu miRNA, má pochopitelně kromě svých pozitiv i určitá negativa. I proto jsou neustále vyvíjeny nové technologie a metodologie, které se snaží překonat tyto limitace, příp. snížit celkové náklady analýzy. Je potřeba si však uvědomit, že se jedná o nesourodou skupinu různých strategií a přístupů, a to jednak z pohledu detekční metody (která je nejčastěji fluorescenční, chemiluminiscenční, kolorimetrická nebo elektrochemická), tak i způsobu přípravy vzorku, typu sondy nebo strategie pro zvýšení citlivosti. Každá detekční metoda má své výhody i nevýhody (tab. 1) a podrobněji jsou rozepsány v jiných přehledových článcích [24,26,31,32]. Mezi nejčastěji publikované přístupy patří tzv. elektrochemické biosenzory, u nichž hybridizace miRNA s vhodnou sondou, značení i samotné měření probíhá na povrchu elektrody. Elektrochemické metody totiž skýtají výhody ve formě levnější instrumentace, rychlého měření, možnosti miniaturizace nebo paralelní detekce vzorků, a jsou proto čím dál častěji používány pro detekci nejenom miRNA, ale i dalších nukleových kyselin [31,33–36]. Na druhé straně mnoho publikovaných přístupů (a to nejenom elektrochemických) nebylo doposud validováno na klinickém materiálu, či dokonce ani na buněčných liniích, takže je těžké určit jejich eventuální aplikovatelnost do praxe. Proto zde uvádíme jen ty práce, u kterých byla demonstrována klinická využitelnost, a mají tak jistý potenciál uspět.
Začněme u způsobu přípravy vzorků, který nejčastěji obnáší pre-amplifikaci dané miRNA, a to buď pomocí RT-PCR (popsanou výše), anebo tzv. izotermální amplifikace (IA). V porovnání s PCR jsou techniky IA obvykle rychlejší (často stačí < 1 hod) a instrumentálně méně náročné (nevyžadují termální cykler, protože reakce probíhá při konstantní teplotě). Existuje spousta typů IA, ovšem pro detekci miRNA je nejčastěji využívána amplifikace otáčivou kružnicí (rolling circle amplification – RCA), případně amplifikace zprostředkovaná smyčkou (loop-mediated isothermal amplification – LAMP) [37]. Jako příklad lze uvést bioluminescenční metodu využívající miRNA jako primer pro RCA reakci a luciferázu pro detekci pyrofosfátu uvolňovaného během amplifikace [38]. RCA byla použita i v práci Li et al [39], kde autoři detekovali tumor supresorovou miR-486-5p u pacientů s nemalobuněčným karcinomem plic, a to pomocí speciální vlásenkové sondy, která po hybridizaci s miRNA a následné změně konformace sloužila jako primer pro DNA polymerázu. Zajímavou alternativou pre-amplifikace, která nespadá mezi techniky IA, je tzv. hybridizační řetězová reakce (hybridization chain reaction – HCR) nevyžadující polymerázu ani jiné enzymy (obr. 3). miRNA slouží v reakci pouze jako iniciátor katalyzující otevření a následnou řetězovou hybridizaci dvou k sobě komplementárních DNA vlásenek, čímž dochází k vytvoření dlouhé DNA struktury složené z množství krátkých sekvencí [40]. Na rozdíl od technik IA a PCR, které vykazují exponenciální amplifikaci, HCR amplifikuje pouze lineárně, a je tudíž méně citlivá. Zajímavou strategii, kombinaci HCR a zlatých nanočástic představili Miao et al [41]. V přítomnosti miR-21 a dvou vlásenek došlo k vytvoření dlouhého polymeru DNA nesoucího výrazný záporný náboj a následně k elektrostatické vazbě kladně nabitých nanočástic způsobujících jejich agregaci. Výsledné agregáty byly detekovány kolorimetricky volným okem, co výrazně zjednodušovalo celý protokol.
Obr. 3. Princip hybridizační řetězové reakce. Po přidání dvou vlásenkových DNA sond (H1 – modrá a H2 – černá) do vzorku s cílovou miRNA (červená) dochází k otevření vlásenkových struktur a k řetězové hybridizaci mezi H1 a H2. 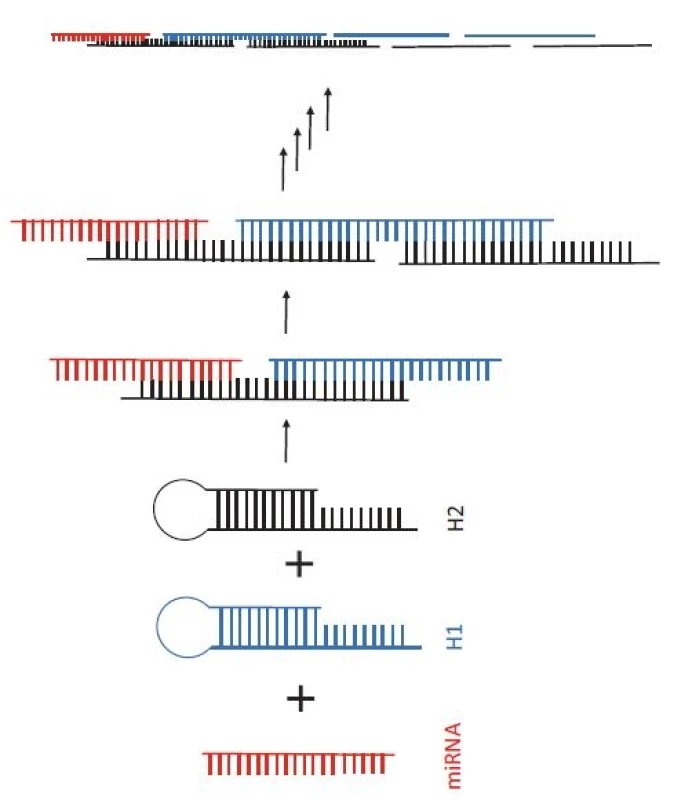
Nejčastějším typem sondy, a to nejenom u standardních metod detekce, ale i u nových přístupů, jsou krátké DNA oligonukleotidy, komplementární k hledané miRNA sekvenci. Pro zvýšení afinity k DNA nebo RNA však může být použit speciální syntetický analog nukleových kyselin, např. LNA (locked nucleic acid), obsahující methylenovou skupinu kovalentně spojující 2‘-kyslík a 4‘-uhlík na ribóze. Jelikož se jedná o modifikaci cukerného zbytku a nikoliv bází, klasické párování zůstává zachováno. LNA-RNA nebo LNA-DNA duplexy vykazují vyšší stabilitu díky zvýšené teplotě tání, lepší odolnost vůči nukleázám a rovněž jednodušší rozlišení nekomplementárních úseků. LNA modifikace najdeme v již zmíněném kitu „miRCURY LNA miRNA PCR Systém“ firmy Qiagen, příp. v metodách založených na in situ hybridizaci pro přímou detekci miRNA v buňkách [26].
Pre-amplifikace miRNA je sice krokem zvyšujícím citlivost metody, je ovšem časově náročná a náchylná na amplifikaci nespecifických sekvencí. Většina nově vyvíjených metod se snaží tento krok obejít a pokouší se zvýšit citlivost samotného měření (tj. amplifikovat signál) použitím různých sofistikovaných strategií. Pro tyto účely jsou velmi populárním nástrojem nanomateriály [31,36,42,43], sloužící nejenom jako značky pro samotnou detekci (např. kvantové tečky nebo zlaté nanočástice, vykazující dobré optické nebo elektrické vlastnosti), ale i jako vhodná platforma pro imobilizaci velkého množství sond (nanočástice, grafén, uhlíkové nanotrubky) díky jejich velké specifické ploše (tj. poměru velikosti plochy k objemu). Nedávno Su et al připravili zlaté nanostruktury s obrovskou specifickou plochou, které modifikovali DNA sondami pro následnou hybridizaci a detekci miR-21 v plicních nádorových buňkách A549 [44]. I když jsou nanomateriály slibným nástrojem v detekci miRNA, jsou stále relativně drahé a příliš málo používány při analýze klinického materiálu.
Další možností zvýšení citlivosti je použití tzv. duplex-specifické nukleázy (DSN). Jedná se o speciální enzym štěpící pouze dvoušroubovicovou DNA nebo DNA v rámci hybridních duplexů DNA-RNA, nikoliv však samotnou jednovláknovou DNA. Vazbou miRNA na komplementární DNA sondu a následném přidání DSN dochází k rozštěpení sondy a uvolnění miRNA, která je poté detekována různými způsoby [45]. Kolorimetrické stanovení není v tom případě vždy dostatečně senzitivní, citlivost se však dá zvýšit pomocí zlatých nanočástic [46]. Elektrochemické přístupy obvykle umožňují tímto postupem stanovit pouze jednu sekvenci miRNA [47], nejlepší se proto jeví aplikace multiplexové fluorescenční detekce za použití více fluoroforů připojených na jednotlivé DNA sondy [48]. I další proteiny specificky interagující s miRNA mohou pomoci při její detekci. Jedním z nich je virový protein p19 vázající krátké RNA-RNA duplexy. Torrente-Rodríguez et al vyvinuli elektrochemickou metodu pro stanovení miR-21 a miR-205 v buněčných liniích i prsních nádorech [49,50]. Postup ovšem vyžadoval použití dražších RNA sond, které byly po hybridizaci s miRNA izolovány pomocí magnetických kuliček modifikovaných proteinem p19. Později nahradili protein p19 speciální protilátkou S9.6, která váže krátké DNA-RNA duplexy, a mohly tak být použity levnější DNA sondy (obr. 4) [51].
Obr. 4. Elektrochemický přístup kombinující magnetické kuličky s G-proteinem, speciální protilátku (Ab S9.6) vázající duplex miRNA a DNA sondy, a detekci enzymatické reakce na uhlíkové elektrodě. Modifikované magnetické kuličky byly k elektrodě přitáhnuty magnetem. Jako substrát byla použita dvojice HQ/peroxid vodíku. Křenová peroxidáza oxidovala HQ na benzochinon, který byl zpětně redukován na elektrodě. 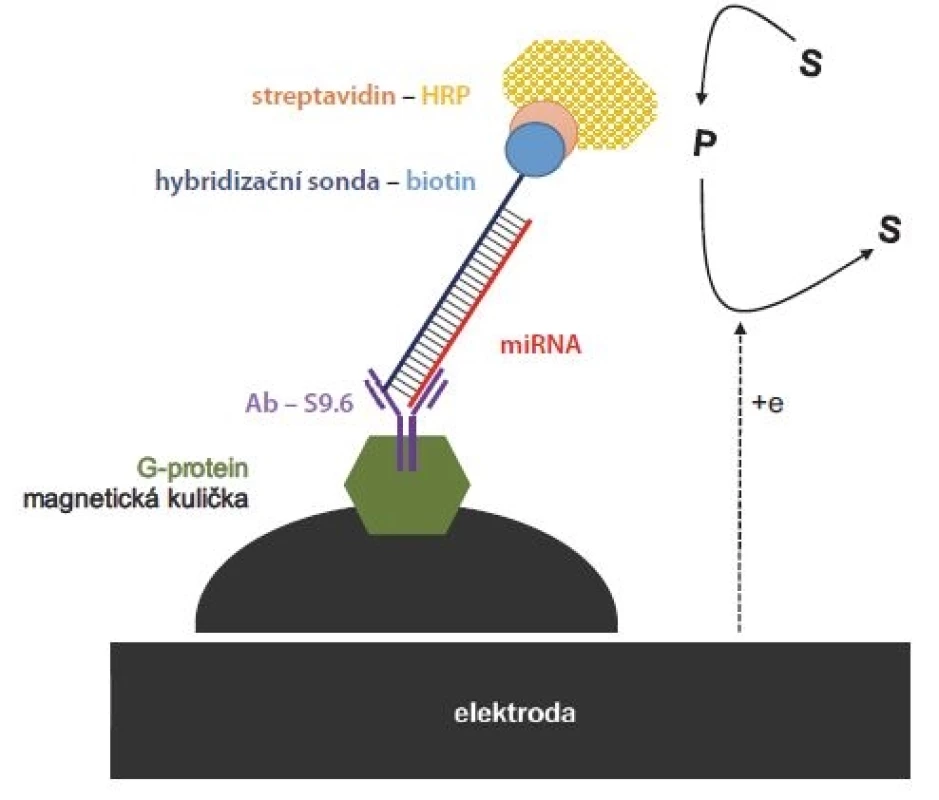
HRP – horseradish peroxidase, HQ – hydrochinon
Upraveno podle [51], se souhlasem American Chemical Society.Inovativní přístup detekce miRNA představuje tzv. NANOPORE technologie, v současnosti poskytována společností Oxford Nanopore Technologies. Je založena na měření změn elektrické vodivosti během transportu DNA nebo RNA přes protein a-hemolysin, tvořící cca 2nm pór v lipidické membráně. Pro tyto účely byly vyvinuty speciálně značené DNA sondy, které po vytvoření duplexu s cílovou miRNA a následném přestupu přes pór dočasně snížily vodivost uvnitř póru. Původní článek, publikovaný v Nature Nanotechnology [52], demonstroval úspěšnou detekci miR-155 v plazmě pacientů s rakovinou plic, kde tato miRNA vykazuje zvýšenou hladinu.
Závěr
Objevení souvislosti mezi hladinami miRNA a patogenezí nádorových či jiných onemocnění mělo za následek masivní rozvoj detekčních metod pro jejich detekci. K standardním metodám, jakými jsou RT-PCR, microarrays nebo NGS, postupně přibývají méně tradiční přístupy cílící zejména na snížení nákladů, urychlení měření nebo zjednodušení postupu. Tyto přístupy však čelí řadě výzev. Zřejmě největší z nich je aplikace vyvíjené metody na klinickém materiálu, která ve většině případů nebyla doposud demonstrována. Častým nedostatkem je i slabá validace pomocí standardních metod, velmi nízký počet detekovaných sekvencí (obvykle jenom jedna, přičemž vhodnější by bylo použití panelu miRNA), nízká reprodukovatelnost nebo neschopnost rozlišit vysoce homologní miRNA z jedné rodiny. Nedávné pokroky v oblasti nanotechnologií, biomateriálů nebo mikrofluidiky by však mohly pomoci překonat tyto překážky k dosažení levné, rychlé a dostatečně citlivé detekce miRNA v klinickém materiálu.
Práce byla podpořena grantem MŠMT – NPU I – LO1413 a GAČR 17-08971S.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
Mgr. Martin Bartošík, Ph.D.
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7 656 53 Brno
e-mail: martin.bartosik@mou.cz
Přijato: 9. 7. 2018
Zdroje
1. Huang Y, Shen XJ, Zou Q et al. Biological functions of microRNAs: a review. J Physiol Biochem 2011; 67 (1): 129–139. doi: 10.1007/s13105-010-0050-6.
2. Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell 1993; 75 (5): 843–854.
3. miRBase. miRBase: the microRNA database. [online]. Available from: http: //mirbase.org.
4. Calin GA, Dumitru CD, Shimizu M et al. Frequent deletions and down-regulation of micro-RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Nat Acad Sci USA 2002; 99 (24): 15524–15529. doi: 10.1073/pnas.242606799.
5. Didiano D, Hobert O. Molecular architecture of a miRNA-regulated 3 ‚ UTR. RNA 2008; 14 (7): 1297–1317. doi: 10.1261/rna.1082708.
6. Bartel DP. MicroRNAs: Genomics, biogenesis, mechanism, and function. Cell 2004; 116 (2): 281–297.
7. Lee H, Han S, Kwon CS et al. Biogenesis and regulation of the let-7 miRNAs and their functional implications. Protein Cell 2016; 7 (2): 100–113. doi: 10.1007/s13238-015-0212-y.
8. Riffo-Campos AL, Riquelme I, Brebi-Mieville P. Tools for sequence-based miRNA target prediction: what to choose? Int J Mol Sci 2016; 17 (12): pii: E1987. doi: 10.3390/ijms17121987.
9. microRNA. Predicted microRNA targets & target downregulation scores. [online]. Available from: http: //34.236.212.39/microrna/home.do.
10. TargetScan. Predicted microRNA targets. [online]. Available from: http: //www.targetscan.org/vert_72/.
11. miRWalk. The miRWalk database. [online]. Available from: http: //mirwalk.umm.uni-heidelberg.de/.
12. miRDB. miRDB target prediction. [online]. Available from: http: //www.mirdb.org/.
13. Slabý O, Svoboda M (ed). MikroRNA v onkologii. 1. vydání. Praha: Galén 2012.
14. Rupaimoole R, Slack FJ. MicroRNA therapeutics: towards a new era for the management of cancer and other diseases. Nat Rev Drug Discov 2017; 16 (3): 203–221. doi: 10.1038/nrd.2016.246.
15. Feng YH, Tsao CJ. Emerging role of microRNA-21 in cancer. Biomed Rep 2016; 5 (4): 395–402. doi: 10.3892/br.2016.747.
16. Hatley ME, Patrick DM, Garcia MR et al. Modulation of K-ras-dependent lung tumorigenesis by microRNA-21. Cancer Cell 2010; 18 (3): 282–293. doi: 10.1016/j.ccr.2010.08.013.
17. Slabý O (ed). MicroRNAs in solid cancer: from biomarkers to therapeutic targets. New York: Nova Science Publishers 2012.
18. Gounaris-Shannon S, Chevassut T. The role of miRNA in haematological malignancy. Bone Marrow Res 2013; 2013 : 269107. doi: 10.1155/2013/269107.
19. Calin GA, Croce CM. Chromosomal rearrangements and microRNAs: a new cancer link with clinical implications. J Clin Invest 2007; 117 (8): 2059–2066. doi: 10.1172/JCI32577.
20. Wilting SM, van Boerdonk RAA, Henken FE et al. Methylation-mediated silencing and tumour suppressive function of hsa-miR-124 in cervical cancer. Mol Cancer 2010; 9 : 167. doi: 10.1186/1476-4598-9-167.
21. Slabý O. MikroRNA vstupují do klinického testování. Klin Onkol 2012; 25 (2): 139–142.
22. Chakraborty C, Sharma AR, Sharma G et al. Therapeutic miRNA and siRNA: moving from bench to clinic as next generation medicine. Mol Ther Nucl Acids 2017; 8 : 132–143. doi: 10.1016/j.omtn.2017.06.005.
23. Kamanu TK, Radovanovic A, Archer JA et al. Exploration of miRNA families for hypotheses generation. Sci Rep 2013; 3 : 2940. doi: 10.1038/srep02940.
24. Graybill RM, Bailey RC. Emerging biosensing approaches for microRNA analysis. Anal Chem 2016; 88 (1): 431–450. doi: 10.1021/acs.analchem.5b04679.
25. Aryani A, Denecke B. In vitro application of ribonucleases: comparison of the effects on mRNA and miRNA stability. BMC Res Notes 2015; 8 : 164. doi: 10.1186/s13104-015-1114-z.
26. de Planell-Saguer M, Rodicio MC. Detection methods for microRNAs in clinic practice. Clin Biochem 2013; 46 (10–11): 869–878. doi: 10.1016/j.clinbiochem.2013.02. 017.
27. Hu Y, Lan W, Miller D. Next-generation sequencing for microRNA expression profile. Meth Mol Biol 2017; 1617 : 169–177. doi: 10.1007/978-1-4939-7046-9_12.
28. Kozomara A, Griffiths-Jones S. miRBase: integrating microRNA annotation and deep-sequencing data. Nucleic Acids Res 2011; 39 (Suppl 1): D152–D157. doi: 10.1093/nar/gkq1027.
29. Geiss GK, Bumgarner RE, Birditt B et al. Direct multiplexed measurement of gene expression with color-coded probe pairs. Nat Biotechnol 2008; 26 (3): 317–325. doi: 10.1038/nbt1385.
30. Juracek J, Peltanova B, Dolezel J et al. Genome-wide identification of urinary cell-free microRNAs for non-invasive detection of bladder cancer. J Cell Mol Med 2018; 22 (3): 2033–2038. doi: 10.1111/jcmm.13487.
31. Kilic T, Erdem A, Ozsoz M et al. microRNA biosensors: opportunities and challenges among conventional and commercially available techniques. Biosens Bioelectron 2018; 99 : 525–546. doi: 10.1016/j.bios.2017.08.007.
32. Tian T, Wang J, Zhou X. A review: microRNA detection methods. Org Biomol Chem 2015; 13 (8): 2226–2238. doi: 10.1039/c4ob02104e.
33. Bartošík M, Hrstka R. Bioelectrochemistry of nucleic acids for early cancer diagnostics – analysis of DNA methylation and detection of microRNAs. Rev Anal Chem 2017; 36 (1): 20160022. doi: 10.1515/revac-2016-0022.
34. Bartošík M, Paleček E, Vojtěšek B. Elektrochemická analýza nukleových kyselin, bílkovin a polysacharidů v biomedicíně. Klin Onkol 2014; 27 (Suppl 1): 53–60. doi: 10.14735/amko20141S53.
35. Paleček E, Bartošík M. Electrochemistry of nucleic acids. Chem Rev 2012; 112 (6): 3427–3481. doi: 10.1021/cr200303p.
36. Ciui B, Jambrec D, Sandulescu R et al. Bioelectrochemistry for miRNA detection. Curr Opin Electrochem 2017; 5 (1): 183–192. doi: 10.1016/j.coelec.2017.09.014.
37. Deng R, Zhang K, Li J. Isothermal amplification for microRNA detection: from the test tube to the cell. Acc Chem Res 2017; 50 (4): 1059–1068. doi: 10.1021/acs.accounts.7b00040.
38. Sun Y, Gregory KJ, Chen NG et al. Rapid and direct microRNA quantification by an enzymatic luminescence assay. Anal Biochem 2012; 429 (1): 11–17. doi: 10.1016/j.ab.2012.06.021.
39. Li Y, Liang L, Zhang C-y. Isothermally sensitive detection of serum circulating miRNAs for lung cancer diagnosis. Anal Chem 2013; 85 (23): 11174–11179. doi: 10.1021/ac403462f.
40. Bi S, Yue S, Zhang S. Hybridization chain reaction: a versatile molecular tool for biosensing, bioimaging, and biomedicine. Chem Soc Rev 2017; 46 (14): 4281–4298. doi: 10.1039/c7cs00055c.
41. Miao X, Ning X, Li Z et al. Sensitive detection of miRNA by using hybridization chain reaction coupled with positively charged gold nanoparticles. Sci Rep 2016; 6 : 32358. doi: 10.1038/srep32358.
42. Jamali AA, Pourhassan-Moghaddam M, Dolatabadi JE et al. Nanomaterials on the road to microRNA detection with optical and electrochemical nanobiosensors. TrAC Trends Anal Chem 2014; 55 : 24–42. doi: 10.1016/j.trac.2013.10.008.
43. Fiammengo R. Can nanotechnology improve cancer diagnosis through miRNA detection? Biomark Med 2017; 11 (1): 69–86. doi: 10.2217/bmm-2016-0195.
44. Su S, Wu Y, Zhu D et al. On-electrode synthesis of shape-controlled hierarchical flower-like gold nanostructures for efficient interfacial DNA assembly and sensitive electrochemical sensing of microRNA. Small 2016; 12 (28): 3794–3801. doi: 10.1002/smll.201601066.
45. Qiu X, Zhang H, Yu H et al. Duplex-specific nuclease-mediated bioanalysis. Trends Biotechnol 2015; 33 (3): 180–188. doi: 10.1016/j.tibtech.2014.12. 008.
46. Shen W, Deng H, Ren Y et al. A real-time colorimetric assay for label-free detection of microRNAs down to subfemtomolar levels. Chem Commun 2013; 49 (43): 4959–4961. doi: 10.1039/c3cc41565a.
47. Ren Y, Deng H, Shen W et al. A Highly sensitive and selective electrochemical biosensor for direct detection of microRNAs in serum. Anal Chem 2013; 85 (9): 4784–4789. doi: 10.1021/ac400583e.
48. Yin BC, Liu YQ, Ye BC. One-step, multiplexed fluorescence detection of microRNAs based on duplex-specific nuclease signal amplification. J Am Chem Soc 2012; 134 (11): 5064–5067. doi: 10.1021/ja300721s.
49. Torrente-Rodriguez RM, Campuzano S, Lopez-Hernandez E et al. Direct determination of miR-21 in total RNA extracted from breast cancer samples using magnetosensing platforms and the p19 viral protein as detector bioreceptor. Electroanalysis 2014; 26 (10): 2080–2087. doi: 10.1002/elan.201400317.
50. Torrente-Rodriguez RM, Campuzano S, Lopez-Hernandez E et al. Simultaneous detection of two breast cancerrelated miRNAs in tumor tissues using p19-based disposable amperometric magnetobiosensing platforms. Biosens Bioelectron 2015; 66 : 385–391. doi: 10.1016/j.bios.2014.11.047.
51. Torrente-Rodriguez RM, Ruiz-Valdepenas Montiel V, Campuzano S et al. Fast electrochemical miRNAs determination in cancer cells and tumor tissues with antibody-functionalized magnetic microcarriers. ACS Sensors 2016; 1 (7): 896–903. doi: 10.1021/acssensors.6b00 266.
52. Wang Y, Zheng D, Tan Q et al. Nanopore-based detection of circulating microRNAs in lung cancer patients. Nat Nanotechnol 2011; 6 (10): 668–674. doi: 10.1038/nnano.2011.147.
Štítky
Detská onkológia Chirurgia všeobecná Onkológia
Článok vyšiel v časopiseKlinická onkologie
Najčítanejšie tento týždeň
2018 Číslo Supplementum 2- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Kombinace metamizol/paracetamol v léčbě pooperační bolesti u zákroků v rámci jednodenní chirurgie
- Antidepresivní efekt kombinovaného analgetika tramadolu s paracetamolem
- Srovnání analgetické účinnosti metamizolu s ibuprofenem po extrakci třetí stoličky
-
Všetky články tohto čísla
- Proteogenomická platforma na identifikáciu nádorovo špecifických antigénov
- Využití mitochondriálních procesů v cílené terapii nádorových onemocnění
- Ferroptóza jako nový typ buněčné smrti a její role v léčbě rakoviny
- Možnosti využití p63 v bioptické diagnostice
- Význam membránových proteinů v léčbě nádorových onemocnění a možnosti jejich dalšího studia
- Vliv metylace DNA na vznik nádorových onemocnění
- Úloha proteinu HSP70 v nádorech a jeho využití jako terapeutický cíl
- Variabilita buněčné populace solidních nádorů a její význam pro diagnostiku a terapii nádorových onemocnění
- Úloha proteínu HSF1 v nádorovej transformácii
- Proteíny HDM2 a HDMX v ľudských nádoroch
- Prima-1 a APR-246 v léčbě nádorových onemocnění
- Acetylsalicylová kyselina a její potenciál v chemoprevenci kolorektálního karcinomu
- Exprese a funkční charakterizace miR-34c u karcinomu děložního hrdla
- Subpopulace cirkulujících myeloidních supresových buněk u pacientů s kolorektálním karcinomem – explorativní analýza jejich potenciálu jako biomarkeru
- Současné metody analýzy mikroRNA
- Klinická onkologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Vliv metylace DNA na vznik nádorových onemocnění
- Ferroptóza jako nový typ buněčné smrti a její role v léčbě rakoviny
- Možnosti využití p63 v bioptické diagnostice
- Současné metody analýzy mikroRNA
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



