Sledování metylace DNA u karcinomu vaječníků pomocí mikročipů.
Monitoring of DNA methylation in ovarian cancer using microarrays.
Objective:
The aim of this pilot study was whole-genome screening of DNA methylation in ovarian cancer patients. Monitoring of DNA methylation was focused on promoter regions and also on CpG islands.
Design:
Pilot study
Material and methods:
Our study included 6 samples obtained from patients with newly diagnosed ovarian cancer and 6 nonmalignant control samples of ovaries. All samples were analysed using a custom designed methylation microarrays based on the methylated DNA immunoprecipitation.
Results:
Using microarrays designed in collaboration with specialists of the HPST company we found in the tumor tissue tendency to overall DNA hypomethylation. Furthermore we managed to get a set of hypermethylated genes for confirmation analyses.
Conclusion:
DNA methylation changes are considered to be a one of the first changes during carcinogenesis. Due to this fact could be newly identified changes, in case of detection in plasma of ovarian cancer patient, used as screening markers in risk population. Monitoring of DNA methylation could contribute to improve the care of ovarian cancer patients, including possible financial saving due to early diagnosis and possible prediction of response to treatment.
Key words:
ovarian cancer, DNA methylation, microarrays.
Authors:
M. Chmelařová 1; E. Ruszová 2; J. Laco 3; E. Dvořáková 4; J. Smetana 5
; K. Hrochová 1; V. Palička 1
Authors‘ workplace:
Ústav klinické biochemie a diagnostiky, Fakultní nemocnice Hradec Králové
1; Oddělení lékařské genetiky, Fakultní nemocnice Hradec Králové
2; Fingerlandův ústav patologie, Fakultní nemocnice Hradec Králové
3; Porodnická a gynekologická klinika, Fakultní nemocnice Hradec Králové
4; Babákova Myelomová skupina, Oddělení patologické fyziologie Masarykova univerzita, Lékařská fakulta
5
Published in:
Klin. Biochem. Metab., 23 (44), 2015, No. 3, p. 100-104
Overview
Cíl:
Cílem této pilotní studie byl celogenomový screening metylace DNA u pacientek s nádory ovarií. Sledování metylace bylo zaměřeno jak do promotorových oblastí vybraných genů, tak do ostrůvků bohatých na CpG.
Typ studie:
Pilotní studie.
Materiál a metody:
Do studie bylo zařazeno šest vzorků získaných od pacientek s nově diagnostikovaným ovariálním karcinomem a šest vzorků nenádorových ovárií. Vzorky byly analyzovány pomocí zákazníkem navržených metylačních mikročipů založených na metylované-DNA imunoprecipitaci.
Výsledky:
Pomocí mikročipů navržených ve spolupráci se specialisty firmy HPST se nám podařilo poukázat u nádorové tkáně na tendenci k celkové hypometylaci DNA. Dále se nám podařilo získat soubor hypermetylovaných genů ke konfirmačním analýzám.
Závěr:
Vzhledem k tomu, že změny v metylaci DNA jsou jedním z prvních projevů v průběhu karcinogeneze, mohly by být nalezené změny, v případě jejich průkazu v plazmě pacientek, v budoucnu využity jako screeningové markery u rizikové populace. Monitorování metylace DNA by mohlo přispět ke zlepšení péče o nemocné s ovariálním karcinomem, včetně možné finanční úspory díky včasné diagnóze a případné predikci odpovědi na léčbu.
Klíčová slova:
karcinom vaječníků, metylace DNA, mikročipy.
Úvod
Ovariální karcinom je celosvětově nejčastější příčinou úmrtí pacientek s gynekologickými zhoubnými nádory. Vzhledem k tomu, že prozatím nemáme k dispozici žádnou efektivní metodu pro časnou detekci, ani neexistuje žádný ideální marker pro záchyt nebo screening [1], většina pacientek je diagnostikována až v pokročilých stádiích nemoci a pětileté přežití je nižší než 25 % [2]. Podobně, jako je tomu u ostatních nádorových onemocnění, dochází i u ovariálního karcinomu ve srovnání s normální zdravou ovariální tkání k celkové hypometylaci heterochromatinu a k lokální hypermetylaci CpG ostrůvků [3,4]. Ztráta metylace je způsobena zejména hypometylací opakujících se sekvencí DNA a demetylací kódujících regionů a intronů [5]. Hypermetylace CpG ostrůvků v místě genového promotoru je častým mechanismem inaktivace důležitých tumor supresorových genů, DNA mismatch repair genů a transkripčních faktorů [3]. Nejzajímavější vyhlídky pro onkology představuje potenciální reverzibilita epigenetických změn a využití alterací v metylaci DNA k predikci senzitivity různých nádorů k chemoterapii a jejím jednotlivým možnostem. Vzhledem k tomu, že změny v metylaci DNA se objevují jako jedny z prvních alterací v rozvoji karcinogeneze, mohlo by být sledování metylačních změn v plazmě pacientek v budoucnu využito k časnému záchytu ovariálního karcinomu. Změny v metylaci DNA lze sledovat pomocí různých molekulárně biologických metod [6]. Současný projekt je zaměřen na využití celogenomového screeningu jak promotorových oblastí, tak ostrůvků bohatých na CpG, a to metylačními mikročipy založenými na metylované DNA imunoprecipitaci (MeDIP).
Mikročipy jsou používány jak k celkové analýze DNA, tak k detekci metylace v promotorových oblastech genů. Principem mikročipů je imobilizace fragmentů jednovlákné DNA na pozitivně nabité nylonové membráně a hybridizace za vhodných podmínek. Hybridizační signály jsou nejčastěji detekovány chemiluminiscenčně. Mikročipy jsou komerčně připravované a poskytované různými firmami jako například Agilent, Illumina, NimbleGen a Affymetrix. DNA sondy, nebo prvky, které tvoří čip, jsou klíčovým faktorem při výběru nejvhodnější platformy k řešení dané otázky. Některé biočipy jako například od firmy Illumina jsou navrhované pro analýzu DNA po hydrogensiřičitanové modifikaci. Některé se hodí pro analýzu po DNA restrikci a některé jako například námi využívané microarrays od firmy Agilent jsou vhodné pro analýzu imunoprecipitované DNA.
Materiál a metody
Do studie bylo zařazeno šest tkáňových vzorků získaných od pacientek s nově diagnostikovaným ovariálním karcinomem ve věku 54-74 let (medián 64 let) a šest vzorků nenádorových ovárií, které byly získány od pacientek léčených pro nemaligní diagnózu (jako např. sestup dělohy s adnexektomií) či z pitev prováděných na Fingerlandově ústavu patologie ve věku 42-66 let (medián 65 let). Všechny vzorky byly revidovány zkušeným patologem. Nádorové vzorky byly klasifikovány dle WHO klasifikace. Soubor zahrnoval nízce a středně diferencované adenokarcinomy v časných stádiích (I-II). Histologicky se jednalo o tři vzorky serózního typu, dva vzorky endometroidního typu a jeden mucinózní adenokarcinom. Studie byla schválena Etickou komisí Fakultní nemocnice Hradec Králové.
DNA ze získaných vzorků byla izolována pomocí QiaAMP DNA Blood Midi Kitu (Qiagen). Následná fragmentace na LMW (Low Molecular Weight) DNA byla provedena dsDNA Shearasou™ (Zymo Research) a pročištěna kolonkami Clean-up (ZymoResearch). Vstupní množství pro MeDIP (Methylated-DNA Immunoprecipitation, Zymo Research) bylo stanoveno na 500 ng. Po obohacení a získání hypermetylované frakce DNA následovala dvoukolová WGA (Whole Genome Amplification) (Illustra WGA2, Elizabeth Pharmacon), jejímž úkolem bylo navýšit množství DNA pro vlastní značení (SureTaq Complete Labeling kitem, Agilent), tak abychom získali potřebné množství 2,5 µg značené DNA po přečištění opět kolonkami Clean-up. Hybridizace na vlastní custom čipy probíhala 40 hodin při 67°C v hybridizační pícce Agilent.
Design čipů (1x850K) spočíval ve sloučení čtyř komerčních prób-skupin Agilent (014791, 015233, 014707, 014706 a obohacení o kompletní monitor genů CDH13, GATA4, HNF1B, HTRA1, NTRK1, APC, MLH1, WIF1, THBS1, PAX5 a doplnění o miRNA TSS (Transcription Start Site)).
Arraye byly scanovány Agilent microarray HR scannerem a vyextrahovány v softwaru Feature Extraction v. 9.5.3.1. Primární data (txt soubory) byla analyzována v Agilent Genomic workbench software v. 6.5. Lite Edition, CH3 modulu, za využití výpočtu Z-scóre pro metylaci a znázornění klouzavého průměru pomocí trojúhelníkového vyhlazovacího algoritmu zohledňujícího nejbližší okolí bodu a citlivého i k malým variacím. K částečnému vyhodnocení dat byl také použit GeneSpring v. 13 software, Agilent.
Výsledky
Pomocí skel navržených ve spolupráci se specialisty firmy HPST jsme analyzovali metylaci DNA u šesti nádorových a šesti nenádorových vzorků ovárií. Podařilo se nám prokázat u nádorové tkáně již dříve publikovanou [5] tendenci k celkové hypometylaci DNA (obr. 1). Paradoxně k tomuto faktu je z obr. 2 patrné, že celkový počet statisticky významně rozdílných spotů je u nádorové skupiny z větší části v hypermetylované oblasti.


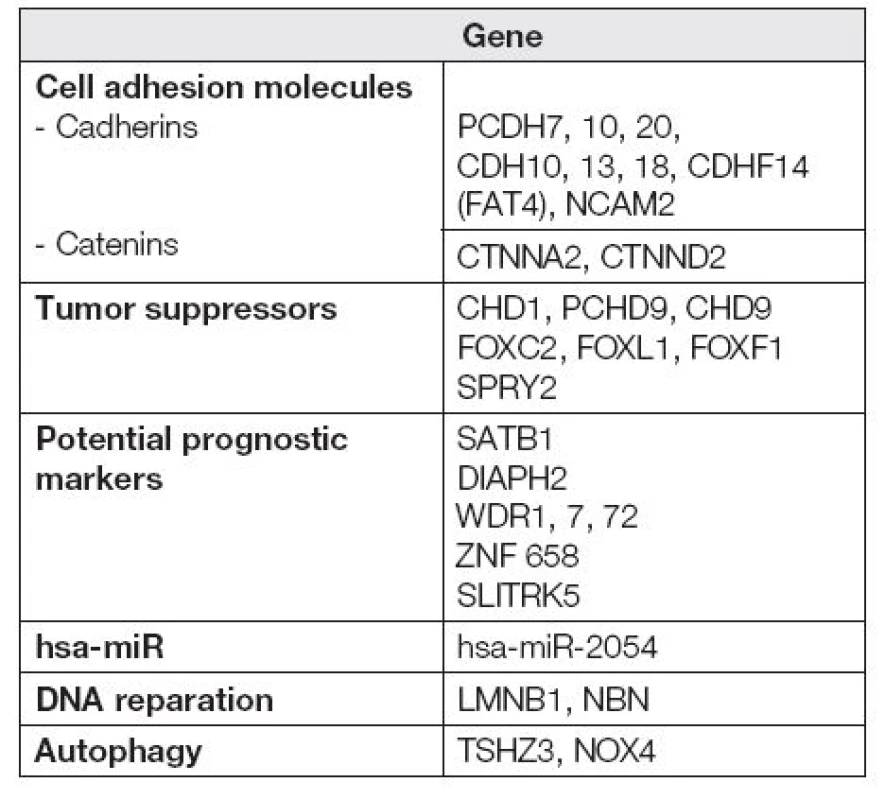
Dále se nám podařilo získat soubor hypermetylovaných genů ke konfirmačním analýzám, z toho v následující tabulce (Tabulka 1) je uvedeno zastoupení nejčastěji hypermetylovaných členů vybraných funkčních rodin.
Diskuse
Vzhledem k tomu, že ovariální karcinom patří mezi příčinu nejčastějšího úmrtí na gynekologické zhoubné nádory a k onemocnění se širokou variabilitou klinické odpovědi, jsou intenzivně studovány především molekulárně-biologické parametry doprovázející toto onemocnění. Důraz je kladen na markery, které by mohly být použity pro screening a mohly přesněji určit prognózu nebo predikovat odpověď na léčbu.
Pouze genetické alterace nejsou ovšem dostatečným vysvětlením všech změn doprovázejících nádorovou transformaci buňky, a to především změn v expresi jednotlivých genů. Proto je velké úsilí věnováno odhalování faktorů ležících mimo samotný sled nukleotidů. V této práci jsme se zaměřili právě na jednu z těchto epigenetických změn, a to na celogenomové sledování metylace DNA.
Pro maligní transformaci je rozhodující poškození zejména tří skupin genů – protoonkogenů, tumor supresorových genů a DNA reparačních genů. Mutace v onkogenech, které způsobují jejich trvalou aktivaci, nebo jejich zvýšená exprese umožňují buněčné dělení, které se v postižené buňce stává autonomním dějem [7]. Na akcelerované proliferaci nádorových buněk se podílí i ztráta negativních regulačních signálů buněčného dělení, které vyplývá z vyřazení tumor supresorových genů mutacemi nebo hypermetylacemi jejich promotorových oblastí. Dále dochází k poruchám reparačních mechanismů, které dovolují v postižené buňce tolerovat vznik mutací, které by za normálních okolností vedly k zástavě buněčného cyklu nebo apoptóze [8].
V procesu nádorové transformace mají nezastupitelnou roli rovněž geny související s mezibuněčnou adhezí. Kadheriny tvoří skupinu transmembránových proteinů podílejících se na stavbě mezibuněčných spojů. Jejich schopnost adheze (přilnutí) je podmíněna přítomností vápenatých kationtů v okolním prostředí [9]. Již vícekrát bylo uvedeno, že nádorové buňky vykazují změny v síle adherence v porovnání s buňkami zdravými, u nádorových buněk dochází zejména k oslabení adheze [10]. Mezi často studované kadheriny v souvislosti s ovariálním nádorovým bujením můžeme zařadit zejména H-kadherin (kadherin 13) kódovaný genem CDH13 [11,12] a E-kadherin kódovaný genem CDH1 [13,14]. Metylace rodiny protokadherinů (podskupina rodiny kadherinů) byla již dříve popsána u nádorů trávícího traktu [15,16], močového měchýře [17] prostaty [18] a nemalobuněčného karcinomu plic [19]. Nádory vaječníků v souvislosti s metylací protokadherinů prozatím studovány nebyly. Naše práce ukazuje na protokadheriny PCDH7, 10, 20 jako potenciálně vhodné cíle pro sledování metylace u ovariálního karcinomu. Naše výsledky tímto potvrzují, že oslabení mezibuněčné komunikace hraje klíčovou roli v narušení buněčné homeostázy [10].
Kadheriny regulují β-kateninovou buněčnou signalizaci a společně tak inhibují mitogenní signály [20]. Samotné kateniny pak představují buněčné proteiny podílející se na vazbě kadherinů a cytoskeletu, rovněž působí jako transkripční faktory a mají význam v ontogenezi. Studium kateninů v souvislosti s nádorovým bujením je z pohledu molekulární biologie v současné době zaměřeno zejména na analýzu potenciálních mutací [21], polymorfismů [22] a jejich exprese [23]. V budoucnu bude nutné zaměřit se na studium regulace proteinů kódovaných těmito geny i z hlediska epigenetického. Naše práce upozorňuje na CTNNA2 a CTNND2 jako potenciálně vhodné cíle pro sledování metylace u ovariálního karcinomu.
Neopomenutelnou roli v průběhu nádorového bujení hraje také autofagie [24]. Autofagie, do níž jsou zapojeny mimo jiné geny TSHZ3, NOX4, je jedním z mechanismů udržení buněčné homeostázy. V nejobecnějším slova smyslu se jedná o lysozomální rozklad proteinů. Autofagie má významnou roli při regulaci embryonálního a postnatálního vývoje. Významu autofagie u patologických stavů se v poslední době věnuje řada vědeckých týmů. S autofagozomálními procesy se pojí etiopatogeneze řady nervových, myodegenerativních a infekčních chorob. Dosavadní publikované výsledky jsou hodně daleko od řešení, proč v dané fázi nádorového bujení se chová autofagie jako tumorový supresor cestou aktivace imunitní odpovědi nebo jako tumorový promotor [25]. Je zřejmé, že studium molekulárně-biologických parametrů, včetně metylace DNA bude mít v budoucnu nezastupitelnou roli.
Pro onkology jsou u nádorových onemocnění velice důležité prognostické faktory, mezi něž můžeme zařadit i metylace specifických míst v DNA sekvenci. Prognostické markery představují ukazatele, díky nimž bude možné odhadnout další průběh onemocnění. Jedná se tedy o velice důležitý zdroj informací, na kterém závisí například strategie léčby. Naše práce poukazuje na několik kandidátních genů -SATB1, DIAPH2, WDR1, 7, 72, ZNF 658, SLITRK5, které by v budoucnu mohly sloužit jako vhodné prognostické markery.
Vzhledem k velké finanční náročnosti a nízkému počtu analyzovaných vzorků plánujeme v budoucnu potvrdit hypermetylaci vybraných genů pomocí citlivějších semi-kvantitativních metod (např. MS-q real-time PCR, pomocí MethylLight sond) na větší skupině pacientek.
V případě potvrzení významu metylace vybraných genů u nádorové skupiny vzorků můžeme uvažovat o dalším využití nalezených alterací s možným reálným dopadem v praxi, zejména v časné diagnostice ovariálního karcinomu, případně predikci odpovědi na léčbu.
Závěr
Vzhledem k tomu, že změny v metylaci DNA jsou jedním z prvních projevů v průběhu karcinogeneze, mohly by být nalezené změny, v případě jejich průkazu v plazmě pacientek, v budoucnu využity jako screeningové markery u rizikové populace.
Monitorování metylace DNA by také mohlo přispět ke zlepšení péče o nemocné s ovariálním karcinomem, včetně možné finanční úspory díky včasné diagnóze a případné predikci odpovědi na léčbu.
Tento projekt byl podpořen MZ ČR - RVO (FNHK00179906)
Do redakce došlo 30. 4. 2015
Adresa pro korespondenci:
Mgr. Marcela Chmelařová, Ph.D.
ÚKBD FN Hradec Králové
Sokolská 581
500 05 Hradec Králové
e-mail: ChmelarovaMarcela
Sources
1. Kučera, R., Topolčan, O., Presl, J. et al. Klinický přínos ROMA indexu při diagnostice ovariálního karcinomu. Klin. Biochem. Metab., 2012, 20 (41), p. 248-251.
2. Karlan, B. Y. Patients at High Risk for Ovarian Cancer Should Undergo Routine Screening. Clinical Ovarian Cancer, 2009, 2 (2), p. 83-89.
3. Barton, C. A., Hacker, N. F., Clark, S. J., O’Brien, P. M. DNA methylation changes in ovarian cancer: implications for early diagnosis, prognosis and treatment. Gynecol. Oncol., 2008, 109 (1), p. 129-39.
4. Chmelařová, M., Křepinská, E., Špaček, J. et al. Methylation analysis of tumor suppressor genes in ova-rian cancer using MS-MLPA. Folia Biol (Praha), 2012, 58 (6), p. 246-250.
5. Esteller, M. Epigenetics in Cancer. N Engl. J. Med., 2008, 358 (11), p. 1148-59.
6. Chmelařová, M. and Palička, V. How to analyze DNA methylation? J Turgut. Ozal. Med. Cent., 2013, 20 (3), p. 282-286
7. Croce, C. M. Oncogenes and Cancer. N Engl. J. Med., 2008, 358 (5), p. 502-511.
8. Sherr, C. J. Principles of Tumor Suppression. Cell, 2004, 116(2), p. 235-46.
9. Hulpiau, P., van Roy, F. Molecular evolution of the cadherin superfamily. Int. J. Biochem. Cell. Biol., 2009, 41(2), p. 349-69.
10. Jeanes, A., Gottardi, C. J., Yap, A. S. Cadherins and cancer: how does cadherin dysfunction promote tumor progression? Oncogene, 2008, 27(55), p. 6920-9.
11. Bol, G. M., Suijkerbuijk, K. P., Bart, J., Vooijs, M., van der Wall, E., van Diest, P. J. Methylation profiles of hereditary and sporadic ovarian cancer. Histopathology, 2010, 57 (3), p. 363-370.
12. Chmelařová, M., Křepinská, E., Špaček, J., Laco, J., Nekvindová, J., Palička, V. Methylation analysis of tumour suppressor genes in ovarian cancer using MS-MLPA. Folia Biol. (Praha), 2012, 58 (6), p. 246-50.
13. Yuecheng, Y., Hongmei, L., Xiaoyan, X. Clinical eva-luation of E-cadherin expression and its regulation me-chanism in epithelial ovarian cancer. Clin. Exp. Metastasis. 2006, 23 (1), p. 65-74.
14. Zhang, Q., Hu, G., Yang Q, Dong, R., et al. A multiplex methylation-specific PCR assay for the detection of early-stage ovarian cancer using cell-free serum DNA. Gynecol. Onco., 2013, 130 (1), p. 132-9.
15. Heitzer, E., Artl, M., Filipits, M., et al. Differential survival trends of stage II colorectal cancer patients relate to promoter methylation status of PCDH10, SPARC, and UCHL1. Mod. Pathol., 2014, 27 (6), p. 906-15.
16. Deng, J., Liang, H., Ying, G., Dong, Q., Zhang, L., Yu, J., Fan, D., Hao, X. Clinical significance of the methylated cytosine-phosphate-guanine sites of protocadherin-10 promoter for evaluating the prognosis of gastric cancer. J Am. Coll. Surg. 2014 Nov, 219 (5), p. 904-13.
17. Beukers, W., Hercegovac, A., Vermeij, M., et al. Hypermethylation of the polycomb group target gene PCDH7 in bladder tumors from patients of all ages. J Urol., 2013, 190 (1), p. 311-6.
18. Wang, L., Xie, P. G., Lin, Y. L., Ma, J. G., Li, W. P. Aberrant methylation of PCDH10 predicts worse biochemical recurrence-free survival in patients with prostate cancer after radical prostatectomy. Med. Sci. Monit., 2014, 20, p. 1363-8.
19. Tang, X., Yin, X., Xiang, T., et al. Protocadherin 10 is frequently downregulated by promoter methylation and functions as a tumor suppressor gene in non-small cell lung cancer. Cancer Biomark., 2012-2013, 12 (1), p. 11-9.
20. Yamada, S., Pokutta, S., Drees, F., Weis, W. I., Nelson, W. J. Deconstructing the cadherin-catenin-actin complex. Cell, 2005, 123 (5), p. 889-901.
21. Fanjul-Fernández, M., Quesada, V., Cabanillas, R., et al. Cell-cell adhesion genes CTNNA2 and CTNNA3 are tumour suppressors frequently mutated in laryngeal carcinomas. Nat. Commun., 2013, 4, p. 2531.
22. Wang, T., Chen, Y. H., Hong, H., et al. Increased nucleotide polymorphic changes in the 5’-untranslated region of delta-catenin (CTNND2) gene in prostate cancer. Oncogene, 2009, 28 (4), p. 555-64.
23. Burger, M. J., Tebay, M. A., Keith, P. A., et al. Expression analysis of delta-catenin and prostate-specific membrane antigen: their potential as diagnostic markers for prostate cancer. Int. J. Cancer, 2002, 100 (2), p. 228-37.
24. Xu, D. W., Zhang, G. Q., Wang, Z. W., Xu, X. Y., Liu, T. X. Autophagy in tumorigenesis and cancer treatment. Asian Pac. J. Cancer. Prev., 2015, 16 (6), p. 2167-75.
25. Mathew, R., Karantza-Wadsworth, V., White, E. Role of autophagy in cancer. Nat. Rev. Cancer, 2007, 7 (12), p. 961-7.
Labels
Clinical biochemistry Nuclear medicine Nutritive therapistArticle was published in
Clinical Biochemistry and Metabolism

2015 Issue 3
-
All articles in this issue
- Presepsin jako diagnostický a prognostický nástroj při posuzování sepse
- Využití solubilních cytokeratinových fragmentů v diagnostice jaterních metastáz
- Sledování metylace DNA u karcinomu vaječníků pomocí mikročipů.
-
Bias měření základních analytů krevního séra.
Výsledky a interpretace soudobých studií. - Problém přípustných limitů preciznosti v regulačních diagramech
- Ocenění
- Program přednáškových bloků
- Seznam posterů
- Abstrakta přednášek
- Abstrakta posterů
- Rejstřík autorů abstraktů
- Clinical Biochemistry and Metabolism
- Journal archive
- Current issue
- About the journal
Most read in this issue
- Presepsin jako diagnostický a prognostický nástroj při posuzování sepse
-
Bias měření základních analytů krevního séra.
Výsledky a interpretace soudobých studií. - Abstrakta posterů
- Problém přípustných limitů preciznosti v regulačních diagramech
