Detekce, identifikace a typizace dermatofytů molekulárně genetickými metodami
Detection, Identification and Typization of Dermatophytes by Molecular Genetic Methods
The molecular genetic methods (MGM) are used frequently in clinical microbiology to identify the agent of infection and also to its early direct detection in clinical specimens. In dermatomycology MGM supply or even substitute the classic time consuming and sophisticated diagnostic methods based on direct microscopy and cultivation. The high sensitivity and rapid agent detection available in few hours or days is an advantage of MGM that enables to start the early targeted therapy or to avoid an unnecessary antifungal treatment. However MGM designated to direct micromycetes detection does not guarantee their precise diagnostics which still requires the pathogen isolation before MGM are used to its identification. Article reviews the possibilities of MGM use in direct dermatophytes detection from clinical specimens and in species and strains identification.
Key words:
molecular genetic methods – dermatophytes – mycosis – detection – identification – typization
Authors:
A. Čmoková 1; P. Hamal 2; L. Svobodová 1; V. Hubka 1,3
Authors‘ workplace:
Katedra botaniky, Přírodovědecká fakulta, Univerzita Karlova v Praze
vedoucí doc. RNDr. Yvonne Němcová, Ph. D.
1; Ústav mikrobiologie, Lékařská fakulta Univerzity Palackého v Olomouci
přednosta prof. MUDr. Milan Kolář, Ph. D.
2; Laboratoř genetiky a metabolismu hub, Mikrobiologický ústav, Akademie věd České republiky, v. v. i., Praha
vedoucí Mgr. Miroslav Kolařík, Ph. D.
3
Published in:
Čes-slov Derm, 89, 2014, No. 4, p. 175-186
Category:
Innovations in Medicine
Overview
V laboratořích klinické mikrobiologie jsou stále častěji využívány molekulárně genetické metody (MGM), a to nejen ke správné identifikaci izolovaného původce onemocnění, ale také k jeho přímé, časné detekci v klinickém materiálu. Také v dermatomykologii MGM vhodně doplňují a někdy dokonce nahrazují klasické, časově a odborně náročné diagnostické postupy, založené zejména na přímé mikroskopii a kultivaci. Výhodou MGM je především vysoká citlivost a rychlost detekce agens, které je možné prokázat v řádu několika hodin až dnů. To může napomoci časnému zahájení cílené léčby, nebo naopak zabránit zbytečnému zatěžování pacienta antimykotiky. MGM určené pro přímou detekci mikromycet nám však i přes značný pokrok stále nezaručují jejich přesnou identifikaci. K tomuto účelu je stále třeba patogen nejprve izolovat, poté je možné využít některých dalších MGM k jeho určení. Cílem tohoto přehledového článku je shrnout možnosti využití MGM pro přímou detekci dermatofytů z klinických vzorků, dále pro identifikaci druhů a typizaci kmenů.
Klíčová slova:
molekulárně genetické metody – dermatofyty – dermatomykózy – detekce – identifikace – typizace
ÚVOD
Klasická diagnostika dermatofytóz většinou zahrnuje zhodnocení klinických projevů onemocnění, dále mikroskopické a kultivační vyšetření odebraných vzorků. Přímá mikroskopie je poměrně citlivá, obvykle však není možné tímto způsobem houbu identifikovat. Kultivace bývá zase poměrně málo úspěšná, zejména v případě onychomykóz, navíc je třeba původce inkubovat na půdách až několik týdnů. Dobře známá morfologická variabilita dermatofytů komplikuje používání klasických metod identifikace, tj. hodnocení vzhledu kultury a mycelia s reprodukčními strukturami. Také doplňkové fyziologické či biochemické testy (růst na speciálních médiích, produkce ureázy, perforace vlasu, aj.) nemívají stabilní výsledky a nejsou ani druhově specifické. Stále většího významu proto i v oblasti dermatomykologické diagnostiky nabývají molekulárně genetické metody (MGM) [15].
MGM užívané k přímému průkazu a identifikaci dermatofytů prodělaly v posledních letech rychlý vývoj, a tak již do značné míry mohou pomoci řešit některé nedostatky klasické diagnostiky [43]. Při detekci houbového patogenu přímo v klinickém vzorku dovedou výrazně zkrátit interval mezi odběrem a stanovením diagnózy, bohužel zpravidla neumožňují jeho přesnou identifikaci. K tomuto účelu musí být zvolen metodický postup umožňující v dodaném materiálu odhalit i velmi malé množství DNA mikromycety, ale nereagující na možnou přítomnost nukleových kyselin z jiných mikroorganismů. Naopak v případě využití MGM k přesnému druhovému určení izolátu bývá k dispozici prakticky neomezené množství DNA z kultury, a je proto třeba zvolit techniku zajišťující co nejpřesnější identifikaci izolátů. Vnitrodruhová typizace izolátů, metodicky založená na podobných principech, má důležitou úlohu v epidemiologii mykotických infekcí, neboť umožňuje odhalit zdroj nákazy a sledovat proces jejího šíření v populaci [21]. Role MGM v rámci současného pojetí diagnostiky dermatofytóz je přehledně demonstrována na obrázku 1. Podobně jako v jiných oborech mikrobiologie se i v rámci molekulární diagnostiky dermatomykóz používá pro určování klinických izolátů stále častěji ekonomicky výhodná a technicky jednoduchá hmotnostní spektrometrie MALDI-TOF [72]. I zde však MGM hrají důležitou roli, neboť databáze druhů hub by se měla opírat o spektra získaná z mikromycet identifikovaných analýzou genomu, navíc by zařazené kmeny měly mít i dostatečnou vnitrodruhovou diverzitu.
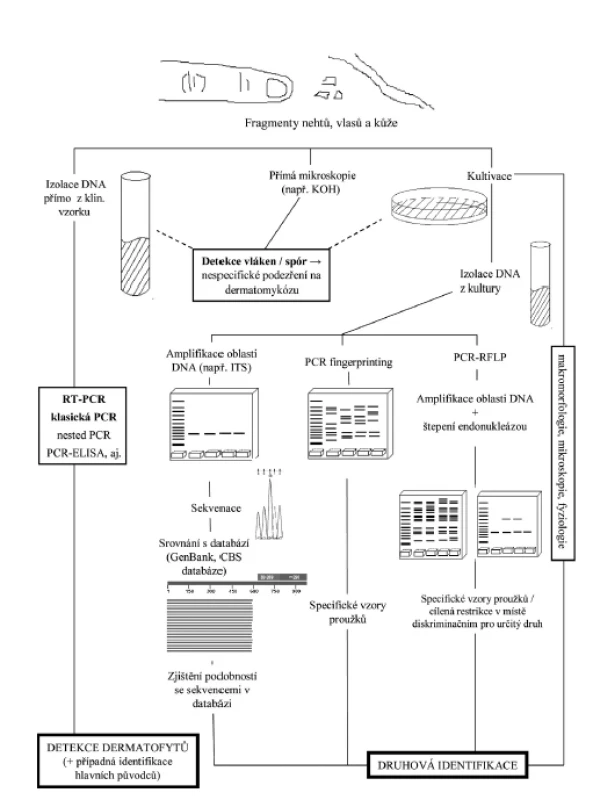
Při využívání MGM v diagnostické praxi je třeba zvolit optimální postup izolace DNA, dále vybrat odpovídající parametry PCR a konečně se rozhodnout pro nejvhodnější metodu k průkazu houbové DNA v klinickém vzorku, respektive k identifikaci nebo typizaci izolátu. A právě na uvedenou problematiku je zaměřen následující přehledový článek.
IZOLACE DNA
V případě detekce dermatofytů přímo v klinických vzorcích (zejména z kožních šupin, vlasů či nehtů) závisí výtěžnost a čistota DNA především na kvantitě a kvalitě odebraného materiálu. Množství DNA infekčního agens ve vzorku je totiž značně omezené, další komplikací bývá kontaminace genetickým materiálem pocházejícím z vláknitých hub přítomných v prostředí nebo z kvasinek kolonizujících kůži.
Při izolaci houbové DNA je nejprve třeba efektivně rozrušit buněčnou stěnu, dále lyzovat cytoplazmu a jadernou membránu, precipitovat proteiny a zbavit DNA řady inhibitorů, které mohou snižovat účinnost následně prováděné polymerázové řetězové reakce (PCR). Jednotlivé kroky lze provádět pomocí chemikálií známými metodickými postupy, nebo lze využít komerční soupravy, které mohou izolaci usnadnit. V takovém případě však může být problémem optimalizace metodického postupu, neboť některé chemikálie v diagnostických kitech bývají předmětem firemního tajemství.
Rozrušení buněčných stěn může být dosaženo manuální homogenizací zmrazeného vzorku ve třecí misce s tekutým dusíkem [25, 87], vortexováním se skleněnými nebo silikátovými kuličkami [19, 81], nebo opakovaným zmrazováním [60]. Lýza membrán je zajištěna detergenty (např. Triton X-100, laurylsíran sodný) s následným uvolněním nukleových kyselin do pufrovaného roztoku. Ten dále obsahuje chelátory, nejčastěji etylendiaminotetraoctovou kyselinu (EDTA), které vychytávají kationty vápníku, sloužící jako kofaktor nukleáz, čímž je zabráněno štěpení uvolněné DNA. Pro zvýšení čistoty nukleové kyseliny se někdy k lyzačnímu roztoku přidává proteináza K, která štěpí bílkoviny včetně histonů vázaných na DNA.
Z metodického hlediska je současným trendem ústup od tradiční, ale toxické fenol-chloroformové metody a redukce počtu kroků izolace DNA na minimum. Často používaným postupem je inkubace s proteinázou K přes noc a následné dokončení izolace komerční soupravou, nebo tzv. automatickým izolátorem [3, 6, 9, 91]. Patentovaná metoda izolace DNA z klinických vzorků ve dvou krocích, trvající 15 minut, se stala součástí produktu Dermatophyte PCR Kit (SSI Diagnostica, HillerŅd, Dánsko) [13, 56]. Ke stejnému účelu byly dále použity soupravy Genomic Prep Cells and Tissue DNA Isolation kit (Amersham Biosciences; Piscataway, USA) [49], Genomic DNA Purification kit (Gentra Systems; Minneapolis, USA) [14] a QIAamp DNA Minikit (Qiagen, Hilden, Německo) [6, 65]. Automatické izolátory DNA jsou přístroje, které byly vyvinuty tak, aby minimalizovaly manipulaci se vzorky a proces od určitého kroku automatizovaly. V diagnostice dermatofytóz byly použity např. FastPrep FP120 Instrument (Thermo Savant, Boston, USA) [16], MagNA Pure Compact Nucleic Acid Extraction Instrument (Roche, Indianapolis, USA) [7], Biorobot EZ1 (Qiagen, Hilden, Německo), nebo QIAsymphony (Qiagen) [3].
V případě identifikace a typizace houbových izolátů MGM probíhá úvodní kultivace nejčastěji na Sabouraudově agaru (SAB) s přídavkem chloramfenikolu a cykloheximidu. Hlavním požadavkem je čistota a stáří kultury (ideálně do 14 dnů). Izolaci DNA lze provádět podle známých protokolů [26, 27, 62], existuje však i velké množství komerčních souprav. K tomuto účelu byly úspěšně použity např. Fast DNA kit (Bio101; Lajolla, USA) [16], ArchivePure DNA Yeast + Gram-+ kit (5PRIME; Gaithersburg, USA) [39, 40], nebo Microbial DNA Isolation Kit (Mo-Bio Laboratories; Carlsbad, USA). Pomocí posledních dvou jmenovaných produktů je izolována DNA z dermatofytů, ale i jiných vláknitých hub také na našem pracovišti [38, 37].
Výhledově se předpokládá, že budou vypracovány doporučené standardizované postupy pro izolaci DNA dermatofytů z kultur i klinických vzorků, což je nutné pro sjednocení a porovnávání metod.
METODY DETEKCE A IDENTIFIKACE DERMATOFYTŮ
Přímý průkaz DNA v klinických vzorcích
Tato skupina metod je založená vesměs na principu PCR a při vhodné optimalizaci může být využita i ke druhové identifikaci dermatofytů. Výhodou je zejména vysoká citlivost (tab. 1) a rychlost detekce [7, 65], která se podle zvoleného postupu pohybuje od několika hodin po 2–3 dny.
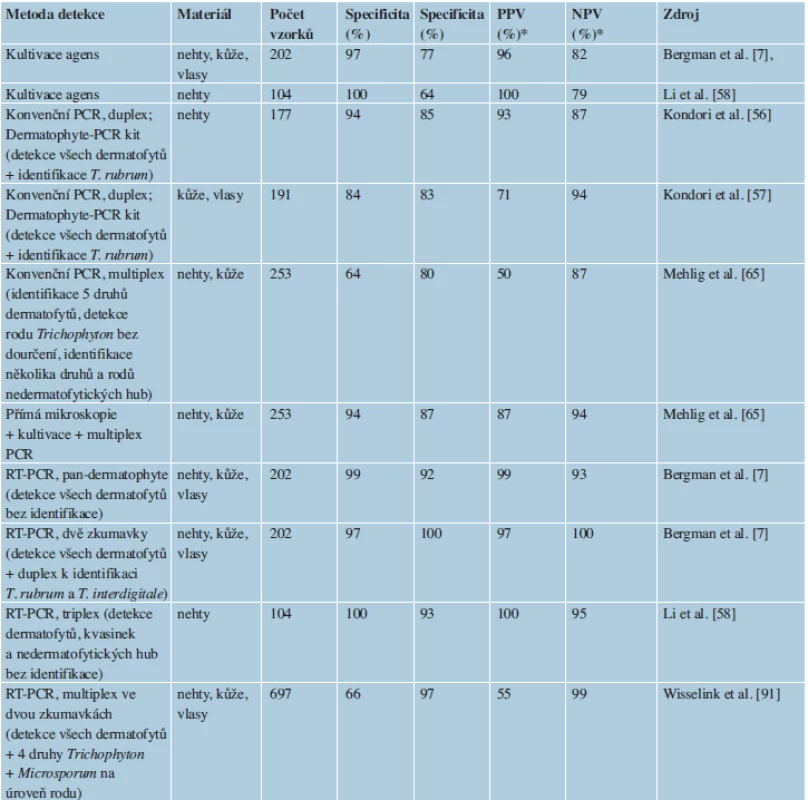
K průkazu DNA kožních houbových patogenů a odlišení od DNA jiných eukaryot a prokaryot bylo navrženo několik sad specifických primerů, které jsou cílené na různé oblasti DNA. Metody, které dovolují pouze detekci dermatofytů bez další specifikace (pan-dermatophyte PCR), jsou zaměřeny na konzervativní úseky DNA, jako je např. gen pro chitin syntázu 1 (CHS1) [48]. Většina dnes používaných metod, které si dávají za úkol nejen dermatofyty detekovat, ale i identifikovat hlavní druhy, jsou zacíleny na oblast ribozomální DNA (obsahuje oblasti 18S, ITS1, 5,8S, ITS2 a 28S), zejména na ITS oblast, která poskytuje dostatečné rozlišení mezi druhy dermatofytů. Výjimkami jsou metody užívající jako marker gen pro topoizomerázu II (TOP II) [6] nebo oblast mikrosatelitu [10]. Jde obvykle o úseky DNA, které jsou přítomné v genomu v mnoha kopiích a k úspěšné detekci je tedy potřeba menší množství izolované DNA, čímž se zvyšuje citlivost.
Metody detekce v klinických vzorcích jsou v současnosti omezeny buď pouze na průkaz dermatofytů jako skupiny bez dalšího určení, nebo dovolují identifikaci pouze nejběžnějších druhů. Dají se použít i v mnohanásobném uspořádání (multiplex), kdy je v jediné reakci přítomno několik sad primerů. Takové uspořádání umožní zvýšit specificitu reakce, nebo dovoluje identifikovat několik druhů dermatofytů za použití jediné reakce. Zajímavá je v tomto směru kombinace metody nested PCR se sekvenováním [88], která může zajistit velmi přesnou identifikaci na úrovni druhu. Sekvenování přímo navazující na PCR reakci provedenou v materiálu z klinického vzorku totiž většinou není možné, protože namnožený produkt, s výjimkou nested PCR, nebývá v dostatečné koncentraci [88]. Riziko laboratorní kontaminace sporami dermatofytů je velmi nízké v porovnání např. s rody Aspergillus, Penicillium, Cladosporium či Alternaria.
Výhodou konvenční (klasické) PCR je jednoduchost provedení a nízká cena i ve srovnání s běžnými kultivačními metodami [33]. Nevýhodou oproti RT-PCR je nutnost nanést produkty před vyhodnocením na gelovou elektroforézu. Metody založené na klasické PCR detekují nejčastěji všechny 3 rody dermatofytů jako celek bez přesnějšího určení, nebo nabízejí multiplex uspořádání s dourčením hlavních druhů. Součástí je téměř vždy dvojice primerů pro účely detekce všech druhů dermatofytů bez identifikace [11–13, 55, 56, 59]. Vyhodnocení probíhá na základě přítomnosti produktu na elektroforetogramu. Multiplex uspořádání u těchto metod většinou přináší možnost přímo určit, že původcem dermatofytózy je Trichophyton rubrum [12, 13, 56], případně jiné druhy [11]. Odlišně navržené metody dokáží specificky identifikovat např. T. rubrum/T. violaceum, ale bez detekce ostatních dermatofytů [10]. Metoda publikovaná kolektivem Kim et al. [55] dovoluje detekci všech dermatofytů a identifikaci na úroveň rodu. Metoda autorů Li et al. [58] je schopná detekovat dermatofyty jako celek a v dalších dvou samostatných reakcích skupinu kvasinek a jakoukoliv houbu. Nejkomplexnější v tomto ohledu je komerční souprava Mentype Mycoderm PCR Amplification kit (Biotype Diagnostic GmbH, Moritzburger, Německo), která je schopná identifikovat ve dvou oddělených PCR T. rubrum, T. interdigitale, Microsporum canis, M. gypseum a Epidermophyton floccosum, dále ještě rod Trichophyton jako celek a několik důležitých nedermatofytických hub [65].
Nested PCR pracuje se systémem tzv. vnějších a vnitřních primerů. V prvním kole PCR, kdy se množí delší fragment DNA, je použita dvojice vnějších primerů. V této fázi mohou vznikat nespecifické produkty. Ve druhém kole se množí kratší fragment uvnitř amplikonu z první PCR pomocí vnitřních primerů. Pokud se v prvním kole amplifikovalo více nespecifických produktů, je pravděpodobné, že při druhé PCR bude specificky namnožen jen jediný produkt odpovídající oblasti uvnitř amplikonu z prvního kola. Podle nastavení metody se může jednat o detekci dermatofytů bez detailnějšího určení (vzorky z nehtů, CHS1, Litz et al. [60]; vzorky z nehtů, CHS1, Garg et al. [24]; kůže a vlasy, velká podjednotka genu pro ribozomální RNA = LSU rDNA, Verrier et al. [88]), nebo se dá produkt první reakce použít pro jednu, či více následných PCR specifických jen pro určitý druh (parafinový řez, vnitřní přepisovaný mezerník genu pro ribozomální RNA = ITS rDNA, detekce T. rubrum, Nagao et al. [71]). V mnohanásobném (multiplex) uspořádání je při druhé PCR do směsi přidáno několik sad primerů specifických pro určité druhy. Jsou navrženy tak, aby výsledné amplikony měly pro různé druhy odlišnou délku a identifikace je tak možná na základě velikosti produktu na elektroforetogramu. Amplikon je také možné podrobit sekvenační reakci za použití primerů z druhé reakce. Přesnost identifikace na základě získané sekvence DNA potom závisí na oblasti, která byla namnožena a na délce produktu. Ebihara et al. [19] navrhli postup pro časnou diagnostiku hub působících onychomykózu, v prvním kroku byla nespecificky namnožena oblast LSU rDNA pro jakoukoliv houbu, v druhé následné PCR mohly být použity primery specifické pro všechny dermatofyty, T. rubrum, T. mentagrophytes a některé nedermatofytické rody hub. Jako seminested PCR je označován postup, kdy je při druhé PCR použit jeden z dvojice primerů z prvního kola. Tímto postupem byl detekován T. rubrum z nehtů. Metoda byla vyvinuta jak pro aktin, tak pro ITS rDNA [33].
Stále více se dnes v diagnostice dermatofytóz využívá PCR v reálném čase (RT-PCR), většinou opět v multiplex uspořádání, nebo s využitím značených DNA sond, které umožňují současnou detekci několika druhů. Metodicky se v podstatě jedná o klasickou PCR, ale speciální termocykler umožňuje průběžně monitorovat přírůstky DNA během každého cyklu. To je umožněno díky fluorescenčnímu substrátu, který se váže na DNA a intenzita fluorescence pak odráží množství nově syntetizované DNA. Produkty amplifikace téhož genu mají u různých druhů různou sekvenci, a tedy i teplotu tání. Produkty pak můžeme rozlišit pomocí odlišných křivek tání a využít tuto informaci k druhové identifikaci nebo odhalení nespecifických produktů amplifikace.
Výhodou metody je vysoká specificita a citlivost. Identifikace nejčastějších druhů dermatofytů může být provedena během několika hodin [12, 13, 55]. Metoda je výhodná i tím, že celý proces je automatizovaný v jediné uzavřené zkumavce, což minimalizuje rizika kontaminace.
V posledních letech bylo publikováno několik postupů založených na RT-PCR a umožňujících detekci různého počtu druhů dermatofytů. První kvalitní práce z roku 2007 byla zaměřená na oblast ITS a umožňovala rozlišit několik základních druhů pomocí značených sond. Práce posloužila jako základ pro další studie a je z dnešního pohledu již překonaná. Alexander et al. [3] použili detekci T. rubrum pomocí RT-PCR jako počáteční krok v diagnostice onychomykózy v kombinaci s klasickou diagnostikou pomocí přímé mikroskopie a kultivace. Jako nejefektivnější postup bylo doporučeno, aby byly kultivovány pouze PCR-negativní a mikroskopicky pozitivní vzorky. Metoda vyvinutá Bergmansem et al. [9] je naopak schopná identifikovat až 11 klinicky významných druhů v jediné multiplex reakci za pomoci systému značených sond a analýzy křivek tání produktů. Žádná z dosud uvedených metod neměla, na rozdíl od následujících, publikovaných autory Wisselink et al. [91], Bergman et al. [7], Miyajima et al. [66], jako svou součást možnost detekce celého druhového spektra dermatofytů, což je zásadní pro průkaz některých málo běžných nebo nepopsaných druhů. Kromě toho jsou všechny tři metodiky schopné identifikovat i několik hlavních druhů dermatofytů; nevýhodou prvních dvou studií [7, 91] je průběh ve dvou oddělených reakcích. Někteří zmínění autoři uvádějí [9, 91], že jejich RT-PCR metoda je schopná zcela nahradit klasické diagnostické metody.
V případě reverzní hybridizace v návaznosti na PCR (PCR-RLB) je v prvním kroku provedena PCR s primery nasedajícími na ITS oblast genomu dermatofytů, přičemž je jeden z primerů značený. Produkty amplifikace jsou následně hybridizovány na membráně se specifickými značenými oligonukleotidovými DNA-sondami. Uvedená metoda byla vyvinuta pro detekci a identifikaci devíti nejběžnějších druhů dermatofytů ze vzorků nehtů, kůže a vlasů [8]. Je poměrně pracná, navíc výsledky mohou být obtížně interpretovatelné u vzorků s malým výchozím množstvím DNA. Výsledky je však možné vyhodnotit již následující den po odběru.
Pro detekci a identifikaci pěti druhů dermatofytů (T. rubrum, T. interdigitale, T. violaceum, M. canis a E. floccosum) byla vyvinuta metoda PCR-ELISA [6]. Začíná pěti separátními PCR reakcemi se značenými, druhově specifickými primery. Amplikony poté hybridizují s komplementárními, druhově specifickými sondami značenými biotinem a zacílenými na TOP II. Produkt hybridizace je vizualizován na mikrotitrační destičce pokryté streptavidinem. Tuto metodu využívá komerční kit Onychodiag (Bio Advance, Francie), který umožňuje identifikaci výše zmíněných druhů v klinických vzorcích během 24 hodin. Metoda je údajně až desetkrát citlivější než konvenční PCR. Hlavními nevýhodami jsou pracnost, riziko kontaminace a riziko zkřížené reaktivity sond.
Některé diagnostické sady mikrodestiček (microarrays) jsou schopné identifikovat i dermatofyty. Souprava vyvinutá autory Sato et al. [81] je založená na amplifikaci ITS rDNA všech mikromycet a následné hybridizaci produktu s prefabrikovanými specifickými DNA sondami navázanými na destičce. Ta obsahuje celkem 26 druhově specifických sond a 3 rodově specifické, dále políčko cílené na všechny mikromycety a jen na vláknité houby. Mezi nevýhody uvedené metody je nutno uvést možnou detekci kožních komenzálů, případně i zkříženou reaktivitu sond.
Identifikace kultur
Metody pro určování kultur hub je možné podle typu výsledku rozdělit na sekvenační, fingerprintové a jiné (viz obr. 1).
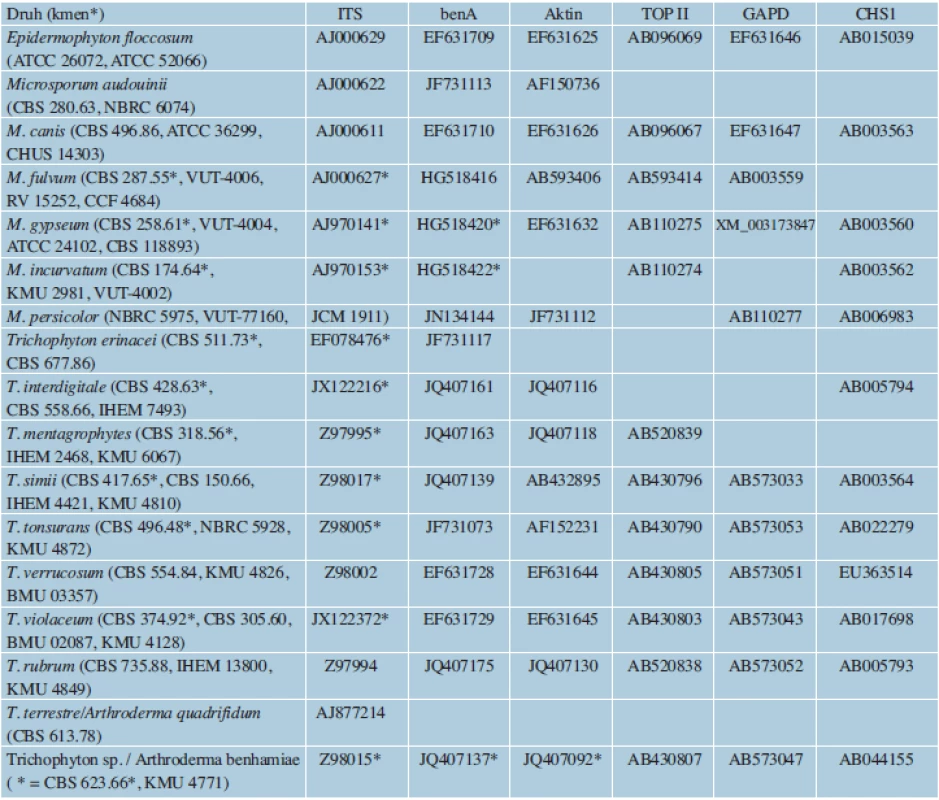
Sekvenování cílových genů zajišťuje při správném výběru oblasti naprosto přesnou druhovou identifikaci. Hlavní nevýhodou je stále vyšší časová a finanční náročnost, zejména při zpracování většího počtu vzorků. Přesto podle některých výpočtů [49] není sekvenování např. ITS oblasti rDNA o mnoho dražší, než je druhová identifikace za použití klasických metod. U dermatofytů bývají nejčastěji vybírány úseky ITS1, 5,8S a ITS2 rDNA, které jsou známy pro všechny druhy dermatofytů a poskytují až na výjimky dostatečné rozlišení. Dalšími možnými oblastmi jsou geny pro ß-tubulin (benA), aktin (ACT), mangan-dependentní superoxiddismutázu (MnSOD), translační elongační faktor 1-α (TEF1-α) a glyceraldehyd-3-fosfát dehydrogenázu (GAPD), TOP II a CHS1 [5, 22, 39, 40, 47, 52, 75, 78]. Přístupová čísla k sekvencím, které pocházející z věrohodně určených kmenů běžných druhů dermatofytů a jsou uložené v databázi GenBank, jsou uvedena v tabulce 2. K rutinnímu určování je obvykle dostačující oblast ITS rDNA [41, 73, 87], sekvence genů kódujících proteiny jsou využívány především ve fylogenetických studiích [5, 52]. ITS je zatím jediný sekvenční marker zastoupený pro všechny v současnosti akceptované druhy v databázi GenBank [32]. Nevýhodou databáze je ale velké množství chybně identifikovaných izolátů [32, 85]. Z tohoto důvodu byla vytvořena a je spravovaná databáze sekvencí ITS rDNA dermatofytů, která zahrnuje jen sekvence patřící správně určeným kmenům (http://www.cls.knaw.nl/dermatofytes).
Fingerprintové metody jsou rychlé, většinou jednoduché a levné. Jsou vhodné zejména při velkém počtu analyzovaných vzorků. Naopak nevýhodou některých z nich jsou nároky na kvalitu DNA, která nesmí být kontaminována [46, 64]. Aby byly výsledky inter-, ale i intralaboratorně porovnatelné, je nutné u nejcitlivějších metod vždy dodržovat stejnou metodiku izolace DNA a při PCR užívat stejný termocykler a chemikálie od stejných výrobců. Problémem limitujícím využití metod pro fylogenetické účely bývá možná komigrace fragmentů o stejné délce, ale odlišné sekvenci, a tedy nereprezentujících homologní úseky DNA. Mezi fingerprintové metody použité u dermatofytů patří náhodná amplifikace polymorfní DNA (RAPD), polymorfismus sekvencí mezi dvěma mikrosatelity (ISSR-PCR) a polymorfismus délky amplifikovaných fragmentů (AFLP).
RAPD je charakteristická použitím jednoho, či více krátkých primerů. V PCR je navíc použita nízká teplota pro jejich nasedání, což způsobuje, že primery nemusí nasedat zcela komplementárně na DNA a výsledný fingerprint je pak bohatší. Metoda je velmi citlivá na změny reakčních podmínek. U dermatofytů byla úspěšně použita ke druhové identifikaci [63, 61, 67]. Někdy se od RAPD odděluje principiálně velmi obdobná metoda zvaná náhodná PCR (APPCR) [61], která užívá primery o delší sekvenci.
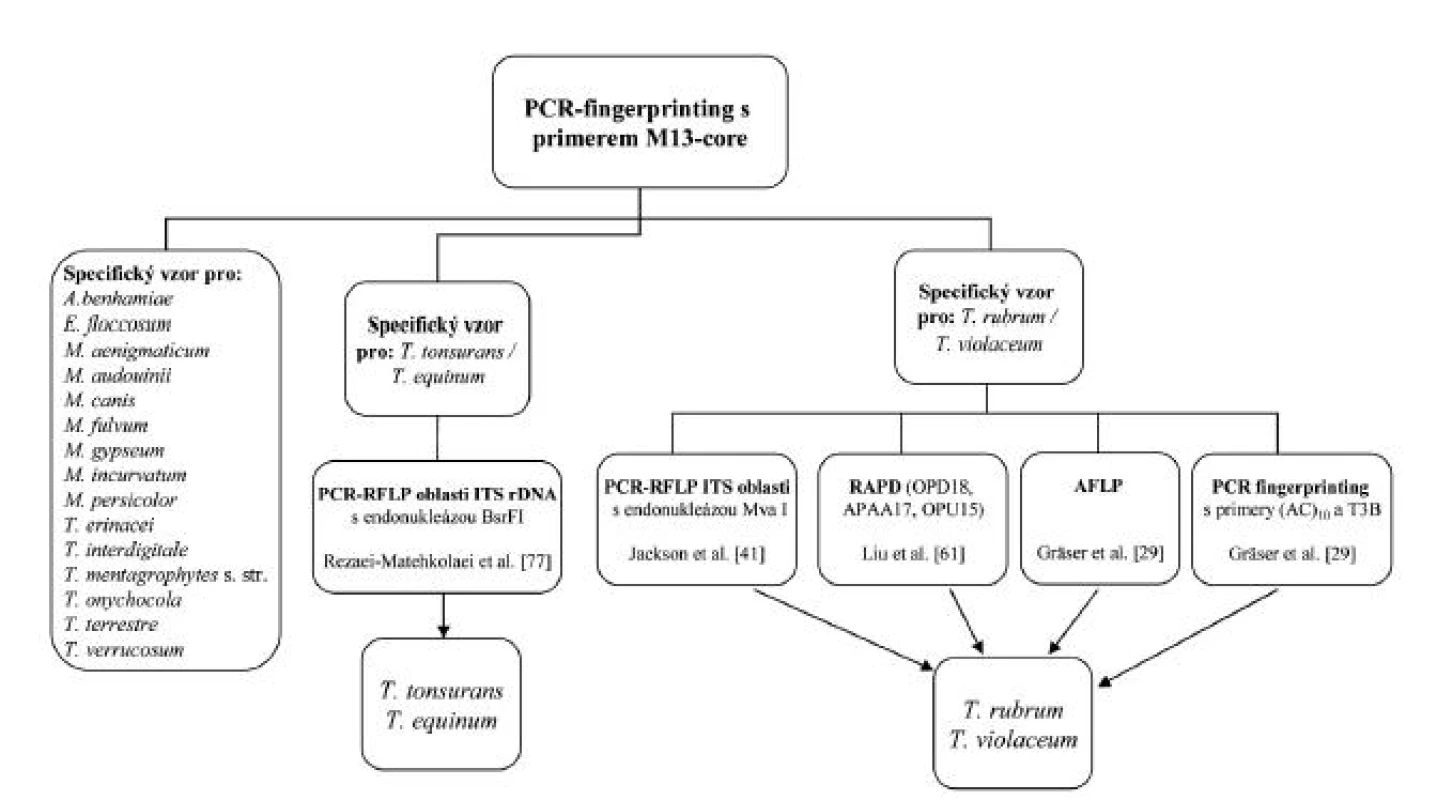
ISSR-PCR využívá primery v podobě repetitivních sekvencí komplementárních k tzv. mini - a mikrosatelitním lokusům. Primer mívá často i krátkou tzv. kotvící sekvenci, která přesahuje okraj lokusu, čímž se sníží počet mikrosatelitů, na které je primer schopný nasedat, a tedy i počet produktů. Výsledné fingerprinty vykazují často vysokou komplexnost a na rozdíl od RAPD má metoda i vysokou reprodukovatelnost [20]. Využitím mikrosatelitního primeru (GACA)4 byly rozpoznány běžné druhy dermatofytů [20, 84]. Navíc byl ve studii použit druhý primer (GTG)5, který úspěšně odlišoval i fylogeneticky velmi blízké druhy T. interdigitale („T. mentagrophytes“) a T. tonsurans [84]. Na našem pracovišti se velmi osvědčila metoda ISSR-PCR s primerem M13-core, který je schopný dobře rozlišit nejdůležitější druhy dermatofytů vyskytující se na našem území (obr. 2) a byl úspěšně použit u zmíněných houbových agens i v minulosti [26].
Metoda AFLP byla u dermatofytů použita jen v omezené míře pro taxonomické účely [29, 28]. Ačkoliv je dobře reprodukovatelný a podává odpovídající představu o polymorfismu srovnávaných organismů na druhové a nižší úrovni, je jeho použití pro identifikaci nepraktické kvůli vyšší časové i finanční náročnosti.
Polymorfismus délky restrikčních fragmentů (PCR--RFLP) je založen na klasické PCR, kdy je v prvním kroku amplifikován specifickými primery určitý úsek DNA, který je následně štěpen restrikčními enzymy (endonukleázami). K namnožení DNA se používají podobné oblasti DNA jako při sekvenování, např. úseky ITS nebo NTS rDNA (Non-Transcribed Spacer; nepřepisovaný mezerník) [35, 41, 79], geny TOP II [34, 45] nebo benA [1]. Dnes se metoda častěji používá k rozlišení blízce příbuzných druhů uvnitř druhových komplexů, které jsou obtížně rozlišitelné jinými MGM [77, 78]. Při určitém zjednodušení je pak postup navržen tak, že je v sekvencích dvou blízkých druhů nalezeno variabilní místo, které u jednoho z nich specificky rozeznává a štěpí určitá endonukleáza. Ta je pak použita ke štěpení amplifikovaného úseku DNA. Na elektroforetogramu se pak u prvního druhu vyskytuje pouze jeden produkt a u druhého dva. Výhodou je, že se tím lze vyhnout ekonomicky náročnějšímu sekvenování, metoda je navíc dobře reprodukovatelná. Podobně finančně výhodné může být při velkém počtu izolátů použití několika restrikčních endonukleáz na jediný amplifikovaný úsek DNA. Většina kultur může být určena podle specifického fingerprintu po prvním restrikčním štěpení, zbylé druhy jsou určeny jen do druhových komplexů a mohou být identifikovány štěpením stejného úseku za použití jiné restriktázy [80]. Velmi blízce příbuzné druhy T. tonsurans a T. equinum nebo druhy M. canis, M. ferrugineum a M. audouinii se dají odlišit restrikčním štěpením genů TEF-1α, benA a oblasti ITS rDNA pomocí různých endonukleáz [77, 78, 80].
RT-PCR je použitelná nejen v detekci dermatofytů, ale i v identifikaci navazující na jejich kultivaci. Taková metodika byla popsána pro 10 druhů dermatofytů, včetně některých vzácně izolovaných [95]. Přínosem studie je především možnost obohatit současné protokoly RT-PCR pro detekci DNA přímo z klinických vzorků o vzácné druhy. Potenciál metody není zatím pro běžné určování druhů velký, protože existuje řada levnějších a jednodušších identifikačních metod, které pokrývají větší množství druhů.
Typizace izolátů
Typizační metody mohou pomoci lépe porozumět epidemiologii, populační biologii a patogenezi dermatofytóz. Jsou využívány při identifikaci endemických kmenů, pátrání po zdroji infekce v rodině, veřejném zařízení, u zvířat, dále také při zjišťování, zda je infekce způsobena jedním, či více kmeny. Je možné rovněž stanovit, které kmeny jsou virulentnější, mají vyšší rezistenci k antimykotikům nebo způsobují specifický klinický obraz infekce. U chronických dermatofytóz může být ověřeno, zda se jedná o recidivu nebo reinfekci. Dále lze získat informaci o distribuci jednotlivých genotypů v populaci, což může odhalit změny v závislosti na převládající léčbě.
Mikrosatelity jsou hypervariabilní DNA sekvence složené z motivů krátkých tandemových repetic, které se v genomech všech doposud studovaných eukaryotických organismů vyskytují s poměrně vysokou frekvencí [59]. Mikrosatelity mají mnohem vyšší mutační rychlost (10-2-10-5 mutací na nukleotidový pár na generaci) oproti frekvenci 10-9, která se uvádí pro bodové mutace v nekódujících oblastech, jako je např. ITS oblast [74]. Tato nestálost vytváří vysokou vnitrodruhovou variabilitu. Mikrosatelity jsou z tohoto důvodu u hub, ale i v jiných říších organismů, používány především v populačních studiích [30]. Aplikace mikrosatelitních markerů na typizaci druhů ukazuje, že některé druhy antropofilních a zoofilní dermatofytů jsou teprve na začátku svého evolučního vývoje a obsahují malé množství polymorfismů [30]. Výhodou u skupiny dermatofytů je fakt, že je známý genom několika druhů, což proces hledání mikrosatelitů usnadňuje. Přesto jsou dostupné primery jen k několika variabilním mikrosatelitům u malého počtu klinicky významných druhů (viz tab. 3), těmi jsou T. rubrum [31, 74], M. canis [82] a M. persicolor [83].
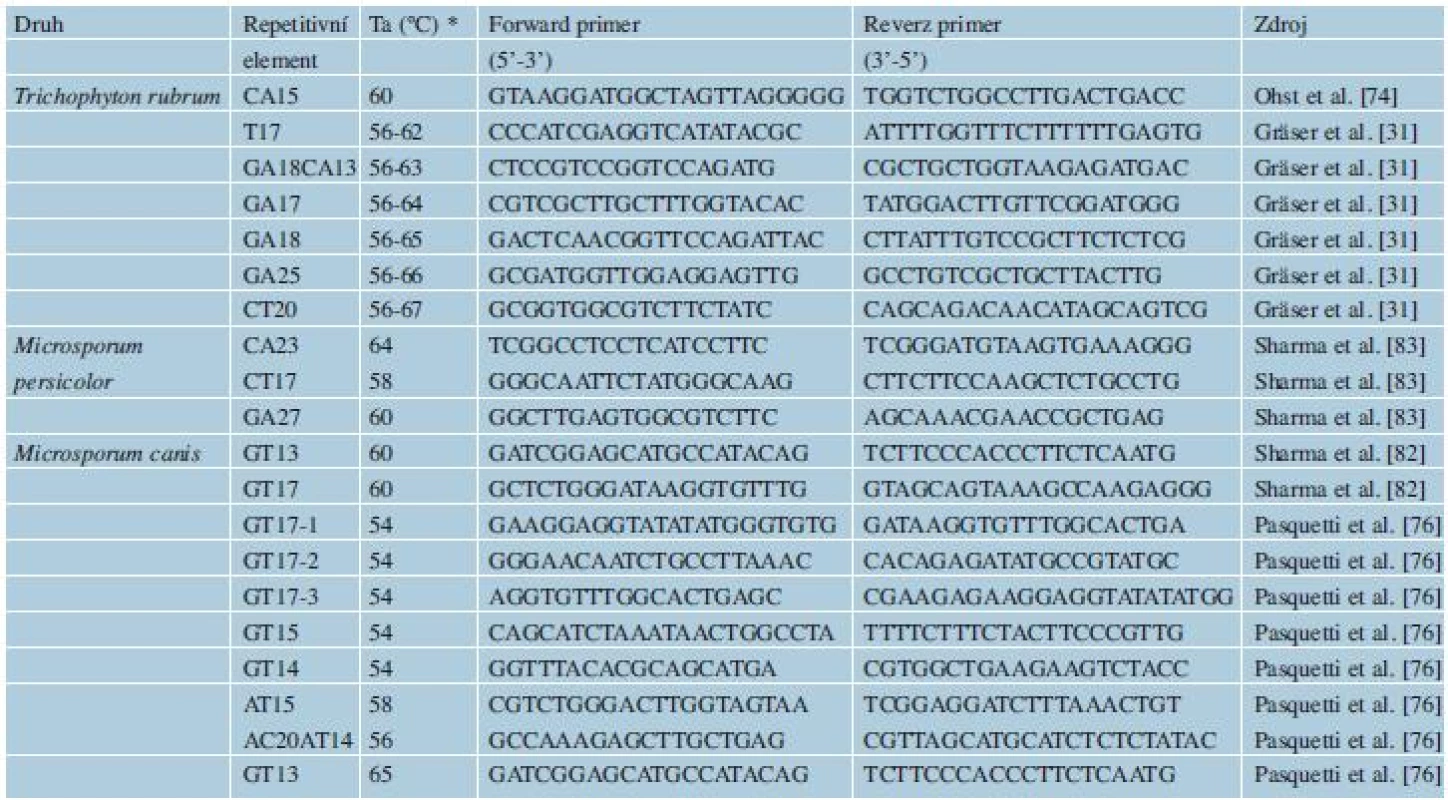
Data získaná analýzou mikrosatelitů jsou užitečná nejen pro epidemiologii, ale přináší i cenné informace z hlediska evoluce a biologie druhů. Analýza mikrosatelitů například podpořila status samostatného druhu pro M. audouinii a M. ferrugineum uvnitř komplexu M. canis [50], další druhy a variety byly synonymizovány s těmito třemi druhy. Jiná studie ukázala, že za většinu lidských infekcí působených M. canis zodpovídá jediný, více virulentní zvířecí genotyp, který se s vyšší úspěšností přenáší na člověka [82]. Populační studie na druhu T. rubrum potvrdily jeho nízkou vnitrodruhovou variabilitu a oprávněnost synonimizace řady dříve uznávaných druhů s T. rubrum. Jediné druhové jméno, které ve skutečnosti reprezentuje jen jakousi částečně izolovanou a geneticky odlišnou populaci, je T. soudanense [31, 74].
Multilokusová sekvenační typizace (MLST) vykazuje při výběru vhodných genů ve srovnání se sekvenováním jednoho PCR produktu vysokou vnitrodruhovou diskriminační schopnost. Problémem je nutnost velmi kvalitních sekvencí (sekvenační chyby vytváří falešnou variabilitu) a vysoká finanční náročnost. Na druhou stranu tkví velká výhoda MLST v možnosti dobrého sdílení výsledků mezi laboratořemi a možnost využít a revidovat data kdykoliv v budoucnu. V rámci skupiny dermatofytů MLST schéma zatím pro populační studie využito nebylo. Z dosud publikovaných molekulárních dat je patrné, že sekvenované lokusy (ACT, TOP II, benA, ITS rDNA a GPD) mají poměrně nízkou diskriminační sílu pro klonální druhy jako T. rubrum a T. verrucosum, ale naopak rozlišily řadu haplotypů uvnitř druhu Arthroderma benhamiae [5, 52, 51]. Využití metody jako možné typizační techniky u dermatofytů tedy nic nebrání a patří jí spolu s mikrosatelity budoucnost v typizaci dermatofytů.
Fingerprintové metody typu RAPD dokážou s různou mírou úspěšnosti odlišit kmeny některých druhů [26, 61, 96]. Nízká reprodukovatelnost výsledků a nezřídka i nedostatečná variabilita odsouvají do značné míry tyto metody ze zájmu populačních studií u hub, kde se prosazuje především analýza mikrosatelitů. V případě dermatofytů ale metoda RAPD zůstává nejběžnější metodou typizace kmenů a stále vzniká řada typizačních schémat na ní založených. Metoda byla s různou mírou úspěchu (žádné vnitrodruhové rozlišení až po rozlišení téměř všech zkoumaných izolátů) aplikována na řadu druhů. Nejčastějším cílem typizace jsou kmeny T. rubrum [4, 35, 36, 92, 93], dále pak T. interdigitale [44, 54], M. canis [18, 84, 94] a E. floccosum [53]. Dobré rozlišení izolátů T. rubrum nabízí metodika polských autorů [36], která kombinovala dva primery a úspěšně rozlišila 47 různých genotypů mezi 55 izoláty. U T. interdigitale („T. mentagrophytes“ podle starších identifikačních schémat) se pomocí RAPD daří například odlišit lidské izoláty působící převážně onychomykózu a tinea pedis od původem zvířecích izolátů, které u člověka působí hlavně tinea corporis [54].
PCR-RFLP až na výjimky nepředstavuje nástroj příliš vhodný k posouzení vnitrodruhové variability. Výjimkou je NTS oblast rDNA, která se u dermatofytů ukázala jako velmi variabilní, a proto je používaná i v diskriminaci jednotlivých izolátů u druhů T. rubrum [17, 92], T. interdigitale [69, 89], A. benhamiae [68, 86] a T. tonsurans [2, 23, 70]. Izoláty jinak geneticky homogenního druhu T. rubrum je také možné rozlišit na základě počtu kopií dvou tandemově se opakujících prvků označovaných jako TRS-1 a TRS-2 (Tandemly Repetitive Subelements) [42]. Hlavní předností RAPD a PCR-RFLP je jednoduchost a nízká finanční náročnost ve srovnání s analýzou mikrosatelitů a MLST, které ale dávají lépe interpretovatelné výsledky.
Poděkování: Tato práce byla podpořena projekty GAUK 1344214 a IGA_LF_2014_021.
Do redakce došlo dne 8. 7. 2014.
Adresa pro korespondenci:
Mgr. Vít Hubka
Katedra botaniky, Přírodovědecká fakulta,
Univerzita Karlova v Praze
Benátská 2
128 01 Praha 2
e-mail: hubka@biomed.cas.cz
Sources
1. ABASTABAR, M., MIRHENDI, H., REZAEI–MATEHKOLAEI, A., et al. Restriction analysis of ß–tubulin gene for differentiation of the common pathogenic dermatophytes. J. Clin. Lab. Anal., 2014, 28, p. 91–96.
2. ABDEL-RAHMAN, S. M., SUGITA, T., GONZÁLEZ, G. M., et al. Divergence among an international population of Trichophyton tonsurans isolates. Mycopathologia, 2010, 169, p. 1–13.
3. ALEXANDER, C., SHANKLAND, G., CARMAN, W., WILLIAMS, C. Introduction of a dermatophyte polymerase chain reaction assay to the diagnostic mycology service in Scotland. Brit. J. Dermatol., 2011, 164, p. 966–972.
4. BAEZA, L. C., MATSUMOTO, M. T., ALMEIDA, A. M. F., MENDES-GIANNINI, M. J. S. Strain differentiation of Trichophyton rubrum by randomly amplified polymorphic DNA and analysis of rDNA nontranscribed spacer. J. Med. Microbiol., 2006, 55, p. 429–436.
5. BEGUIN, H., PYCK, N., HENDRICKX, M., et al. The taxonomic status of Trichophyton quinckeanum and T. interdigitale revisited: a multigene phylogenetic approach. Med. Mycol., 2012, 50, p. 871–882.
6. BEIFUSS, B., BEZOLD, G., GOTTLOBER, P., et al. Direct detection of five common dermatophyte species in clinical samples using a rapid and sensitive 24-h PCR-ELISA technique open to protocol transfer. Mycoses, 2011, 54, p. 137–145.
7. BERGMAN, A., HEIMER, D., KONDORI, N., ENROTH, H. Fast and specific dermatophyte detection by automated DNA extraction and real – time PCR. Clin. Microbiol. Infect., 2013, 19, p. E205–E211.
8. BERGMANS, A., SCHOULS, L., VAN DER ENT, M., et al. Validation of PCR–reverse line blot, a method for rapid detection and identification of nine dermatophyte species in nail, skin and hair samples. Clin. Microbiol. Infect., 2008, 14, p. 778–788.
9. BERGMANS, A., VAN DER ENT, M., KLAASSEN, A., et al. Evaluation of a single–tube real–time PCR for detection and identification of 11 dermatophyte species in clinical material. Clin. Microbiol. Infect., 2010, 16, p. 704–710.
10. BRASCH, J., BECK – JENDROSCHEK, V., GLÄSER, R. Fast and sensitive detection of Trichophyton rubrum in superficial tinea and onychomycosis by use of a direct polymerase chain reaction assay. Mycoses, 2011, 54, p. e313–e317.
11. BRILLOWSKA–DAABROWSKA, A., SWIERKOWSKA, A., LINDHARDT SAUNTE, D. M., ARENDRUP, M. C. Diagnostic PCR tests for Microsporum audouinii, M. canis and Trichophyton infections. Med. Mycol., 2010, 48, p. 486–490.
12. BRILLOWSKA–DABROWSKA, A., NIELSEN, S. S., NIELSEN, H. V., ARENDRUP, M. C. Optimized 5-hour multiplex PCR test for the detection of tinea unguium: performance in a routine PCR laboratory. Med. Mycol., 2010, 48, p. 828–831.
13. BRILLOWSKA–DĄBROWSKA, A., SAUNTE, D. M., ARENDRUP, M. C. Five-hour diagnosis of dermatophyte nail infections with specific detection of Trichophyton rubrum. J. Clin. Microbiol., 2007, 45, p. 1200–1204.
14. CAFARCHIA, C., GASSER, R. B., FIGUEREDO, L. A., et al. An improved molecular diagnostic assay for canine and feline dermatophytosis. Med. Mycol., 2013, 51, p. 136–143.
15. CAFARCHIA, C., IATTA, R., LATROFA, M. S., GRÄSER, Y., OTRANTO, D. Molecular epidemiology, phylogeny and evolution of dermatophytes. Infect. Genet. Evol., 2013, 20, p. 336–351.
16. CANO, J., REZUSTA, A., SOLE, M., et al. Inter-single-sequence-repeat-PCR typing as a new tool for identification of Microsporum canis strains. J. Dermatol. Sci., 2005, 39, p. 17–21.
17. DE ASSIS SANTOS, D., DE CARVALHO ARAUJO, R. A., KOHLER, L. M., et al. Molecular typing and antifungal susceptibility of Trichophyton rubrum isolates from patients with onychomycosis pre - and post-treatment. Int. J. Antimicrob. Agents, 2007, 29, p. 563–569.
18. DOBROWOLSKA, A., DEBSKA, J., KOZŁOWSKA, M., STĄCZEK, P. Strains differentiation of Microsporum canis by RAPD analysis using (GACA)4 and (ACA)5 primers. Pol. J. Microbiol., 2011, 60, p. 145–148.
19. EBIHARA, M., MAKIMURA, K., SATO, K., ABE, S., TSUBOI, R. Molecular detection of dermatophytes and nondermatophytes in onychomycosis by nested polymerase chain reaction based on 28S ribosomal RNA gene sequences. Brit. J. Dermatol., 2009, 161, p. 1038–1044.
20. FAGGI, E., PINI, G., CAMPISI, E., et al. Application of PCR to distinguish common species of dermatophytes. J. Clin. Microbiol., 2001, 39, p. 3382–3385.
21. FARI, E., GRÄSER, Y. An epidemic of tinea corporis caused by Trichophyton tonsurans among children (wrestlers) in Germany. Mycoses, 2000, 43, p. 191–196.
22. FRÉALLE, E., RODRIGUE, M., GANTOIS, N., et al. Phylogenetic analysis of Trichophyton mentagrophytes human and animal isolates based on MnSOD and ITS sequence comparison. Microbiology, 2007, 153, p. 3466–3477.
23. GAEDIGK, A., GAEDIGK, R., ABDEL-RAHMAN, S. M. Genetic heterogeneity in the rRNA gene locus of Trichophyton tonsurans. J. Clin. Microbiol., 2003, 41, p. 5478–5487.
24. GARG, J., TILAK, R., SINGH, S., et al. Evaluation of pan-dermatophyte nested PCR in diagnosis of onychomycosis. J. Clin. Microbiol., 2007, 45, p. 3443–3445.
25. GARG, J., TILAK, R., GARG, A., et al. Rapid detection of dermatophytes from skin and hair. BMC Res. Notes, 2009, 2, p. 60.
26. GRÄSER, Y., EL FARI, M., PRESBER, W., STERRY, W., TIETZ, H. J. Identification of common dermatophytes (Trichophyton, Microsporum, Epidermophyton) using polymerase chain reactions. Brit. J. Dermatol., 1998, 138, p. 576–582.
27. GRÄSER, Y., EL FARI, M., VILGALYS, R., et al. Phylogeny and taxonomy of the family Arthrodermataceae (dermatophytes) using sequence analysis of the ribosomal ITS region. Med. Mycol., 1999, 37, p. 105–114.
28. GRÄSER, Y., KUIJPERS, A. F. A., PRESBER, W., DE HOOG, G. S. Molecular taxonomy of Trichophyton mentagrophytes and T. tonsurans. Med. Mycol., 1999, 37, p. 315–330.
29. GRÄSER, Y., KUIJPERS, A. F. A., PRESBER, W., DE HOOG, G. S. Molecular taxonomy of the Trichophyton rubrum complex. J. Clin. Microbiol., 2000, 38, p. 3329–3336.
30. GRÄSER, Y., DE HOOG, S., SUMMERBELL, R. Dermatophytes: recognizing species of clonal fungi. Med. Mycol., 2006, 44, p. 199–209.
31. GRÄSER, Y., FRÖHLICH, J., PRESBER, W., DE HOOG, S. Microsatellite markers reveal geographic population differentiation in Trichophyton rubrum. J. Med. Microbiol., 2007, 56, p. 1058–1065.
32. GRÄSER, Y., SCOTT, J., SUMMERBELL, R. The new species concept in dermatophytes – a polyphasic approach. Mycopathologia, 2008, 166, p. 239–256.
33. GUPTA, A., ZAMAN, M., SINGH, J. Fast and sensitive detection of Trichophyton rubrum DNA from the nail samples of patients with onychomycosis by a double–round polymerase chain reaction – based assay. Brit. J. Dermatol., 2007, 157, p. 698–703.
34. GUTZMER, R., MOMMERT, S., KÜTTLER, U., WERFEL, T., KAPP, A. Rapid identification and differentiation of fungal DNA in dermatological specimens by Light Cycler PCR. J. Med. Microbiol., 2004, 53, p. 1207–1214.
35. HRYNCEWICZ–GWOZDZ, A., JAGIELSKI, T., DOBROWOLSKA, A., SZEPIETOWSKI, J. C., BARAN, E. Identification and differentiation of Trichophyton rubrum clinical isolates using PCR-RFLP and RAPD methods. Eur. J. Clin. Microbiol. Infect. Dis., 2011, 30, p. 727–731.
36. HRYNCEWICZ–GWOZDZ, A., JAGIELSKI, T., SADAKIERSKA-CHUDY, A., et al. Molecular typing of Trichophyton rubrum clinical isolates from Poland. Mycoses, 2011, 54, p. e726–736.
37. HUBKA, V., KUBATOVA, A., MALLATOVA, N., et al. Rare and new aetiological agents revealed among 178 clinical Aspergillus strains obtained from Czech patients and characterised by molecular sequencing. Med. Mycol., 2012, 50, p. 601–610.
38. HUBKA, V., KOLAŘÍK, M., KUBÁTOVÁ, A., PETERSON, S. W. Taxonomical revision of the genus Eurotium and transfer of species to Aspergillus. Mycologia, 2013, 105, p. 912–937.
39. HUBKA, V., CMOKOVA, A., SKOREPOVA, M., MIKULA, P., KOLARIK, M. Trichophyton onychocola sp. nov. isolated from human nail. Med. Mycol., 2014, 52, p. 285–292.
40. HUBKA, V., DOBIASOVA, S., DOBIAS, R., KOLARIK, M. Microsporum aenigmaticum sp. nov. from M. gypseum complex, isolated as a cause of tinea corporis. Med. Mycol., 2014, 52, p. 387–396.
41. JACKSON, C. J., BARTON, R. C., EVANS, E. G. V. Species identification and strain differentiation of dermatophyte fungi by analysis of ribosomal-DNA intergenic spacer regions. J. Clin. Microbiol., 1999, 37, p. 931–936.
42. JACKSON, C. J., BARTON, R. C., KELLY, S. L., EVANS, E. G. V. Strain identification of Trichophyton rubrum by specific amplification of subrepeat elements in the ribosomal DNA nontranscribed spacer. J. Clin. Microbiol., 2000, 38, p. 4527–4534.
43. JENSEN, R. H., ARENDRUP, M. C. Molecular diagnosis of dermatophyte infections. Curr. Opin. Infect. Dis., 2012, 25, p. 126–134.
44. KAC, G., BOUGNOUX, M., FEUILHADE DE CHAUVIN, M., SENE, S., DEROUIN, F. Genetic diversity among Trichophyton mentagrophytes isolates using random amplified polymorphic DNA method. Brit. J. Dermatol., 1999, 140, p. 839–844.
45. KAMIYA, A., KIKUCHI, A., TOMITA, Y., KANBE, T. PCR and PCR–RFLP techniques targeting the DNA topoisomerase II gene for rapid clinical diagnosis of the etiologic agent of dermatophytosis. J. Dermatol. Sci., 2004, 34, p. 35–48.
46. KANBE, T., SUZUKI, Y., KAMIYA, A., et al. PCR-based identification of common dermatophyte species using primer sets specific for the DNA topoisomerase II genes. J. Dermatol. Sci., 2003, 32, p. 151–161.
47. KANO, R., NAKAMURA, Y., WATARI, T., et al. Molecular analysis of chitin synthase 1 (CHS1) gene sequences of Trichophyton mentagrophytes complex and T. rubrum. Curr. Microbiol., 1998, 37, p. 236–239.
48. KANO, R., HIRAI, A., MURAMATSU, M., WATARI, T., HASEGAWA, A. Direct detection of dermatophytes in skin samples based on sequences of the chitin synthase 1 (CHS1) gene. J. Vet. Med. Sci., 2003, 65, p. 267–270.
49. KARDJEVA, V., SUMMERBELL, R., KANTARDJIEV, T., et al. Forty-eight-hour diagnosis of onychomycosis with subtyping of Trichophyton rubrum strains. J. Clin. Microbiol., 2006, 44, p. 1419–1427.
50. KASZUBIAK, A., KLEIN, S., DE HOOG, G., GRÄSER, Y. Population structure and evolutionary origins of Microsporum canis, M. ferrugineum and M. audouinii. Infect. Genet. Evol., 2004, 4, p. 179–186.
51. KAWASAKI, M., ANZAWA, K., WAKASA, A., et al. Different genes can result in different phylogenetic relationships in Trichophyton species. Jap. J. Med. Mycol., 2008, 49, p. 311–318.
52. KAWASAKI, M., ANZAWA, K., USHIGAMI, T., KAWANISHI, J., MOCHIZUKI, T. Multiple gene analyses are necessary to understand accurate phylogenetic relationships among Trichophyton species. Med. Mycol. J., 2011, 52, p. 245–254.
53. KHOSRAVI, A., BEHZAD, F., SABOKBAR, A., et al. Molecular typing of Epidermophyton floccosum isolated from patients with dermatophytosis by RAPD–PCR. J Basic Microbiol., 2010, 50, p. S68–S73.
54. KIM, J., TAKAHASHI, Y., TANAKA, R., et al. Identification and subtyping of Trichophyton mentagrophytes by random amplified polymorphic DNA. Mycoses, 2001, 44, p. 157–165.
55. KIM, J. Y., CHOE, Y. B., AHN, K. J., LEE, Y. W. Identification of dermatophytes using multiplex polymerase chain reaction. Ann. Dermatol., 2011, 23, p. 304–312.
56. KONDORI, N., ABRAHAMSSON, A. L., ATAOLLAHY, N., WENNERĀS, C. Comparison of a new commercial test, Dermatophyte-PCR kit, with conventional methods for rapid detection and identification of Trichophyton rubrum in nail specimens. Med. Mycol., 2010, 48, p. 1005–1008.
57. KONDORI, N., TEHRANI, P. A., STRÖMBECK, L., FAERGEMANN, J. Comparison of Dermatophyte PCR kit with conventional methods for detection of dermatophytes in skin specimens. Mycopathologia, 2013, 176, p. 237–241.
58. LI, X.-F., WEI, T., HONG, W., et al. Direct detection and differentiation of causative fungi of onychomycosis by multiplex polymerase chain reaction-based assay. Eur. J. Dermatol., 2011, 21, p. 37–42.
59. LI, Y. C., KOROL, A. B., FAHIMA, T., BEILES, A., NEVO, E. Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review. Mol. Ecol., 2002, 11, p. 2453–2465.
60. LITZ, C., CAVAGNOLO, R. Polymerase chain reaction in the diagnosis of onychomycosis: a large, single–institute study. Brit. J. Dermatol., 2010, 163, p. 511–514.
61. LIU, D., COLOE, S., PEDERSEN, J., BAIRD, R. Use of arbitrarily primed polymerase chain reaction to differentiate Trichophyton dermatophytes. FEMS Microbiol. Lett., 1996, 136, p. 147–150.
62. LIU, D., COLOE, S., BAIRD, R., PEDERSEN, J. Rapid mini-preparation of fungal DNA for PCR. J. Clin. Microbiol., 2000, 38, p. 471–471.
63. LIU, D., COLOE, S., BAIRD, R., PEDERSEN, J. Application of PCR to the identification of dermatophyte fungi. J. Med. Microbiol., 2000, 49, p. 493–497.
64. LOEFFLER, J., HEBART, H., BIALEK, R., et al. Contaminations occurring in fungal PCR assays. J. Clin. Microbiol., 1999, 37, p. 1200–1202.
65. MEHLIG, L., GARVE, C., RITSCHEL, A., et al. Clinical evaluation of a novel commercial multiplex–based PCR diagnostic test for differential diagnosis of dermatomycoses. Mycoses, 2014, 57, p. 27–34.
66. MIYAJIMA, Y., SATOH, K., UCHIDA, T., et al. Rapid real--time diagnostic PCR for Trichophyton rubrum and Trichophyton mentagrophytes in patients with tinea unguium and tinea pedis using specific fluorescent probes. J. Dermatol. Sci., 2013, 69, p. 229–235.
67. MOCHIZUKI, T., SUGIE, N., UEHARA, M. Random amplification of polymorphic DNA is useful for the differentiation of several anthropophilic dermatophytes. Mycoses, 1997, 40, p. 405–409.
68. MOCHIZUKI, T., KAWASAKI, M., ISHIZAKI, H., et al. Molecular epidemiology of Arthroderma benhamiae, an emerging pathogen of dermatophytoses in Japan, by polymorphisms of the non-transcribed spacer region of the ribosomal DNA. J. Dermatol. Sci., 2001, 27, p. 14–20.
69. MOCHIZUKI, T., ISHIZAKI, H., BARTON, R. C., et al. Restriction fragment length polymorphism analysis of ribosomal DNA intergenic regions is useful for differentiating strains of Trichophyton mentagrophytes. J. Clin. Microbiol., 2003, 41, p. 4583–4588.
70. MOCHIZUKI, T., KAWASAKI, M., TANABE, H., et al. Molecular epidemiology of Trichophyton tonsurans isolated in Japan using RFLP analysis of non-transcribed spacer regions of ribosomal RNA genes. Jpn. J. Infect. Dis., 2007, 60, p. 188–192.
71. NAGAO, K., SUGITA, T., OUCHI, T., NISHIKAWA, T. Identification of Trichophyton rubrum by nested PCR analysis from paraffin embedded specimen in trichophytia profunda acuta of the glabrous skin. Jap. J. Med. Mycol., 2005, 46, p. 129–132.
72. NENOFF, P., ERHARD, M., SIMON, J. C., et al. MALDI-TOF mass spectrometry-a rapid method for the identification of dermatophyte species. Med. Mycol., 2013, 51, p. 17–24.
73. NINET, B., JAN, I., BONTEMS, O., et al. Identification of dermatophyte species by 28s ribosomal DNA sequencing with a commercial kit. J. Clin. Microbiol., 2003, 41, p. 826–830.
74. OHST, T., DE HOOG, S., PRESBER, W., STAVRAKIEVA, V., GRÄSER, Y. Origins of microsatellite diversity in the Trichophyton rubrum-T. violaceum clade (dermatophytes). J. Clin. Microbiol., 2004, 42, p. 4444–4448.
75. OKEKE, C. N., TSUBOI, R., KAWAI, M., HIRUMA, M., OGAWA, H. Isolation of an intron-containing partial seguence of the gene encoding dermatophyte actin (ACT) and detection of a fragment of the transcript by reverse transcription-nested PCR as a means of assessing the viability of dermatophytes in skin scales. J. Clin. Microbiol., 2001, 39, p. 101–106.
76. PASQUETTI, M., PEANO, A., SOGLIA, D., et al. Development and validation of a microsatellite marker-based method for tracing infections by Microsporum canis. J. Dermatol. Sci., 2013, 70, p. 123–129.
77. REZAEI–MATEHKOLAEI, A., MAKIMURA, K., DE HOOG, G. S., et al. Discrimination of Trichophyton tonsurans and Trichophyton equinum by PCR-RFLP and by beta-tubulin and translation elongation factor 1-alpha sequencing. Med. Mycol., 2012, 50, p. 760–764.
78. REZAEI–MATEHKOLAEI, A., MAKIMURA, K., DE HOOG, G. S., et al. Multilocus differentiation of the related dermatophytes Microsporum canis, Microsporum ferrugineum and Microsporum audouinii. J. Med. Microbiol., 2012, 61, p. 57–63.
79. REZAEI–MATEHKOLAEI, A., MAKIMURA, K., SHIDFAR, M., et al. Use of single-enzyme PCR-restriction digestion barcode targeting the internal transcribed spacers (ITS rDNA) to identify dermatophyte species. Iran J. Public Health, 2012, 41, p. 82–94.
80. REZAEI-MATEHKOLAEI, A., MAKIMURA, K., DE HOOG, S., et al. Molecular epidemiology of dermatophytosis in Tehran, Iran, a clinical and microbial survey. Med. Mycol., 2013, 51, p. 203–207.
81. SATO, T., TAKAYANAGI, A., NAGAO, K., et al. Simple PCR-based DNA microarray system to identify human pathogenic fungi in skin. J. Clin. Microbiol., 2010, 48, p. 2357–2364.
82. SHARMA, R., DE HOOG, S., PRESBER, W., GRASER, Y. A virulent genotype of Microsporum canis is responsible for the majority of human infections. J. Med. Microbiol., 2007, 56, p. 1377–1385.
83. SHARMA, R., PRESBER, W., RAJAK, R. C., GRÄSER, Y. Molecular detection of Microsporum persicolor in soil suggesting widespread dispersal in central India. Med. Mycol., 2008, 46, p. 67–73.
84. SPESSO, M., NUNCIRA, C., BURSTEIN, V., et al. Microsatellite-primed PCR and random primer amplification polymorphic DNA for the identification and epidemiology of dermatophytes. Eur. J. Clin. Microbiol. Infect. Dis., 2013, 32, p. 1009–1015.
85. SUMMERBELL, R. C., MOORE, M. K., STARINK-WILLEMSE, M., VAN IPEREN, A. ITS barcodes for Trichophyton tonsurans and T. equinum. Med. Mycol., 2007, 45, p. 193–200.
86. TAKEDA, K., NISHIBU, A., ANZAWA, K., MOCHIZUKI, T. Molecular epidemiology of a major subgroup of Arthroderma benhamiae isolated in Japan by restriction fragment length polymorphism analysis of the non-transcribed spacer region of ribosomal RNA gene. Jpn. J. Infect. Dis., 2012, 65, p. 233–239.
87. UCHIDA, T., MAKIMURA, K., ISHIHARA, K., et al. Comparative study of direct polymerase chain reaction, microscopic examination and culture-based morphological methods for detection and identification of dermatophytes in nail and skin samples. J. Dermatol., 2009, 36, p. 202–208.
88. VERRIER, J., KRÄHENBÜHL, L., BONTEMS, O., et al. Dermatophyte identification in skin and hair samples using a simple and reliable nested polymerase chain reaction assay. Brit. J. Dermatol., 2013, 168, p. 295–301.
89. WAKASA, A., ANZAWA, K., KAWASAKI, M., MOCHIZUKI, T. Molecular typing of Trichophyton mentagrophytes var. interdigitale isolated in a university hospital in Japan based on the non–transcribed spacer region of the ribosomal RNA gene. J. Dermatol., 2010, 37, p. 431–440.
90. WHITE, T. C., OLIVER, B. G., GRÄSER, Y., HENN, M. R. Generating and testing molecular hypotheses in the dermatophytes. Eukaryot Cell, 2008, 7, p. 1238–1245.
91. WISSELINK, G., VAN ZANTEN, E., KOOISTRA-SMID, A. Trapped in keratin; a comparison of dermatophyte detection in nail, skin and hair samples directly from clinical samples using culture and real-time PCR. J. Microbiol. Methods, 2011, 85, p. 62–66.
92. YANG, X., SUGITA, T., TAKASHIMA, M., et al. Differentiation of Trichophyton rubrum clinical isolates from Japanese and Chinese patients by randomly amplified polymorphic DNA and DNA sequence analysis of the non-transcribed spacer region of the rRNA gene. J. Dermatol. Sci., 2009, 54, p. 38–42.
93. YAZDANPARAST, A., JACKSON, C. J., BARTON, R. C., EVANS, E. Molecular strain typing of Trichophyton rubrum indicates multiple strain involvement in onychomycosis. Brit. J. Dermatol., 2003, 148, p. 51–54.
94. YU, J., WAN, Z., CHEN, W., WANG, W., LI, R. Molecular typing study of the Microsporum canis strains isolated from an outbreak of tinea capitis in school. Mycopathologia, 2004, 157, p. 37–41.
95. YÜKSEL, T., ?LKIT, M. Identification of rare macroconidia-producing dermatophytic fungi by real-time PCR. Med. Mycol., 2012, 50, p. 346–352.
96. ZHONG, Z., LI, R., LI, D., WANG, D. Typing of common dermatophytes by random amplification of polymorphic DNA. Jap. J. Med. Mycol., 1997, 38, p. 239–246.
Labels
Dermatology & STDs Paediatric dermatology & STDsArticle was published in
Czech-Slovak Dermatology

2014 Issue 4
-
All articles in this issue
- Současný vývoj v taxonomii dermatofytů a doporučení pro pojmenovávání klinicky významných druhů
- Molekulární epidemiologie dermatofytóz v České republice – výsledky dvouleté studie
- Detekce, identifikace a typizace dermatofytů molekulárně genetickými metodami
- Případ tinea corporis vyvolaný Microsporum incurvatum, geofilním druhem příbuzným M. gypseum
- Naše první zkušenosti s infekcemi vyvolanými Arthroderma benhamiae (Trichophyton sp.)
- Arthroderma benhamiae jako původce tinea capitis profunda a tinea corporis u dětských pacientů
- Czech-Slovak Dermatology
- Journal archive
- Current issue
- About the journal
Most read in this issue
- Naše první zkušenosti s infekcemi vyvolanými Arthroderma benhamiae (Trichophyton sp.)
- Případ tinea corporis vyvolaný Microsporum incurvatum, geofilním druhem příbuzným M. gypseum
- Arthroderma benhamiae jako původce tinea capitis profunda a tinea corporis u dětských pacientů
- Současný vývoj v taxonomii dermatofytů a doporučení pro pojmenovávání klinicky významných druhů
