-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Antigenní variabilita kmenů Bordetella pertussis izolovaných v letech 1967–2010 v České republice – možné vysvětlení vzestupu nemocnosti pertusí?
Antigenic variability of Bordetella pertussis strains isolated in 1967–2010 in the Czech Republic – possible explanation for the rise in cases of pertussis?
Objective:
Comparison of antigenic structures of Bordetella pertussis (B. pertussis) strains isolated from 1967 to 2010 in the Czech Republic.Material and methods:
Seventy strains of B. pertussis were referred to the National Reference Laboratory (NRL) for Pertussis and Diphtheria within the surveillance of pertussis from all over the Czech Republic (CR) between 1967 and 2010. To study the strains, the analysis was performed of the genome sequences encoding the surface immunogenic structures – the pertussis toxin S1 subunit gene (ptxA), pertactin gene region 1 (prnA), type 3 fimbriae gene (fim3) – and pertussis toxin promoter (ptxP) responsible for the regulation of the production of pertussis toxin.Results:
For the study set of B. pertussis strains, the sequencing analysis revealed changes in all genomic regions studied. The isolates from three periods differ in the allelic profile. In period I (1967−1978) with the use of whole cell pertussis vaccine (wP), the following two profiles were the most common: ptxP(1), ptxA(2), prnA(1), fim3(1) and ptxP(1), ptxA(1), prnA(3), fim3(1). In period 2 (1990−2007) with the switch to acellular pertussis vaccine (aP), the most common profile was: ptxP(3), ptxA(1), prnA(2), fim3(2). Period 3 (2008−2010) with the use of aP was characterized by the predominance of the following two profiles which had never been found in period 1: ptxP(3), ptxA(1), prnA(2), fim3(2) and ptxP(3) ptxA(1), prnA(2), fim3(1).Conclusions:
Sequencing of the genomic regions ptxP, ptxA, prnA, and fim3 of B. pertussis strains isolated in the CR between 1967 and 2010 confirmed changes in the allelic variants of these regions. The incidence of strains carrying the new allelic variants was increasing after 1995 at the expense of those carrying the original variants. The study results can be interpreted as a partial genetic escape of pathogenic strains of B. pertussis beyond the reach of the pertussis vaccines.Keywords:
Bordetella pertussis strain – isolate – sequencing – epidemiology – vaccine
Autoři: J. Zavadilová 1; D. Lžičařová 1; M. Musílek 1
; P. Křížová 1
; K. Fabiánová 1,2
Působiště autorů: Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha 1; Ústav epidemiologie, 3. LF UK, Praha 2
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 64, 2015, č. 3, s. 130-138
Kategorie: Původní práce
Souhrn
Cíl práce:
Porovnání antigenních struktur kmenů Bordetella pertussis (B. pertussis) z let 1967–2010 v České republice.Materiál a metodiky:
Do studie bylo zařazeno 70 kmenů B. pertussis, které byly v rámci surveillance pertuse doručeny do Národní referenční laboratoře (NRL) pro pertusi a difterii v letech 1967–2010 z celé České republiky (ČR). Kmeny byly studovány analýzou genomových sekvencí kódujících povrchové imunogenní struktury – gen pro S1 podjednotku pertusového toxinu (ptxA), oblast 1 genu pro pertaktin (prnA), gen pro fimbrie typu 3 (fim3) – a promotor genu pro pertusový toxin (ptxP) odpovědný za řízení produkce pertusového toxinu.Výsledky:
Ve studovaném souboru kmenů B. pertussis byly sekvenační analýzou prokázány změny ve všech sledovaných oblastech genomu. Alelický profil izolátů ve třech srovnávaných obdobích se liší. V prvním období (1967−1978, používání celobuněčné pertusové vakcíny (wP)) byly nejčastěji zjištěny dva profily: ptxP(1), ptxA(2), prnA(1), fim3(1); ptxP(1), ptxA(1), prnA(3), fim3(1). Ve druhém období (1990−2007, přechod na acelulární pertusovou vakcínu (aP)) byl nejčastěji zjištěn profil ptxP(3), ptxA(1), prnA(2), fim3(2). Ve třetím období (2008−2010, používání aP) zcela převládaly dva alelické profily, které se v prvním období vůbec nevy-skytovaly: ptxP(3), ptxA(1), prnA(2), fim3(2); ptxP(3) ptxA(1), prnA(2), fim3(1). Závěry: Sekvenací oblastí genomu ptxP, ptxA, prnA a fim3 u kmenů B. pertussis izolovaných v ČR v období 1967–2010 byly potvrzeny změny alelických variant těchto oblastí. Výskyt kmenů nesoucích nové alelické varianty narůstá po roce 1995 na úkor kmenů nesoucích varianty původní. Výsledky studie lze interpretovat jako částečný genetický únik patogenních kmenů B. pertussis mimo účinnost pertusových vakcín.Klíčová slova:
kmen Bordetella pertussis – izolát – sekvenace – epidemiologie – vakcínaÚVOD
Před zavedením očkování patřilo onemocnění pertusí mezi hlavní příčiny nemocnosti a úmrtnosti dětí. Plošná vakcinace proti pertusi (dávivému, černému kašli) byla ve většině států zavedena v polovině 20. století, což přispělo k významnému snížení nemocnosti a úmrt-nosti dětí na pertusi. V sedmdesátých letech se pak v zemích s dobrou dostupností vakcín a vysokou proočkovaností dětské populace snížily počty nemocných na minimum.
V posledních dvou desetiletích 20. století a poté až do současné doby je však ze států se stabilní dobrou proočkovaností proti pertusi hlášen nárůst incidence onemocnění, a to nejen v dětské populaci, ale také u dospívajících a dospělých. Zvýšená incidence pertuse je zaznamenávána v mnoha oblastech a státech USA, Kanady, Austrálie a Evropy [1–9].
V ČR bylo zahájeno pravidelné očkování proti pertusi v roce 1958. K vakcinaci byla použita wP vyrobená v Ústavu sér a očkovacích látek z „domácích“ aktuálně kolujících kmenů B. pertussis v kombinaci s difterickým a tetanickým toxoidem (ALDITEPERA). Očkování dětí probíhalo v pětidávkovém schématu. Od roku 2001 byla pro pravidelné očkování používána wP zahraničního výrobce. Od roku 2002 byla již na českém trhu k dispozici i vakcína aP aplikovaná z indikace pediatra nebo za úhradu. Od roku 2005 aP nahradila pátou dávku očkování proti pertusi a v roce 2007 se aP stala součástí pravidelného očkování. Od roku 2009 bylo zavedeno přeočkování 6. dávkou aP. Očkovací látka aP používaná v rámci pravidelného očkování obsahuje tři komponenty: pertusový toxin (PT), pertaktin (PRN) a filamentózní hemaglutinin (FHA). Od roku 2010 je k dispozici na trhu alternativní aP proti pertusi, která obsahuje PT, PRN, FHA a antigeny fimbrií Fim2 a Fim3. Tato vakcína je však určena pouze pro přeočkování.
Incidence a úmrtnost pertuse po zavedení pravidelného očkování v ČR rychle klesala z původních desetitisíců případů ročně na 5–48 případů od druhé poloviny 70. let do roku 1992; většina případů byla u dětí mladších tří let. Od roku 1993 má hlášená incidence pertuse v ČR trvale vzestupný trend. V ČR se proočkovanost udržuje na velmi dobré úrovni, která umožňuje nastolení kolektivní imunity. Například u dětí do dvou let věku dosahuje 99 % [10].
Návrat pertuse do populace s vysokou proočkovaností může být způsoben mnoha faktory. Mezi nejčastěji uváděné patří zkvalitnění surveillance a laboratorní diagnostiky onemocnění, lepší povědomí odborné a laické veřejnosti o pertusi, druh používaných vakcín a vyvanutí („waning“) imunity po očkování [7, 11].
K významným faktorům nárůstu onemocnění patří také adaptace původce onemocnění, bakterie B. pertussis, na imunitu zprostředkovanou očkováním; tedy šíření kmenů B. pertussis antigenně odlišných od kmenů použitých ve vakcínách. Nové klony B. pertussis vzniklé selektivním tlakem vakcinace mají vlastnosti, které jim umožňují vyhnout se dostatečné účinnosti používaných vakcín [12].
Pro sekvenační analýzu byly zvoleny oblasti genomu ptxP, ptxA, prnA a fim3. Jde o genomové sekvence kódující struktury významně se uplatňující v patogenezi pertuse a dostatečně polymorfní pro účely typizace. Tyto struktury jsou zároveň složkami aP dostupných v ČR.
Oblast genomu PtxP je součástí promotoru ptx-ptl operonu kódujícího PT a jeho transportní systém, odpovědný za řízení produkce PT. Exprese genů tohoto operonu je regulována BvgA systémem, který řídí expresi stovek genů v závislosti na změnách vnějších podmínek [13–15]. Polymorfizmus ptxP byl zjištěn v oblasti vazebných míst pro RNA polymerázu a pro transkripční faktor BvgA [16]. V současné době je známo 18 alelických variant sekvence ptxP [17].
Oblast genomu PtxA B. pertussis kóduje S1 podjednotku PT. PT je tvořen celkem 6 podjednotkami, S1–S5, S1 je podjednotkou katalytickou. Toxin působí mechanismem ADP ribosylace G-proteinů eukaryotních buněk. Polymorfní oblast podjednotky S1 má zásadní význam pro vazbu na receptor T lymfocytů (TCR) [18]. Ostatní podjednotky mají transportní funkci, tvoří transportní systém typu IV [13]. V oblastech kódujících ostatní podjednotky PT nebyl pozorován významnější polymorfismus [19]. PT je jediným známým faktorem patogenity odpovědným za klinickou manifestaci pertuse – u imunologicky naivních jedinců (v našich podmínkách vesměs dosud neočkované děti, především kojenci) způsobuje výraznou leukocytózu s absolutní lymfocytózou a syndrom plicní hypertenze, který může být příčinou respiračního a oběhového selhání a smrti [20, 21].
Další významnou funkcí PT je inhibice časné zánětlivé odpovědi, především produkce prozánětlivých cytokinů a chemokinů a migrace neutrofilních leukocytů do místa infekce, což usnadňuje kolonizaci sliznic hostitele v iniciální fázi infekce [13, 14]. V současné době je známo 8 alelických variant sekvence ptxA [17]. Inaktivovaný PT je součástí všech aP dostupných v ČR.
PrnA je variabilní oblast 1 genu kódujícího PRN, autotransportní sekreční protein s funkcí adhezinu [13, 22]. Vazebným místem pro adhezi je u PRN triplet aminokyselin Arg-Gly-Asp (RGD), známý jako místo vazby (protein-protein) s eukaryotní buňkou. Variabilní oblast 1 se nachází v blízkosti tohoto RGD motivu [22]. Variabilní oblast 1 PRN obsahuje epitop významný pro tvorbu protektivních protilátek. Polymorfismus v této oblasti ovlivňuje strukturu a tím i protektivitu těchto specifických protilátek [23, 24]. Jednotlivé alelické varianty prnA se liší počtem repetic aminokyselin GGXXP (Gly-Gly-X-X-Pro) ve variabilní oblasti 1, celkem je známo 13 variant [22]. V genu pro PRN se nachází i druhá polymorfní oblast prnB, její sekvenace je však doporučována pouze v případě zjištění nové alelické varianty prnA [22]. PRN je obsažen ve většině aP používaných v ČR.
Oblasti fim2 a fim3 kódují velkou (major) podjednotku fimbrií, vláknitých povrchových struktur bílkovinné povahy, jejichž předpokládanou funkcí je zprostředkování adherence k respiračnímu řasinkovému epitelu [14]. Fimbrie hrají roli i v imunomodulaci časné (T-independentní) IgM odpovědi, indukují Th2-dependentní odpověď [13]. Dosud jsou známy 4 alelické varianty fim3 [17]. Oblast fim2 nebyla v předkládané studii sekvenována a je uvedena jen pro úplnost. V laboratorní charakteristice kmenů B. pertussis je velká podjednotka fimbrií strukturou určující sérotyp Fim2/3/2,3 [25]. Fimbrie typu 2 a 3 obsahuje jedna z aP používaných v ČR pro přeočkování (5 let, 10–11 let, dospělí).
MATERIÁL A METODIKA
Kmeny Bordetella pertussis
Do studie bylo zařazeno 70 kmenů B. pertussis, které byly v rámci surveillance pertuse doručeny do NRL pro pertusi a difterii v letech 1967–2010 z celé ČR. Z let 1972–1976, 1991–1992, 1996–1998, 2000–2003 a 2005–2006 se kmeny nedochovaly. Studované období více než 40 let bylo rozděleno na 3 časové úseky:
- 1967−1978 (20 izolátů) – období wP + klesající nemocnost pertuse.
- 1990−2007 (9 izolátů) – období přechodu z wP na aP + počátek vzestupu nemocnosti pertuse.
- 2008−2010 (41 izolátů) – období aP + pokračující vzestup nemocnosti pertuse.
Do roku 2004 byly kmeny uchovávány v lyofilizovaném stavu, od roku 2007 jsou uchovávány zmrazením při -70 °C. Kmeny B. pertussis byly kultivovány na Bordet--Gengou a Charcoal agaru a inkubovány při teplotě 35 ± 1 °C po dobu 96 hodin v normální atmosféře. Potvrzení druhové identifikace bylo provedeno sklíčkovou aglutinací s diagnostickým sérem Bordetella pertussis (Remel Ltd, USA). Sérotypizace pro určení typu fimbrií byla provedena rovněž sklíčkovou aglutinací s použitím monoklonálních protilátek Monoclonal Antibody for Serotyping Bordetella pertussis Fimbrial Antigen 2, 1 st WHO IS (NIBSC, UK) a Monoclonal Antibody for Serotyping Bordetella pertussis Fimbrial Antigen 3, 1 st WHO IS (NIBSC, UK) podle [25].
Sekvenace DNA
Izolace DNA byla provedena soupravou QIAamp DNA Mini Kit (Qiagen, SRN).
Kmeny byly studovány analýzou genomových sekvencí kódujících povrchové imunogenní struktury – gen pro S1 podjednotku pertusového toxinu (ptxA), oblast 1 genu pro pertaktin (prnA), gen pro fimbrie typu 3 (fim3) – a promotor genu pro pertusový toxin (ptxP) odpovědný za řízení produkce pertusového toxinu. Amplifikace genových oblastí byly provedeny podle publikovaných postupů [19, 16, 22] při užití HotStarTaq Master Mix kitu (Qiagen, SRN) a termocykléru Labcycler (SensoQuest, SRN). Pro přečištění amplikonů byl použit kit Wizard SV 96 DNA Purification System (Promega, USA) podle návodu výrobce. Sekvenační produkty byly získány při užití BigDye Terminator v3.1 kitu (Applied Biosystems, USA) a termocykléru Veriti (Applied Biosystems, USA), přečištěny srážením 70% etanolem a centrifugací. Sekvence genových úseků byly získány analyzátorem 3130xL (Applied Biosystems, USA) pomocí software Lasergene (DNASTAR, USA).
VÝSLEDKY
Ve studii bylo analyzováno 70 kmenů B. pertussis z období 1967–2010, které bylo rozděleno na 3 časové úseky:
1. období (1967−1978), 2. období (1990−2002), 3. období (2008–2010).
Přehled zjištěných alelických variant je uveden v tabulce 1.
Tab. 1. Souhrnná charakteristika kmenů B. pertussis, Česká republika, 1967–2010 Table 1. Summary characteristics of B. pertussis strains, Czech Republic, 1967–2010 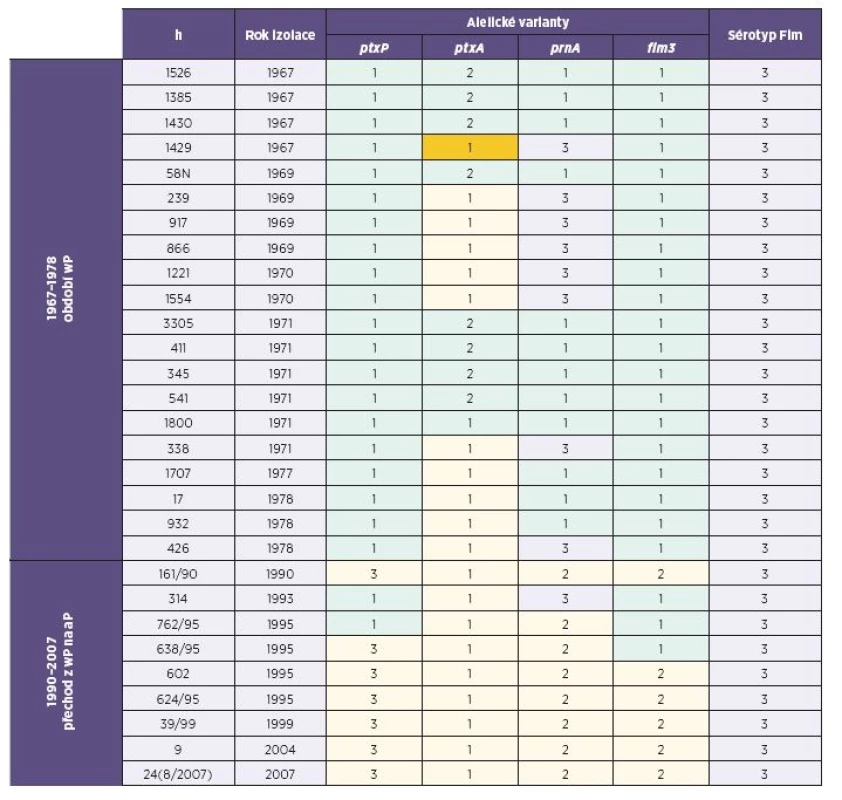

wP - celobuněčná pertusová vakcína, aP - acelulární pertusová vakcína, ptxP - promotor genu pro pertusový toxin, ptxA - gen pro S1 podjednotku pertusového toxinu, prnA - oblast 1 genu pro pertaktin, fim3 - gen pro fimbrie typu 3, Fim - fimbrie, parapert - B. parapertussis wP – whole cell pertussis vaccine, aP – acellular pertussis vaccine, ptxP – pertussis toxin promotor, ptxA – pertussis toxin S1 subunit gene, prnA – pertactin gene region 1, fim3 – type 3 fimbriae gene, Fim – fimbriae, parapert – B. parapertusis Sekvenací oblasti ptxP bylo zjištěno, že u izolátů z roku 1978 a starších se vyskytovala výhradně alelická varianta ptxP(1). U izolátů z let 1990 až 2007 se současně s variantou ptxP(1) vyskytovala i varianta ptxP(3), která od roku 2008 zcela převážila.
Pokud jde o sekvenci ptxA, u kmenů izolovaných do roku 1978 se vyskytovaly současně varianty ptxA(1) a ptxA(2). U kmenů izolovaných od roku 1990 se již vyskytuje pouze varianta ptxA(1).
V případě oblasti prnA se u kmenů izolovaných do roku 1978 vyskytovaly současně varianty prnA(1) a prnA(3), v období 1990−2007 varianty prnA(2) a prnA(3), od roku 2008 se vyskytuje pouze varianta prnA(2). V roce 2010 se vyskytl netypický kmen, u něhož se sekvence oblasti prnA shodovala se sekvencí typickou pro kmen Bordetella parapertussis.
Sekvenace oblasti fim3 ukázala, že kmeny izolované do r. 1978 byly nositeli alelické varianty fim3(1), v obdobích 1990−2007 a 2008−2010 se vyskytují varianty fim3(1) a fim3(2).
Alelický profil izolátů ve třech srovnávaných obdobích se liší – tabulka 2 a grafy 1–3. V prvním období (1967−1978, wP vakcína) byly nejčastěji zjištěny dva profily: ptxP(1), ptxA(2), prnA(1), fim3(1); ptxP(1), ptxA(1), prnA(3), fim3(1). Ve druhém období (1990−2007, přechod na aP vakcínu) byl nejčastěji zjištěn profil ptxP(3), ptxA(1), prnA(2), fim3(2). Ve třetím období (2008−2010, aP vakcína) zcela převládaly dva alelické profily, které se v prvním období vůbec nevyskytovaly: ptxP(3), ptxA(1), prnA(2), fim3(2); ptxP(3) ptxA(1), prnA(2), fim3(1).
Tab. 2. Alelický profil izolátů B. pertussis, Česká republika, 1967-2010 Table 2. Allelic profiles of B. pertussis isolates, Czech Republic, 1967–2010 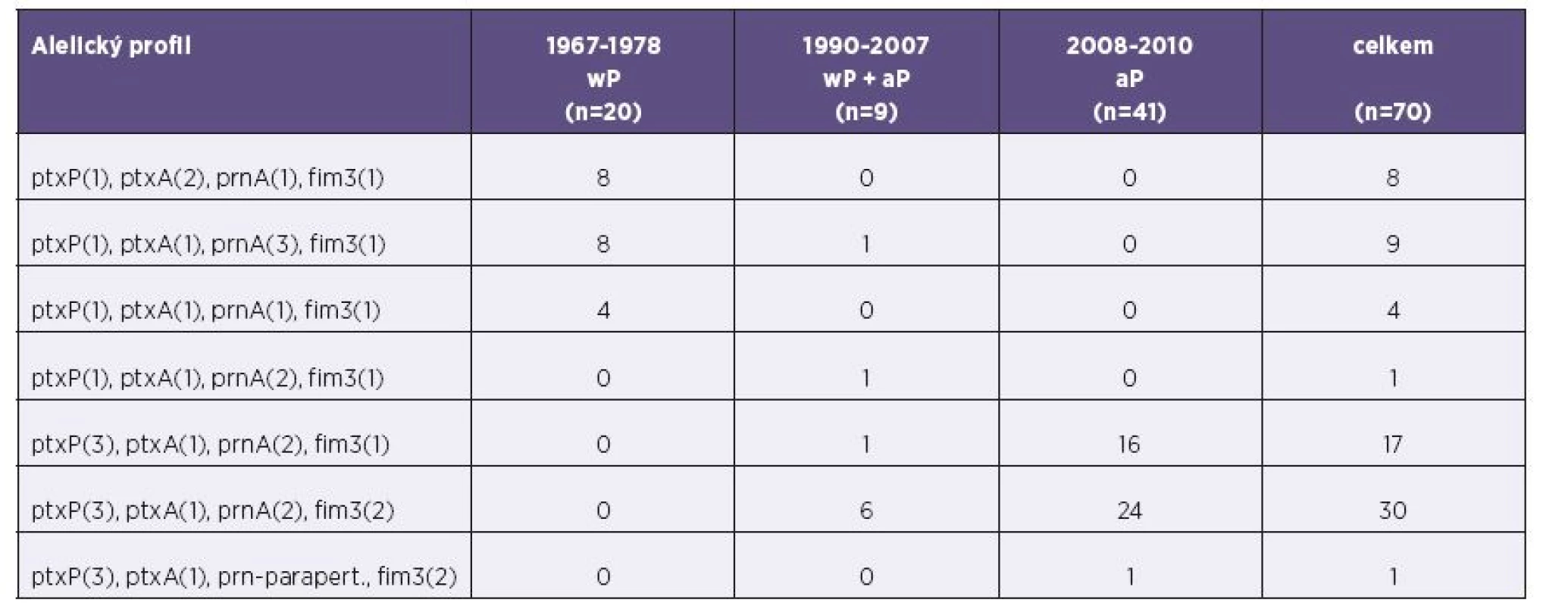
wP - celobuněčná pertusová vakcína, aP - acelulární pertusová vakcína, ptxP - promotor genu pro pertusový toxin, ptxA - gen pro S1 podjednotku pertusového toxinu, prnA - oblast 1 genu pro pertaktin, fim3 - gen pro fimbrie typu 3, parapert - B. parapertussis wP – whole cell pertussis vaccine, aP – acellular pertussis vaccine, ptxP – pertussis toxin promotor, ptxA – pertussis toxin S1 subunit gene, prnA – pertactin gene region 1, fim3 – type 3 fimbriae gene, parapert – B. parapertussis Graf 1. Alelický profil izolátů B. pertussis, Česká republika, 1967–1978 Fig. 1. Allelic profiles of B. pertussis isolates, Czech Republic, 1967–1978 
ptxP – promotor genu pro pertusový toxin, ptxA – gen pro S1 podjednotku pertusového toxinu, prnA – oblast 1 genu pro pertaktin, fim3 – gen pro fimbrie typu 3 ptxP – pertussis toxin promoter gene, ptxA – pertussis toxin S1 subunit gene, prnA – pertactin gene region 1, fim3 – type 3 fimbriae gene Sérotyp izolovaných kmenů je uveden v tabulce 1. U izolátů z období 1967–1978 a 1990–2007 se vyskytoval výhradně sérotyp Fim3, v období 2008–2010 se vyskytují sérotypy Fim3, Fim2, Fim2,3.
Graf 2. Alelický profil izolátů B. pertussis, Česká republika, 1990–2007 Fig. 2. Allelic profiles of B. pertussis isolates, Czech Republic, 1990–2007 
ptxP – promotor genu pro pertusový toxin, ptxA – gen pro S1 podjednotku pertusového toxinu, prnA – oblast 1 genu pro pertaktin, fim3 – gen pro fimbrie typu 3 ptxP – pertussis toxin promoter gene, ptxA – pertussis toxin S1 subunit gene, prnA – pertactin gene region 1, fim3 – type 3 fimbriae gene Graf 3. Alelický profil izolátů B. pertussis, Česká republika, 2008–2010 Fig. 1. 3. Allelic profiles of B. pertussis isolates, Czech Republic, 2008–2010 
ptxP – promotor genu pro pertusový toxin, ptxA – gen pro S1 podjednotku pertusového toxinu, prnA – oblast 1 genu pro pertaktin, fim3 – gen pro fimbrie typu 3, parapert – B. parapertussis ptxP – pertussis toxin promoter gene, ptxA – pertussis toxin S1 subunit gene, prnA – pertactin gene region 1, fim3 – type 3 fimbriae gene, parapert – B. parapertussis DISKUSE
Změny ve struktuře antigenních determinant B. pertussis vedoucí k zvýšení odolnosti kmenů cirkulujících v hostitelské populaci vůči selekčnímu tlaku postvakcinačních protilátek mohou být jednou z příčin narůstající incidence pertuse v populacích s vysokou proočkovaností [15]. Nejčastější alelický profil (ptxA(1),ptxP(3), prnA(2), fim3(2) a fim2(1) současně cirkulujících kmenů ve světě je odlišný od profilu kmenů použitých k výrobě vakcíny [26, 27] a izoláty, které neexprimují PRN přibývají [28–30]. Alelická varianta ptxP(3) v současnosti ve světě dominuje [31] a některé studie indikují, že kmeny s přítomností varianty ptxP(3) mají zvýšenou virulenci ve srovnání s kmeny s variantou ptxP(1) [32]. Nejnovější studie ve Velké Británii prokázala, že geny kódující antigeny aP vakcíny se mění více, než geny ostatních povrchových proteinů. Toto platilo již před zavedením očkování proti pertusi, ale po zavedení aP vakcíny jsou tyto změny více vyjádřeny [33].
Dalšími příčinami nárůstu incidence pertuse, o kterých se uvažuje, jsou vyvanutí (waning) postvakcinační i post-infekční imunity a změny v diagnostice a fungování systému surveillance. V podmínkách ČR jde také o zlepšení povědomí lékařské veřejnosti o problematice pertuse, především u starších dětí, dospívajících a dospělých, a uplatnění citlivých a efektivních diagnostických postupů. Nejedná se tedy přímo o změny, ale spíše o zefektivnění postupů v rámci stávajícího programu surveillance. Pro poslední uvedený důvod hovoří významné rozdíly v relativním počtu hlášených případů mezi některými zeměmi s vysoce proočkovanou populací, proti pak údaje ze zemí, kde se v rámci surveillance sleduje počet hospitalizací v souvislosti s diagnózou pertuse (důvody pro hospitalizaci jsou v čase stabilnější, protože vycházejí ze závažnosti stavu pacienta) a kde je pozorován nárůst hospitalizací svědčící pro skutečný, nikoli arteficiální nárůst incidence pertuse [34].
Genetická variabilita oblastí kódujících antigenní determinanty nebo faktory patogenity se začala sledovat v zemích s dlouhodobě vysoce proočkovanou populací po nárůstu incidence pertuse [35, 36–40]. Byla prováděna sekvenační analýza oblastí genomu kódujících různé faktory patogenity s cílem nalézt oblasti genomu s optimální mírou polymorfismu a sledovat jejich změny v čase. Polymorfismus byl prokázán v genech bipA, fhaB, fim2, fim3, ompQ, ptxS3, tcfA, a vag8 [41, 42]. Významná variabilita byla zjištěna v oblastech kódujících S1 podjednotku PT, promotor genu pro PT, oblast 1 genu pro PRN, velkou podjednotku fimbrií typu 2 a 3 [19, 26]. Bottero et al. [39] prokázali projevy variability vybraných oblastí ve stuktuře proteinů.
Studie v Nizozemí a ve Finsku prokázaly polymorfismus v genech, které kódují dva důležité faktory virulence B. pertussis: PRN a PT. [35]. Právě protilátky proti PRN a PT byly pro ochranu před infekcí považovány za velmi významné a důležité [43–44].
K sekvenační typizaci B. pertussis není možné použít tzv. housekeeping geny, jako je tomu u jiných bakteriálních druhů. Druh B. pertussis je geneticky vysoce monomorfní [45] a v oblasti housekeeping genů není v rámci druhu možné nalézt dostatečně polymorfní oblasti pro vytvoření systému sekvenační typizace [46]. Sekvenační typizace B. pertussis je proto založena na analýze oblastí kódujících vybrané antigenní determinanty nebo faktory patogenity [19].
Při sekvenační analýze souboru studovaných kmenů B. pertussis v ČR byly zjištěny 2 alelické varianty oblasti ptxP, a to ptxP(1) a ptxP(3). Varianta ptxP(3) se poprvé objevila v r. 1990 a zcela převážila po r. 1995. PtxP(3) je alelická varianta promotoru genu pro ptx-ptl operon, podmiňující zvýšenou produkci pertusového toxinu. Mutace ptxP(3) poskytuje pravděpodobně svému nositeli výhodu související jen nepřímo se selekčním tlakem protilátek [15]. PT usnadňuje mechanismem imunosuprese kolonizaci sliznic dýchacích cest hostitele [13, 14]. K potlačení kolonizace může při jeho zvýšené produkci být zapotřebí vyšší koncentrace sérových protilátek proti PT [15]. Bart et al. [47] datují v práci, zabývající se charakteristikou celosvětové populace B. pertussis a jejích genetických změn ve vztahu k očkování, vznik alelické varianty ptxP(3) do rozmezí let 1974–1977, její predominanci pak po roce 1995. Tomu odpovídají výsledky předkládané studie. V Nizozemsku zaznamenali Mooi et al. [16] časovou koincidenci nárůstu počtu hlášených případů pertuse s nárůstem výskytu kmenů nesoucích alelickou variantu ptxP(3). Porovnání četnosti hospitalizací a úmrtnosti v obdobích s různou mírou výskytu varianty ptxP(3) v této práci nepřímo ukazuje na vyšší míru virulence kmenů nesoucích tuto variantu.
V oblasti ptxA kódující S1 podjednotku PT byly sekvenací kmenů pocházejících z území ČR zjištěny 2 alelické varianty, ptxA(1) a ptxA(2). Do roku 1971 byly obě zastoupeny rovnoměrně, po roce 1971 byla u všech studovaných kmenů zjištěna pouze varianta ptxA(1). Vznik alely ptxA(1) předpokládají Bart et al. mezi roky 1921 a 1932, nárůst výskytu celosvětově pozorují od 60. let, výskyt nad 90 % po roce 1995 [47].
Sekvenací oblasti 1 genu pro PRN (prnA) byly ve studovaném souboru kmenů nalezeny 3 různé alelické varianty, prnA(1), prnA(3) a prnA(2). V období před rokem 1995 převažovaly první dvě uvedené varianty, varianta prnA(2) se poprvé objevila v roce 1990 a zcela převažovala od roku 1995. Od roku 1995 nebyly již v souboru zjištěny alelické varianty prnA(1) ani prnA(3). Z evropských zemí s vysokou proočkovaností vykazuje podobný trend Finsko [27], ve Francii a Nizozemsku naopak výskyt variant prnA(1) a prnA(3) přetrvává [35, 48]. Postupný nárůst výskytu kmenů nesoucích prnA(2) je patrný i v celosvětovém trendu [47]. Protilátky proti PRN jsou specifické pro varianty PRN kódované různými alelickými variantami. Varianta prnA(1) je obsažena v genomu vakcinačních kmenů [49] a izolátů z období před zahájením plošné vakcinace a krátce po něm. Navozuje srovnatelně účinnou imunitní odpověď proti variantě prnA(3), ale nikoli proti prnA(2) [23]. U varianty prnA(3) se předpokládá jiná selekční výhoda, např. lepší schopnost adheze [15, 50].
V oblasti fim3 byly nalezeny alelické varianty fim3(1) a fim3(2). Fim3(2) se poprvé objevila v roce 1990, od té doby se vyskytují obě varianty. Tyto dvě varianty predominují také celosvětově, výskyt fim3(2) narůstá významněji po roce 2000 [47]. Vznik alelické varianty fim3(2) je datován do období 1986–1989 [47]. Odlišnost alelických variant fim3(2) a fim3(1) se projevuje i ve fenotypu jako záměna aminokyseliny na pozici 87 [51].
Samotná sekvenační analýza zmíněných oblastí genomu B. pertussis nedává odpověď na otázku, zda kmeny nesoucí nové alelické varianty patří k mnoha vzájemně nepříbuzným klonům, u kterých došlo ke shodným změnám mechanismem genetického transferu a které se šíří v různých populačních celcích vlivem selekčního tlaku postvakcinačních protilátek, nebo zda jde o celosvětovou expanzi kmenů náležejících k témuž klonu. Pro porozumění tomu, zda a jak se populace B. pertussis přizpůsobuje selekčnímu tlaku postvakcinačních protilátek a do jaké míry jsou kmeny nesoucí totožné alelické varianty polymorfních oblastí genomu kódujících faktory patogenity evolučně příbuzné, je klíčová znalost struktury a evoluční historie populace B. pertussis na celosvětové úrovni [50]. Výsledky sekvenační analýzy v kombinaci s metodou PFGE (pulzní gelová elektroforéza) [45], microarray hybridizační techniky v kombinaci se sekvenací vybraných oblastí genomu [52, 53] a analýzy SNP (Single Nucleotide Polymorphism) mutací pro určení vzájemné genetické příbuznosti izolátů B. pertussis z různých geografických lokalit [17, 50] hovoří pro druhou možnost. Celosvětově se šířící klon je nositelem alelických variant ptxP(3), ptxA(1), prnA(2) [50]. Pro vliv selekčního tlaku vakcinačních protilátek na vznik a šíření tohoto klonu svědčí vyšší podíl SNP mutací v oblastech kódujících antigenní determinanty oproti oblastem tzv. „backbone“ genů (geny, jejichž produkty odpovídají za udržování základních životních funkcí organismu) [50].
Nejrozšířenější aP vakcína, používaná k plošné vakcinaci také v ČR, obsahuje antigeny připravené z typového kmene Tohama I. Tento kmen je nositelem alelických variant ptxP(1), ptxA(2) a prnA(1) [47], tedy těch, které jsou v populaci B. pertussis na území ČR na ústupu, případně již vymizely. To může být příčinou snížené účinnosti aP vakcíny.
Výsledky studie odpovídají nálezům v dalších evropských i mimoevropských regionech s dlouhodobě vysokou proočkovaností populace. Narůstající hlášenou incidenci pertuse v ČR, způsobenou kromě dalších faktorů také antigenně změněnými kmeny, se současným očkovacím schématem nedaří potlačit.
ZÁVĚRY
Sekvenací oblastí genomu ptxP, ptxA, prnA a fim3 u kmenů B. pertussis izolovaných v České republice v období 1967–2010 byly potvrzeny změny alelických variant těchto oblastí. Výskyt kmenů nesoucích nové alelické varianty narůstá po roce 1995 na úkor kmenů nesoucích varianty původní. Výsledky studie lze interpretovat jako částečný genetický únik patogenních kmenů B. pertussis mimo účinnost vakcín.
Poděkování
Práce byla podpořena výzkumným grantem NT/14058-3 Interní grantové agentury (IGA) MZ ČR.
Poděkování všem, kteří se podílejí na surveillance pertuse v České republice a posílají izoláty B. pertussis do NRL pro pertusi a difterii.
Do redakce došlo dne 4. 3. 2015.
Adresa pro korespondenci:
Mgr. Jana Zavadilová
Centrum epidemiologie a mikrobiologie
Státní zdravotní ústav
Šrobárova 48
100 42 Praha 10
e-mail: jzavadilova@szu.cz
Zdroje
1. Resurgence of pertussis – United States, 1993, Morb Mortal Wkly Rep, 1993;42(49):952–953.
2. Milord F. Resurgence of pertussis in Montérégie, Quebec – 1990–1994. Can Commun Dis Rep, 1995;21(5):40–44.
3. Poynten M, McIntyre PB, Mooi FR, et al. Temporal trends in circulating Bordetella pertussis strains in Australia. Epidemiol Infect, 2004;132(2):185–193.
4. Celentano LP, Massari M, Paramatti D, et al. Resurgence of pertussis in Europe. Pediatr Infect Dis J, 2005;24(9):761–765.
5. Gonçalves G, Machado E, Gouveia E, et al. Resurgence of pertussis in northern Portugal: two severe cases in very young children. Euro Surveill, 2005;10(25):pii=2731 [online]. [cit. 2014–11–14]. Dostupný na www: http://www.eurosurveillance.org/ViewArticle.aspx?ArticleId=2731.
6. Grilc E, Pirnat N. Pertussis outbreak in recently vaccinated children in a kindergarten in Ljubljana during a resurgence in pertussis incidence. Euro Surveill, 2005;10(33):pii=2779 [online]. [cit. 2010–08–05]. Dostupný na www: http://www.eurosurveillance.org/ViewArticle.aspx?ArticleId=2779.
7. de Melker HE, Conyn-van Spaendock MA, Rümke HC, et al. Pertussis in The Netherlands: an outbreak despite high levels of immunization with whole-cell vaccine. Emerg Infect Dis, 1997;3(2):175–178.
8. Gzyl A, Augustynowicz E, Rabczenko D, et al. Pertussis in Poland. Int J Epidemiol, 2004;33(2):358–365.
9. Gustafsson L, Hessel L, Storsaeter J, et al. Long-term follow-up of Swedish children vaccinated with acellular pertussis vaccines at 3, 5, and 12 months of age indicates the need for a booster dose at 5 to 7 years of age. Pediatrics, 2006;118(3):978–984.
10. Dlhý J. Administrativní kontrola proočkovanosti v České republice k datu 31. 12. 2010. Zprávy Centra epidemiologie a mikrobiologie, SZÚ, Praha, 2012; 21(3):92–97.
11. Bass JW, Stephenson SR. The return of pertussis. Pediatr Infect Dis J, 1987;6(2):141–44.
12. Lam C, Octavia S, Bahrame Z, et al. Selection and emergence of pertussis toxin promoter ptxP3 allele in the evolution of Bordetella pertussis. Infect Genet Evol, 2012;12(2):492–495.
13. Mattoo S, Cherry JD. Molecular pathogenesis, epidemiology, and clinical manifestations of respiratoty infections due to Bordetella pertussis and other Bordetella subspecies. Clin Microbiol Rev, 2005;18(2):326–382.
14. Melvin JA, Scheller EV, Miller JF, et al. Bordetella pertussis pathogenesis: current and future challenges. Nat Rev Microbiol, 2014;12(4):274–288.
15. Mooi FR. Bordetella pertussis and vaccination: the persistence of a genetically monomorphic pathogen. Infect Genet Evol, 2010;10(1):36–49.
16. Mooi FR, van Loo IH, van Gent M, et al. Bordetella pertussis strains with increased toxin production associated with pertussis resurgence. Emerg Infect Dis, 2009;15(8):1206–1213.
17. van Gent M, Bart MJ, van der Heide HG, et al. Small mutations in Bordetella pertussis are associated with selective sweeps. PLoS One, 2012;7(9):e46407 [online]. [cit. 2014–09–21]. Dostupný na www: http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0046407.
18. De Magistris MT, Di Tommaso A, Domenighini M et al. Interaction of the pertussis toxin peptide containing residues 30 – 42 with DRI and the T-cell receptors of 12 human T-cell clones. Proc Natl Acad Sci USA, 1992;89(7):2990–2994.
19. van Loo IHM, Heuvelman KJ, King AJ, et al. Multilocus Sequence Typing of Bordetella pertussis based on surface protein genes. J Clin Microbiol, 2002;40(6):1994–2001.
20. Cherry JD, Paddock CD. Pathogenesis and histopathology of pertussis: Implications for vaccination. Expert Rev Vaccines, 2014; 13(9):1115–1123.
21. Paddock CD, Sanden GN, Cherry JD, et al. Pathology and pathogenesis of fatal Bordetella pertussis infection in infants. Clin Infect Dis, 2008;47(3):328–338.
22. Mooi FR, Hallander H, Wirsing von König CH, et al. Epidemiological typing of Bordetella pertussis isolates: recommendations for a standard methodology. Eur J Clin Microbiol Infect Dis, 2000;19(3):174–181.
23. He Q, Mäkinen J, Berbers G, et al. Bordetella pertussis protein pertactin induces type-specific antibodies: one possible explanation for the emergence of antigenic variants? J Infect Dis, 2003;187(8):1200–1205.
24. King AJ, Berbers G, van Oirschot, et al. Role of the polymorphic region 1 of the Bordetella pertussis protein pertactin in immunity. Microbiology, 2001;147(11):2885–2895.
25. van Gent M, de Greef SC, van der Heide HG, et al. An investigation into the cause of the 1983 whooping cough epidemic in the Netherlands. Vaccine, 2009;27(13):1898–1903.
26. Litt DJ, Neal SE, Fry NK. Changes in genetic diversity of the Bordetella pertussis population in the United Kingdom between 1920 and 2006 reflect vaccination coverage and emergence of a single dominant clonal type. J Clin Microbiol, 2009;47(3):680–688.
27. Van Loo IH, Mooi FR. Changes in the Dutch Bordetella pertussis population in the first 20 years after the introduction of whole-cell vaccines. Microbiology, 2002;148(7):2011–218.
28. Bouchez V, Brun D, Cantinelli T, et al. First report and detailed characterization of B. pertussis isolates not expressing Pertussis Toxin or Pertactin. Vaccine, 2009;27(43):6034–6041.
29. Lam C, Octavia S., Rifacort L, et al. Rapid increase in pertactin-deficient Bordetella pertussis isolates, Australia. Emerg Infect Dis, 2014;20(4):626–633.
30. Otsuka N, Han HJ, Toyoizumi–Ajisaka H, et al. Prevalence and genetic characterization of pertactin – deficient Bordetella pertussis in Japan. PLoS One, 2012;7(2):e31985 [online]. [cit. 2012–02–14]. Dostupný na www: http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0031985.
31. Kallonen T, He Q. Bordetella pertussis strain variation and evolution postvaccination. Expert Rev Vaccines, 2009;8(7):863–875.
32. King Aj, van der Lee S, Mohangoo A, et al. Genome-wide gene expression analysis of Bordetella pertussis isolates associated with a resurgence in pertussis: elucidation of factors involved in the increased fitness of epidemic strains. PLoS One, 2013;8(6):e66150 [online]. [cit. 2013–06–11]. Dostupný na www: http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0066150.
33. Sealey KL, Harris SR, Fry NK, et al. Genomic analysis of isolates from the United Kingdom 2012 pertussis outbreak reveals that vaccine antigen genes are unusually fast evolving. J Infect Dis, 2014; pii: jiu665 [online]. [cit. 2014–12–08]. Dostupný na www: http://jid.oxfordjournals.org/content/early/2015/01/06/infdis.jiu665.long.
34. Berbers GA, de Greeff S, Mooi FR. Improving pertussis vaccination. Hum Vaccin, 2009;5(7):497–503.
35. Mooi FR, van Oirschot H, Heuvelman K, et al. Polymorphism in the Bordetella pertussis virulence factors P.69/pertactin and pertussis toxin in The Netherlands: temporal trends and evidence for vaccine-driven evolution. Infect Immun, 1998;66(2):670–675.
36. Mooi FR, He Q, van Oirschot H, et al. Variation in the Bordetella pertussis virulence factors pertussis toxin and pertactin in vaccine strains and clinical isolates in Finland. Infect Immun, 1999;67(6):3133–3134.
37. Fry NK, Neal S, Harrison TG, et al. Genotypic variation in the Bordetella pertussis virulence factors pertactin and pertussis toxin in historical and recent clinical isolates in the United Kingdom. Infect Immun, 2001;69(9):5520–5528.
38. Hallander HO, Advani A, Donelly D, et al. Shifts of Bordetella pertussis variants in Sweden from 1970 to 2003, during three periods marked by different vaccination programs. J Clin Microbiol, 2005;43(6):2856–2865.
39. Bottero D, Gaillard ME, Fingermann M, et al. Pulsed-field gel electrophoresis, pertactin, pertussis toxin S1 subunit polymorphisms, and surfaceome analysis of vaccine and clinical Bordetella pertussis strains. Clin Vaccine Immunol, 2007;14(11):1490–1498.
40. Gzyl A, Augustynowicz E, van Loo I, et al. Temporal nucleotide changes in pertactin and pertussis toxin genes in Bordetella pertussis strains isolated from clinical cases in Poland. Vaccine, 2001;20(3–4):299–303.
41. Packard ER, Parton R, Coote JG, et al. Sequence variation and conservation in virulence-related genes of Bordetella pertussis isolates from the UK. J Med Microbiol, 2004;53(5):355–365.
42. Borisova O, Kombarova SY, Zakharova NS, et al. Antigenic divergence between Bordetella pertussis clinical isolates from Moscow, Russia, and vaccine strains. Clin Vaccine Immunol, 2007;14(3):234–238.
43. Cherry JD, Gornbein J, Heininger U, et al. A search for serologic correlates of immunity to Bordetella pertussis cough illnesses. Vaccine, 1998;16(20):1901–1906.
44. Taranger J, Trollfors B, Lagergård T, et al. Correlation between pertussis toxin IgG antibodies in postvaccination sera and subsequent protection against pertussis. J Infect Dis, 2000;181(3):1010–1013.
45. Advani A, Van der Heide HG, Hallander HO, et al. Analysis of Swedish Bordetella pertussis isolates with three typing methods: characterization of an epidemic lineage. J Microbiol Methods, 2009;78(3):297–301.
46. Diavatopoulos DA, Cummings CA, Schouls LM et al. Bordetella pertussis, the causative agent of whooping cough, evolved from a distinct, human-associated lineage of B. bronchiseptica. PLoS Pathog, 2005;1(4):e45 [online]. [cit. 2014–10–02]. Dostupný na www: http://journals.plos.org/plospathogens/article?id=10.1371/journal.ppat.0010045.
47. Bart MJ, Harris SR, Advani A, et al. Global population structure and evolution of Bordetella pertussis and their relationship with vaccination. Mbio, 2014;5(2):e01074 [online]. [cit. 2014–12–20]. Dostupný na www: http://mbio.asm.org/content/5/2/e01074-14.full.pdf+html.
48. Weber C, Boursaux-Eude C, Coralie G, et al. Polymorphism of Bordetella pertussis isolates circulating for the last 10 years in France, where a single effective whole-cell vaccine has been used for more than 30 years. J Clin Microbiol, 2001;39(12):4396–4403.
49. van Amersfoorth SC, Schouls LM, van der Heide HG et al. Analysis of Bordetella pertussis populations in European countries with different vaccination policies. J Clin Microbiol, 2005;43(6):2837–2843.
50. Octavia S, Maharjan RP, Sintchenko V, et al. Insight into evolution of Bordetella pertussis from comparative genomic analysis: evidence of vaccine-driven selection. Mol Biol Evol, 2011;28(1):707–715.
51. Tsang RS, Lau AK, Sill ML, et al. Polymorphisms of the fimbria fim3 gene of Bordetella pertussis strains isolated in Canada. J Clin Microbiol, 2004;42(11):5364–5367.
52. King AJ, van Gorkom T, Pennings JL, et al. Comparative genomic profiling of Dutch clinical Bordetella pertussis isolates using DNA microarrays: identification of genes absent from epidemic strains. BMC Genomics, 2008;9 : 311 [online]. [cit. 2012–03–25]. Dostupný na www: http://www.biomedcentral.com/1471-2164/9/311.
53. King AJ, van Gorkom T, van der Heide HG, et al. Changes in the genomic content of circulating Bordetella pertussis strains isolated from the Netherlands, Sweden, Japan and Australia: adaptive evolution or drift? BMC Genomics, 2010;11 : 64 [online]. [cit. 2012–03–25]. Dostupný na www: http://www.biomedcentral.com/1471-2164/11/64.
Štítky
Hygiena a epidemiológia Infekčné lekárstvo Mikrobiológia
Článek Nové knihy
Článok vyšiel v časopiseEpidemiologie, mikrobiologie, imunologie
Najčítanejšie tento týždeň
2015 Číslo 3- Parazitičtí červi v terapii Crohnovy choroby a dalších zánětlivých autoimunitních onemocnění
- Očkování proti virové hemoragické horečce Ebola experimentální vakcínou rVSVDG-ZEBOV-GP
- Koronavirus hýbe světem: Víte jak se chránit a jak postupovat v případě podezření?
-
Všetky články tohto čísla
- Lidská prionová onemocnění v České republice
- Genetický a molekulový podklad vývoja autoimunitného diabetes mellitus
- Antigenní variabilita kmenů Bordetella pertussis izolovaných v letech 1967–2010 v České republice – možné vysvětlení vzestupu nemocnosti pertusí?
- Vyhodnocení epidemie spalniček v Ústeckém kraji
- Klonální analýza populace meningokoků z invazivního onemocnění a od zdravých nosičů izolovaných v České republice v období 1971 – 2014 (květen)
- Kampylobakteriózy na Klinice infekčních chorob Fakultní nemocnice Brno v letech 2011–2013: retrospektivní studie
- Klinická rezistence lidského cytomegaloviru při léčbě gancyklovirem u pacientů po alogenní transplantaci hematopoetických buněk – zkušenosti jednoho centra
- Hodnocení tvorby biofilmu vybraných patogenů vyskytujících se v potravinářském průmyslu
- Nové knihy
- Epidemiologie, mikrobiologie, imunologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Lidská prionová onemocnění v České republice
- Klinická rezistence lidského cytomegaloviru při léčbě gancyklovirem u pacientů po alogenní transplantaci hematopoetických buněk – zkušenosti jednoho centra
- Genetický a molekulový podklad vývoja autoimunitného diabetes mellitus
- Hodnocení tvorby biofilmu vybraných patogenů vyskytujících se v potravinářském průmyslu
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



