-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Genetické vyšetření u pacientů s hypertrofickou kardiomyopatií
Genetic testing in patients with hypertrophic cardiomyopathy
Hypertrophic cardiomyopathy (HCM) is a frequent inherited heart disease with an estimated prevalence of up to 1 in 200 individuals. In the majority of cases, HCM is inherited as an autosomal dominant genetic trait with pathogenic variants detected in sarcomeric proteins. Nevertheless, the genetic basis of HCM is more complex. More than 2 000 mutations in 27 genes have been described in association with HCM. The genetic and allelic heterogeneity makes molecular analysis by conventional methods time-consuming and expensive. Next Generation Sequencing (NGS) based genomic testing allows rapid analysis of a large number of genes or even a whole genome at similar cost and accuracy to conventional sequencing methods. Screening large numbers of genes results in the identification of many genetic variants of uncertain significance and makes the results interpretation difficult. The process of classification the genetic variants is very complex. Genetic screening is an important tool for clinical management of HCM patients and enables mutation-specific confirmatory testing of the appropriate family members. The yield of genetic testing is variable, about 20–40 %. For predicting the diagnostic yield of genetic testing, clinical scoring systems have been developed. Identifying the genetic basis of HCM creates remarkable opportunities to understand how disease develops, and by extension, how to disrupt the disease progression in the future.
Keywords:
Genetics – next generation sequencing – genetic testing – genotype – hypertrophic cardiomyopathy
Autoři: MUDr. Jiří Bonaventura, Ph.D.; Josef Veselka
Působiště autorů: Kardiologická klinika 2. LF UK a FN Motol, Praha
Vyšlo v časopise: Vnitř Lék 2019; 65(10): 652-658
Kategorie: Přehledné referáty
Souhrn
Hypertrofická kardiomyopatie (Hypertrophic CardioMyopathy – HCM) se řadí mezi nejčastější hereditární srdeční choroby. V klasické podobě je popisována jako onemocnění způsobené mutací genů pro sarkomerické proteiny s autosomálně dominantní dědičností. Genetika HCM je však mnohem složitější. V současné době je popsáno minimálně 27 genů asociovaných s HCM a více než 2 000 kauzálních mutací. Onemocnění HCM se vyznačuje velkou fenotypovou a genotypovou heterogenitou. Tato heterogenita, společně s neúplnou penetrancí a variabilní expresivitou, komplikuje interpretaci genetického vyšetření. Současná klasifikace nalezených genetických variant je velmi komplexní a vyžaduje spolupráci kardiologa, klinického genetika a molekulárního biologa. Genetické vyšetření je doporučováno u všech pacientů s HCM pro kaskádovitý screening příbuzných. Diagnostická výtěžnost vyšetření je však pouze 20–40 %. K lepší selekci pacientů indikovaných ke genetickému vyšetření pomáhají skórovací systémy predikující jeho výtěžnost. Budoucností genetického vyšetřování je identifikace rizikových jedinců v preklinickém stadiu a plné pochopení molekulární patogeneze onemocnění s možností cíleného ovlivnění jeho vývoje.
Klíčová slova:
genetika – sekvenování nové generace – genetické vyšetření – genotyp – hypertrofická kardiomyopatie
Historie
Na základě genetických studií z 80. a 90. let minulého století byly objeveny mutace v genech kódujících proteiny sarkomery asociované s těžkou hypertrofií LK. V roce 1990 byla popsána první mutace v genu pro těžký řetězec β-myozinu (MYH7) zodpovědná za vznik HCM [6]. Během následující dekády pak byly postupně reportovány další geny asociované se vznikem onemocnění [7]. Jejich stručný přehled uvádíme v tab. 1 a tab. 2. V současné době, tedy téměř 30 let od publikace první kauzální mutace MYH7, je popsáno více než 2 000 mutací a geny asociované s HCM postupně přibývají [8–11]. Databáze Online Mendelian Inheritance in Man (OMIM) uvádí aktuálně 27 kauzálních genů [12]. Ze sarkomerických genů je 8 považováno za silně asociované s HCM. Jedná se o geny MYH7, MYBPC3, TNNI3, TNNT2, TPM1, ACTC1, MYL2, MYL3 [1,13], které jsou blíže popsány v tab. 1. Toto spektrum sarkomerických genů bylo postupem času rozšířeno i na nesarkomerické geny kódující např. desmosomální proteiny či iontové kanály [14,15]. Ačkoli je HCM je především onemocněním sarkomerických proteinů, genetický podklad je zřejmě složitější. Mutace genů pro sarkomerické proteiny jsou totiž kromě HCM dále asociovány s dilatační kardiomyopatií (DCM), restriktivní kardiomyopatií (RCM) i nonkompaktní kardiomyopatií (LVNC) [16,17].
Tab. 1. Geny asociované s hypertrofickou kardiomyopatií 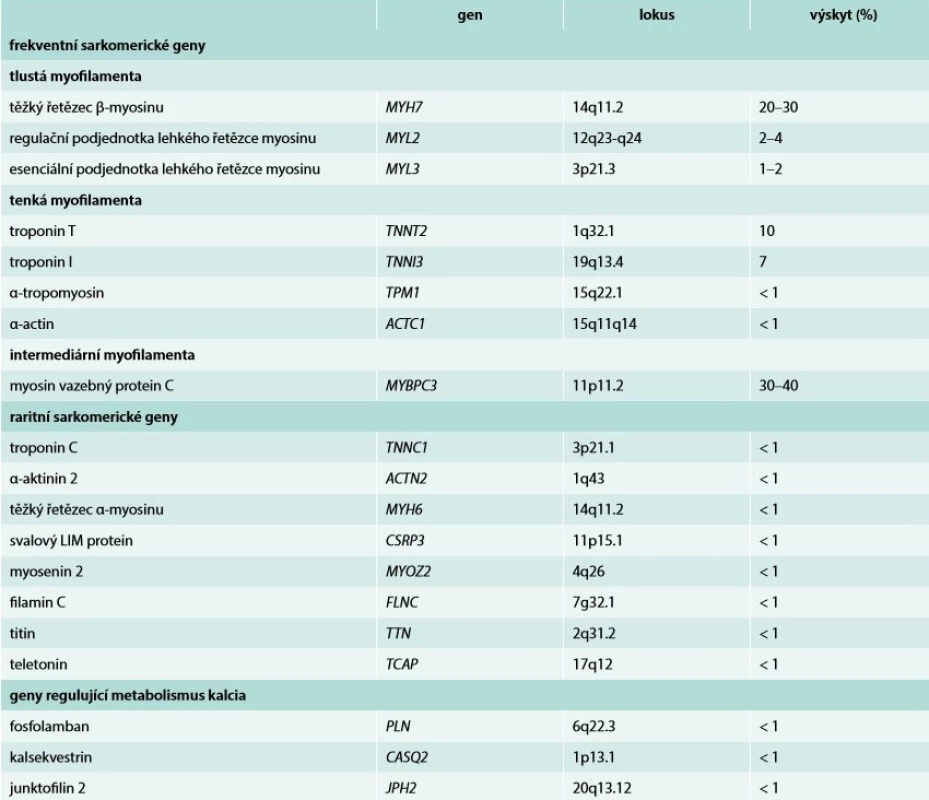
Tab. 2. Ostatní geny a syndromy asociované s hypertrofickou kardiomyopatií 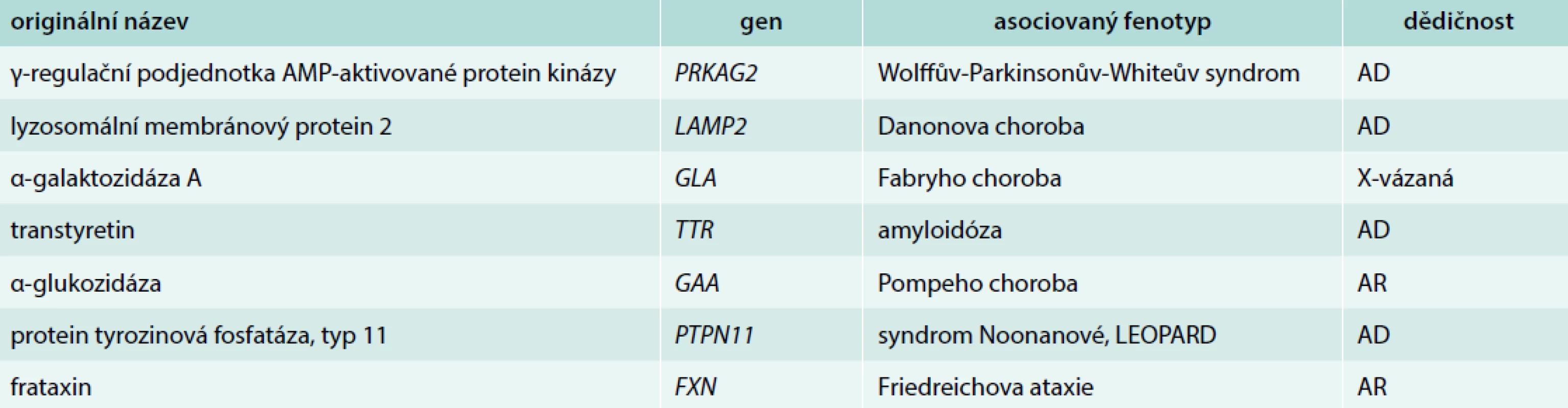
Sekvenování nové generace
Pro pacienty s HCM je charakteristická velká fenotypová i genotypová heterogenita. Heterogenita HCM komplikuje genetické vyšetření jak z hlediska časové, tak i finanční náročnosti [18,19]. Dříve bylo molekulárně genetické vyšetření možné pouze ve výzkumných centrech, byly vyšetřovány rodiny se zjevnou dědičnou HCM a nákladné vyšetření DNA zahrnovalo pouze několik genů s nejsilnější asociací s HCM. V posledních 2 dekádách byla vyvinuta řada metod označovaných jako tzv. sekvenování nové generace (Next Generation Sequencing – NGS). Jedná se o vysokokapacitní metody, které v porovnání s klasickými metodami sekvenování (např. Sangerova metoda) umožňují analýzu velkého množství genetického materiálu rychleji a levněji. NGS využívá principu paralelizace procesu sekvenování, umožňuje sekvenování tisíců až milionů vláken DNA současně. Kromě klasických vyšetření genetické variability, mutační analýzy konkrétních genů a kvantifikace jednotlivých alel je pomocí NGS možné vyšetřit celý exom (Whole Exome Sequencing – WES), případně provést i celogenomové sekvenování (Whole Genome Sequencing – WGS). Výsledkem NGS je produkce obrovského množství výstupních dat s následnou potřebou data utřídit a analyzovat. Vyšetřování velkého množství genů má totiž za následek nálezy velkého množství tzv. variant nejasného významu (Variants of Uncertain Significance – VUS) [19,20] a ztěžuje tak interpretaci výsledku genetické analýzy, jak bude popsáno dále v textu. Výhodou NGS je naopak díky relativně nízké ceně a rychlosti vyšetření jeho postupné rozšíření do klinické praxe.
Klasifikace genetických variant
Pro klinické využití molekulárně-genetického vyšetření má zásadní význam klasifikace nalezených variant. Vzhledem k velkému množství výstupních dat je v současnosti používán kombinovaný přístup, jehož podkladem jsou následující údaje:
- frekvence variant v kontrolní populaci s využitím mezinárodních databází (např. 1 000 Genomes Project, Exome Sequencing Project, Exome Aggregation Consortium) [21–23]
- publikované varianty asociované s onemocněním (např. Clinvar, Human Gene Mutation Database) [24,25]
- in silico klasifikace s užitím softwaru (např. Polyphen 2, Sorting Intolerant From Tolerant) predikujícího možný dopad mutace na strukturu a funkci výsledného proteinu
- mutace v tzv. evolučně vysoce konzervovaných funkčních doménách proteinů
- segregační analýzy genotypu s fenotypem v postižených rodinách (silná evidence)
- funkční studie na animálních modelech či in vitro (nákladné, složité)
V roce 2015 byla publikována doporučení pro klasifikaci genetických variant Americkou společností pro lékařskou genetiku a genomiku (American College of Medical Genetics and Genomics – ACMG) [26], která vychází z výše uvedených principů. Tato klasifikace dělí nalezené varianty do 5 následujících tříd:
- benigní
- pravděpodobně benigní
- VUS – varianta nejasného významu
- pravděpodobně patogenní
- patogenní
Genetický screening
Genetický screening má v péči o pacienty s HCM a jejich příbuzné významné postavení. Standardním postupem je získání detailní rodinné anamnézy (alespoň 3 generace) a molekulárně-genetické vyšetření probanda se zaměřením na minimálně všechny známé geny asociované s HCM dle databáze OMIM [12]. Pokud je klinické podezření na specifickou příčinu či HCM v rámci komplexního syndromu, pak je vhodné panel rozšířit (tab. 2).
V případě pozitivního nálezu poté provedeme specifické vyšetření prvostupňového příbuzného na konkrétní gen a mutaci již nalezenou u probanda. Pokud je i u příbuzného zachycena patogenní mutace, je možné kaskádovité vyšetření dalších příbuzných (vzhledem k převažující AD dědičnosti). Detailní rodinná anamnéza a sestavení rodokmenu nám pomůže odhalit pravděpodobně dědičnou příčinu onemocnění a obvykle stanovit i typ dědičnosti. Je možná genetická analýza post mortem tkáňových vzorků s kaskádovitým screeningem příbuzných. Relativně častým nálezem u HCM rodin jsou tzv. privátní mutace, tedy takové, které jsou specifické pro konkrétní rodinu nebo malou populaci pacientů. Hlavní klinickou výhodou je, pokud specifická příčinná mutace u probanda není nalezena u prvostupňového příbuzného. Příbuzného je pak možno vyřadit z dispenzarizace, pravděpodobnost onemocnění je nízká, nicméně de novo mutace jsou samozřejmě možné. Vždy proto v případě symptomatologie upozorňujeme pacienty na nutnost návštěvy lékaře. Vyšetření dětí je podle současných doporučení vhodné okolo 10. roku věku [1]. Hranice byla stanovena na základě pediatrických studií, které ukázaly raritní výskyt závažných komplikací HCM před nástupem puberty [27,28].
Pokud je molekulárně genetické vyšetření u probanda negativní, pokračujeme v zavedeném pravidelném klinickém sledování prvostupňových příbuzných dle aktuálních doporučení. U HCM se jedná o echokardiografické vyšetření, 12svodové EKG, holterovskou EKG monitoraci, bicyklovou ergometrii, u vybraných pacientů (obvykle při nedostatečné echokardiografické vyšetřitelnosti) zobrazení srdce magnetickou rezonancí, případně u mladých pacientů s časným nástupem onemocnění screening metabolických chorob [1,29].
Opačnou klinickou situací je klinicky negativní fenotyp příbuzného s nálezem patogenní mutace. Na rozdíl od DCM, při níž je např. mutace v genu LMNA spojena s nepříznivou prognózou a je dokonce dle ESC guidelines součástí indikace k implantaci ICD [30], u HCM nemáme k dispozici dostatek dat o vývoji onemocnění u asymptomatických nosičů mutací bez klinicky vyjádřeného fenotypu. Riziko náhlé srdeční smrti je u jedinců bez vyjádřené hypertrofie obecně nízké. Výjimkou mohou být mutace v TNNT2, jak naznačují některé publikace [31,32], nicméně se nejedná o silné důkazy. Není jednoznačné, zda pouze na základě pozitivního genotypu bez vyjádřeného fenotypu (G+/F-) činit specifická doporučení a navrhovat omezení, např. u profesionálních atletů [10,33,34]. Většina (G+/F-) pacientů má totiž pravděpodobně příznivou prognózu [1,34–36]. Vzhledem k variabilní penetranci, která je věkově vázaná (55 % do 30 let věku až po 95 % nad 50 let věku [37]), je však nutno pokračovat v pravidelném klinickém sledování těchto jedinců [38].
Obecně vzato, genetické vyšetření je vhodné u všech HCM pacientů s cílem umožnit kaskádovitý screening příbuzných probanda [1]. Jeho hlavní přínos tedy obvykle nespočívá ve zpřesnění diagnostiky HCM (např. u atletického srdce či arteriální hypertenze), protože negativní výsledek neznamená vyloučení diagnózy HCM. Pokud jsou však přítomny neobvyklé symptomy či známky suspektní ze specifické příčiny (např. syndromy, metabolická onemocnění), lze genetické vyšetření využít ke stanovení definitivní diagnózy. Nezbytnou podmínkou je provádění v expertních centrech se zkušenostmi s molekulárně-genetickým vyšetřováním a jeho klinickou interpretací. Mezioborová spolupráce je nevyhnutelná. Bez ohledu na použitou metodu by měla genetická analýza vždy zahrnovat nejčastější sarkomerické geny (tab. 1). V případě příznaků ukazujících na možné vzácnější příčiny je nutné vyšetřit i další geny (tab. 2).
Diagnostická výtěžnost genetického vyšetření
Diagnostická výtěžnost genetického vyšetření (tj. záchyt patogenní mutace) je velmi variabilní a relativně nízká. Pravděpodobnost záchytu patogenní mutace je obecně vyšší u mladších pacientů a pacientů s pozitivní rodinnou anamnézou (může dosahovat 50–60 %) [1]. U ostatních pacientů se pohybuje maximálně okolo 30–40 % [3] a se zavedením přísnějších klasifikačních kritérií ACMG je záchyt ještě nižší [26,39,40]. Vysoká senzitivita genetického testování s nástupem metod NGS tak byla postupně vystřídána vyšší specificitou související s aplikací komplexních klasifikačních kritérií. Ukazuje se, že genetická data a klasifikaci patogenních mutací z minulých dekád bude vhodné revidovat v kontextu současných poznatků [26].
Relativně nízká výtěžnost genetického vyšetření si vyžádala vznik klinických skórovacích systémů pro predikci jeho pozitivity. Nejznámějším a pravděpodobně nejpřesnějším je tzv. Mayo HCM Genotype Predictor score (Mayo Score), který je založen na základních klinických a echokardiografických parametrech a byl poprvé publikován v roce 2014 [41]. Mezi prediktory výtěžnosti genetického vyšetření patří: věk < 45 let, tloušťka stěny levé komory ≥ 20 mm, rodinná anamnéza HCM, rodinná anamnéza náhlé srdeční smrti a tzv. reverzní (katenoidní) tvar septa. Negativním prediktorem je pak přítomnost arteriální hypertenze. V původní kohortě pacientů dosahovala celková výtěžnost genetického vyšetření 34 % při vyšetřování 9 genů pro sarkomerické proteiny (ACTC1, MYBPC3, MYH7, MYL2, MYL3, TNNC1, TNNI3, TNNT2, TPM1) [41]. S rozvojem NGS se v současné době použité panely genů mohou výrazně lišit v počtu a typu jednotlivých genů v závislosti na lokální dostupnosti a finančních možnostech pracovišť.
Na Kardiologické klinice 2. LF UK a FN Motol, Praha byla provedena genotypizace rozsáhlého souboru českých pacientů s HCM a validace Mayo Score za použití nejnovějších klasifikačních kritérií patogenních variant dle ACMG [40]. V naší práci jsme použili ke genetickému vyšetření panely obsahující až 229 genů asociovaných se srdečními onemocněními [40], které byly vytvořeny na zakázku. Všechny naše panely genů samozřejmě obsahovaly i 9 genů vyšetřovaných v původní Mayo Score publikaci. Celková výtěžnost genetického vyšetření činila pouhých 21 % [40]. Tuto nízkou výtěžnost si vysvětlujeme použitím přísných ACMG kritérií [26], která v době publikace Mayo Score práce nebyla k dispozici. V roce 2016 autoři Mayo Score publikovali validační studii své původní práce [42]. Ve validační studii byl genetický test považován za pozitivní (G+), pokud byla genetická varianta klasifikována jako patogenní, pravděpodobně patogenní nebo VUS [42]. Pokud bychom stejný postup (tj. zahrnutí VUS do pozitivního vyšetření) použili i v našem souboru HCM-pacientů, výtěžnost genetického vyšetření by stoupla na 42 % [40]. Ve všech zmiňovaných případech Mayo Score konzistentně predikuje genetickou výtěžnost. Jeho použití se jeví v klinické praxi, v níž je patrna limitace finančními prostředky, jako racionální. Vzhledem k časové náročnosti molekulárně-genetického vyšetření (obvykle měsíce) tak lze pečlivou selekcí pacientů zamezit velkému množství negativních výsledků a nadužívání metody NGS.
Vztah fenotypu a genotypu
Genetická heterogenita HCM je obrovská [8–10]. Většina dosud publikovaných genů asociovaných s HCM kóduje sarkomerické proteiny [13,43]. Pouze relativně malá část identifikovaných mutací je klasifikována jako patogenní či pravděpodobně patogenní, některé z nich jsou tzv. privátní mutace a tedy není možná korelace genotyp-fenotyp na větším souboru pacientů [9,10,26]. S dostupností NGS můžeme vyšetřovat stále větší množství genů. Výsledkem těchto vyšetření pravděpodobně není větší záchyt patogenních mutací, ale záchyt většího množství VUS, jejichž interpretace je na základě dostupných dat často svízelná a k jejich definitivnímu zařazení bude potřeba více korelovaných genotypů s fenotypy a provedení segregačních analýz [19,20]. Užitečnou veřejně dostupnou databází pro reportování mutací a jejich vztahu k fenotypu je např. ClinVar [24].
Data pro vztahy genotyp-fenotyp jsou zatím nedostatečná. Některé práce ukazují, že nález patogenní mutace (G+) u HCM pacientů zhoršuje kardiovaskulární mortalitu, zvyšuje riziko cévní mozkové příhody a progrese symptomatologie ve třídě NYHA ve srovnání s genotyp-negativními (G-) pacienty [31]. Většina publikovaných dat o vztahu konkrétních mutovaných genů a závažnosti fenotypu je na relativně malém počtu pacientů z jednoho centra. V dětském věku byla popsána asociace mutace MYH7 s časným nástupem onemocnění a rizikem kardiovaskulárních příhod (Major Adverse Cardiac Events – MACE) [44]. Recentně (prosinec roku 2018) publikovaná data z portugalského registru dospělých HCM-pacientů naznačují asociaci mutace ve stejném genu se systolickou dysfunkcí LK [45]. V tomto relativně rozsáhlém registru nemocných s HCM (528 pacientů bylo geneticky vyšetřeno) byl nález jakékoli patogenní mutace (G+) asociován s vyšším rizikem náhlé srdeční smrti [45].
Dalším důležitým faktorem v interpretaci nálezů je neúplná penetrance a variabilní expresivita, která je pro HCM typická. Není výjimkou, že naprosto identické mutace stejných genů i ve stejné rodině vedou ke zcela odlišnému fenotypu. To naznačuje, že roli v dlouhodobém klinickém průběhu onemocnění mají i jiné faktory než pouze mutace na úrovni sarkomerických proteinů. Tyto další genetické a epigenetické faktory, stejně jako modifikace environmentální, pravděpodobně hrají důležitou roli, kterou zatím neumíme uspokojivě charakterizovat. Pokud tedy hovoříme o HCM jako onemocnění s autosomálně dominantní mendelovskou dědičností, jedná se o výraznou (a nejspíše nepravdivou) simplifikaci [46–48]. Mezi genetické modifikátory se řadí obvykle metylace a acetylace DNA, v poslední době se diskutuje význam miRNA [11]. Je možné, že nově objevené varianty nesarkomerických genů, např. pro iontové kanály, desmosomy či titin, nejsou primární příčinou HCM, nýbrž právě zmiňovanými modifikátory [14,49].
V klinické praxi, vzhledem k její prevalenci, je častá koincidence HCM s arteriální hypertenzí. Hemodynamický stav při arteriální hypertenzi nutně modifikuje fenotyp HCM. Setkáme se též s pacienty s těžkou aortální stenózou, konkomitantní arteriální hypertenzí a HCM. Fyzická zátěž, životní styl, a kromě arteriální hypertenze i další komorbidity jako např. diabetes mellitus, obezita a chronická renální insuficience vedou nutně též k modifikaci výsledného fenotypu HCM. Obezita a arteriální hypertenze jsou asociovány s větším objemem myokardu LK. Obezita je dále asociována s arteriální hypertenzí a rovněž i diabetes mellitus, tedy je v klinické praxi často nemožné rozlišit, co je skutečnou příčinou či hlavním modifikátorem hypertrofie LK. V současné době je velmi zkoumaným onemocněním tzv. senilní (wild-type transtyretinová) amyloidóza, u které se předpokládá relativně vysoká prevalence a poddiagnostikování choroby v běžné populaci [50]. V terénu všech výše uvedených komorbidit je stanovení etiologie hypertrofie LK svízelné. Nález VUS při genetickém vyšetření, do kterého jsme předtestově vkládali neadekvátní očekávání, nás v diagnostice příliš neposune. Hlavním klinickým přínosem genetického vyšetření tedy nadále zůstává možnost kaskádovitého vyšetřování příbuzných (v případě VUS) a provedení segregačních analýz, nikoli „zpřesňování“ diagnostiky probanda. Výsledek genetického vyšetření v populaci příbuzných má zásadní vliv na další sledování, méně často vede k diagnostice před klinickou manifestací choroby. Znovu je třeba zopakovat, že nález patogenní mutace nutně nemusí znamenat rozvoj HCM. V populaci mladých sportovců zůstává zásadním klinickým nástrojem pro odlišení sportovního srdce od HCM echokardiografické a EKG vyšetření [51].
Budoucnost
Objasnění genetické příčiny srdečních onemocnění otevírá příležitost plně pochopit patogenezi onemocnění a v budoucnu i uvažovat o jeho specifické léčbě. Dlouhodobým cílem genetického vyšetřování je identifikace rizikových jedinců ještě před klinickou manifestací choroby. Pokud budeme schopni charakterizovat jasnou příčinu na úrovni molekulárně-genetické, kromě pochopení patogeneze budeme moci uvažovat o cílené modifikaci těchto příčin. V experimentálních studiích na myších již byly některé mutantní alely „utlumeny“ pomocí interferující RNA [52]. Tyto „mutation-silencing therapies“ pravděpodobně nejsou efektivní v regresi již rozvinuté hypertrofie a k zamezení rozvoje onemocnění by bylo potřeba je aplikovat ve velmi časných stadiích života jedince [53].
Recentní rychlý rozvoj technik tzv. editace genomu a jejich aplikace na animálních modelech slibuje nadějné možnosti modifikace i humánních zárodečných mutací. Nejvíce diskutovaný je v současnosti (i z etického pohledu) systém CRISPR-Cas9. Jedná se o užitečný nástroj pro rozpoznávání specifických sekvencí genomu s následnou indukcí tzv. double-strand breaks (DSBs) [54]. V srpnu roku 2017 byla v časopisu Nature publikována „oprava“ zárodečné mutace v genu MYBPC3 u lidského embrya pomocí systému CRISPR-Cas9 s následnou indukcí endogenní DNA reparace v preimplantační diagnostice [55]. Z etického hlediska je možnost editace lidské zárodečné linie přinejmenším kontroverzní. Zvláště u HCM, s přihlédnutím k neúplné penetranci a variabilní expresivitě, bude od preimplantační či jiné prenatální diagnostiky např. k umělému přerušení těhotenství jistě komplikovaná cesta. Nález patogenní mutace i jinak zdravého embrya či plodu totiž nemusí znamenat rozvoj onemocnění během života a i v případě rozvoje HCM je klinický průběh variabilní a prognóza je u většiny pacientů téměř srovnatelná s běžnou populací [36].
Závěr
Pro HCM je charakteristická výrazná nejen fenotypová, ale i genotypová variabilita. Při genetickém vyšetření současnými metodami NGS u více než poloviny pacientů nezachytíme žádnou významnou mutaci [40]. S moderními metodami přichází možnost vyšetření velkého množství genů, včetně WES i WGS. Objem těchto dat, zejména interpretace VUS, si vyžaduje úzkou spolupráci kardiologa, molekulárního biologa a klinického genetika. Faktory ovlivňující rozvoj HCM u genotyp negativních pacientů (G-/F+) i asymptomatických nosičů mutací (G+/F-) dosud nejsou uspokojivě vysvětleny. K plnému porozumění vztahu genotyp-fenotyp, vlivu genetického pozadí a komorbidit na rozvoj a průběh onemocnění by bylo potřeba rozsáhlých genetických studií na velkých souborech příbuzných pacientů. I přes relativně vysokou prevalenci onemocnění toho bude možno dosáhnout pouze mezinárodní spoluprací velkých center, standardizace podrobného genetického vyšetřování a mezioborovou spoluprací kardiologů, klinických genetiků a molekulárních biologů.
Podpořeno z programového projektu Ministerstva zdravotnictví ČR s reg. č. 15–34904A.
Doručeno do redakce 29. 3. 2019
Přijato po recenzi 23. 9. 2019
MUDr. Jiří Bonaventura
Kardiologická klinika 2. LF UK a FN Motol, Praha
Zdroje
- Elliott PM, Anastasakis A, Borger M et al. 2014 ESC Guidelines on diagnosis and management of hypertrophic cardiomyopathy. Eur Heart J 2014; 35(39): 2733–2779. Dostupné z DOI: <http://dx.doi.org/10.1093/eurheartj/ehu284>.
- Veselka J, Anavekar NS, Charron P. Hypertrophic obstructive cardiomyopathy. Lancet 2017; 389(10075): 1253–1267. Dostupné z DOI: <http://dx.doi.org/10.1016/S0140–6736(16)31321–6>.
- Richard P, Charron P, Carrier L et al. Hypertrophic cardiomyopathy: Distribution of disease genes, spectrum of mutations, and implications for a molecular diagnosis strategy. Circulation 2003;107(17): 2227–2232. Dostupné z DOI: <http://dx.doi.org/10.1161/01.CIR.0000066323.15244.54>. Erratum in Circulation 2004;109(25):3258.
- Maron BJ, Doerer JJ, Haas TS et al. Sudden Deaths in Young Competitive Athletes: Analysis of 1866 Deaths in the United States, 1980–2006. Circulation 2009; 119(8): 1085–1092. Dostupné z DOI: <http://dx.doi.org/10.1161/CIRCULATIONAHA.108.804617>.
- Semsarian C, Ingles J, Maron MS et al. New perspectives on the prevalence of hypertrophic cardiomyopathy. J Am Coll Cardiol 2015; 65(12): 1249–1254. Dostupné z DOI: <http://dx.doi.org/10.1016/j.jacc.2015.01.019>.
- Geisterfer-Lowrance AA, Kass S, Tanigawa G et al. A molecular basis for familial hypertrophic cardiomyopathy: a beta cardiac myosin heavy chain gene missense mutation. Cell 1990; 62(5): 999–1006. Dostupné z DOI: <http://dx.doi.org/10.1016/0092–8674(90)90274-i>.
- Ho CY, Charron P, Richard P et al. Genetic advances in sarcomeric cardiomyopathies: State of the art. Cardiovasc Res 2015; 105(4): 397–408. Dostupné z DOI: <http://dx.doi.org/10.1093/cvr/cvv025>.
- Ingles J, Burns C, Barratt A et al. Application of Genetic Testing in Hypertrophic Cardiomyopathy for Preclinical Disease Detection. Circ Cardiovasc Genet 2015; 8(6): 852–859. Dostupné z DOI: <http://dx.doi.org/10.1161/CIRCGENETICS.115.001093>.
- Alfares AA, Kelly MA, McDermott G et al. Results of clinical genetic testing of 2,912 probands with hypertrophic cardiomyopathy: Expanded panels offer limited additional sensitivity. Genet Med 2015; 17(11): 880–888. Dostupné z DOI: <http://dx.doi.org/10.1038/gim.2014.205>.
- Maron BJ, Maron MS, Semsarian C. Genetics of Hypertrophic Cardiomyopathy After 20 Years: Clinical Perspectives. J Am Coll Cardiol 2012; 60(8): 705–715. Dostupné z DOI: <http://dx.doi.org/10.1016/j.jacc.2012.02.068>.
- Sabater-Molina M, Pérez-Sánchez I, Hernández del Rincón JP et al. Genetics of hypertrophic cardiomyopathy: A review of current state. Clin Genet 2018; 93(1): 3–14. Dostupné z DOI: <http://dx.doi.org/10.1111/cge.13027>.
- Online Mendelian Inheritance in Man®. Dostupné z WWW: <https://www.omim.org/>.
- Gersh BJ, Maron BJ, Bonow RO et al. 2011 ACCF/AHA guideline for the diagnosis and treatment of hypertrophic cardiomyopathy: A report of the American College of cardiology foundation/American heart association task force on practice guidelines. Circulation 2011; 124(24): e783-e831. Dostupné z DOI: <http://dx.doi.org/10.1161/CIR.0b013e318223e2bd>.
- Lopes LR, Syrris P, Guttmann OP et al. Novel genotype-phenotype associations demonstrated by high-throughput sequencing in patients with hypertrophic cardiomyopathy. Heart 2015; 101(4): 294–301. Dostupné z DOI: <http://dx.doi.org/10.1136/heartjnl-2014–306387>.
- Walsh R, Buchan R, Wilk A et al. Defining the genetic architecture of hypertrophic cardiomyopathy: re-evaluating the role of non-sarcomeric genes. Eur Heart J 2017; 38(46): 3461–3468. Dostupné z DOI: <http://dx.doi.org/10.1093/eurheartj/ehw603>.
- McNally E, Dellefave L. Sarcomere mutations in cardiogenesis and ventricular noncompaction. Trends Cardiovasc Med 2009; 19(1): 17–21. Dostupné z DOI: <http://dx.doi.org/10.1016/j.tcm.2009.03.003>.
- Mogensen J, Kubo T, Duque M et al. Idiopathic restrictive cardiomyopathy is part of the clinical expression of cardiac troponin I mutations. J Clin Invest 2003; 111(2): 209–216. Dostupné z DOI: <http://dx.doi.org/10.1172/JCI16336>. Erratum in J Clin Invest 2003; 111(6): 925.
- Bortot B, Athanasakis E, Brun F et al. High-throughput Genotyping Robot-assisted Method for Mutation Detection in Patients With Hypertrophic Cardiomyopathy. Diagnostic Mol Pathol 2011; 20(3): 175–179. Dostupné z DOI: <http://dx.doi.org/10.1097/PDM.0b013e31820b34fb>.
- Fokstuen S, Munoz A, Melacini P et al. Rapid detection of genetic variants in hypertrophic cardiomyopathy by custom DNA resequencing array in clinical practice. J Med Genet 2011; 48(8): 572–576. Dostupné z DOI: <http://dx.doi.org/10.1136/jmg.2010.083345>.
- Meder B, Haas J, Keller A et al. Targeted Next-Generation Sequencing for the Molecular Genetic Diagnostics of Cardiomyopathies. Circ Cardiovasc Genet 2011; 4(2): 110–122. Dostupné z DOI: <http://dx.doi.org/10.1161/CIRCGENETICS.110.958322>.
- IGSR and the 1000 Genomes Project. Dostupné z WWW: <http://www.internationalgenome.org/>.
- NHLBI Exome Sequencing Project (ESP). Dostupné z WWW: <http://evs.gs.washington.edu/EVS/>.
- Exome Aggregation Consortium. Dostupné z WWW. <http://exac.broadinstitute.org/>.
- ClinVar. Dostupné z WWW: <https://www.ncbi.nlm.nih.gov/clinvar>.
- Human Gene Mutation Database. Dostupné z WWW: <http://www.hgmd.cf.ac.uk/ac/index.php>.
- Richards S, Aziz N, Bale S et al. Standards and guidelines for the interpretation of sequence variants: A joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med 2015; 17(5): 405–424. Dostupné z DOI: <http://dx.doi.org/10.1038/gim.2015.30>.
- Jensen MK, Havndrup O, Christiansen M et al. Penetrance of hypertrophic cardiomyopathy in children and adolescents: a 12-year follow-up study of clinical screening and predictive genetic testing. Circulation 2013; 127(1): 48–54. Dostupné z DOI: <http://dx.doi.org/10.1161/CIRCULATIONAHA.111.090514>.
- Cardoso B, Gomes I, Loureiro P et al. Clinical and genetic diagnosis of familial hypertrophic cardiomyopathy: Results in pediatric cardiology. Rev Port Cardiol 2017; 36(3): 155–165. Dostupné z DOI: <http://dx.doi.org/10.1016/j.repc.2016.09.009>.
- Hershberger RE, Givertz MM, Ho CY et al. Genetic Evaluation of Cardiomyopathy – A Heart Failure Society of America Practice Guideline. J Card Fail 2018; 24(5): 281–302. Dostupné z DOI: <http://dx.doi.org/10.1016/j.cardfail.2018.03.004>.
- Priori SG, Blomström-Lundqvist C, Mazzanti A et al. 2015 ESC Guidelines for the management of patients with ventricular arrhythmias and the prevention of sudden cardiac death. Europace 2015; 17(11): 1601–87. Dostupné z DOI: <http://dx.doi.org/10.1093/europace/euv319>.
- Olivotto I, Girolami F, Ackerman MJ et al. Myofilament protein gene mutation screening and outcome of patients with hypertrophic cardiomyopathy. Mayo Clin Proc 2008; 83(6): 630–638. Dostupné z DOI: <http://dx.doi.org/10.4065/83.6.630>.
- Varnava AM, Elliott PM, Baboonian C et al. Hypertrophic Cardiomyopathy. Circulation 2001; 104(12): 1380–1384. Dostupné z DOI: <http://dx.doi.org/10.1161/hc3701.095952>.
- Ho CY. Genetics and clinical destiny: improving care in hypertrophic cardiomyopathy. Circulation 2010; 122(23): 2430–2340; discussion 2440. Dostupné z DOI: <http://dx.doi.org/10.1161/CIRCULATIONAHA.110.978924>.
- Maron BJ, Yeates L, Semsarian C. Clinical challenges of genotype positive (+) phenotype negative (-) family members in hypertrophic cardiomyopathy. Am J Cardiol 2011; 107(4): 604–608. Dostupné z DOI: <http://dx.doi.org/10.1016/j.amjcard.2010.10.022>.
- Maron BJ, Maron MS, Semsarian C. Genetics of hypertrophic cardiomyopathy after 20 years: clinical perspectives. J Am Coll Cardiol 2012; 60(8): 705–715. Dostupné z DOI: <http://dx.doi.org/10.1016/j.jacc.2012.02.068>.
- Maron BJ. Clinical Course and Management of Hypertrophic Cardiomyopathy. N Engl J Med 2018; 379(7): 655–668. Dostupné z DOI: <http://dx.doi.org/10.1056/NEJMra1710575>.
- Charron P, Carrier L, Dubourg O et al. Penetrance of familial hypertrophic cardiomyopathy. Genet Couns 1997; 8(2): 107–114.
- Richard P, Charron P, Carrier L et al. Hypertrophic cardiomyopathy: Distribution of disease genes, spectrum of mutations, and implications for a molecular diagnosis strategy. Circulation 2003; 107(17): 2227–2232. Dostupné z DOI: <http://dx.doi.org/10.1161/01.CIR.0000066323.15244.54>. Erratum in Circulation 2004; 109(25): 3258.
- Amendola LM, Jarvik GP, Leo MC et al. Performance of ACMG-AMP Variant-Interpretation Guidelines among Nine Laboratories in the Clinical Sequencing Exploratory Research Consortium. Am J Hum Genet 2016; 98(6): 1067–1076. Dostupné z DOI: <http://dx.doi.org/10.1016/j.ajhg.2016.03.024>.
- Bonaventura J, Norambuena P, Tomašov P et al. The utility of the Mayo Score for predicting the yield of genetic testing in patients with hypertrophic cardiomyopathy. Arch Med Sci 2019; 15(3): 641–649. Dostupné z DOI: <http://dx.doi.org/10.5114/aoms.2018.78767>.
- Bos JM, Will ML, Gersh BJ et al. Characterization of a phenotype-based genetic test prediction score for unrelated patients with hypertrophic cardiomyopathy. Mayo Clin Proc 2014; 89(6): 727–737. Dostupné z DOI: <http://dx.doi.org/10.1016/j.mayocp.2014.01.025>.
- Murphy SL, Anderson JH, Kapplinger JD et al. Evaluation of the Mayo Clinic Phenotype-Based Genotype Predictor Score in Patients with Clinically Diagnosed Hypertrophic Cardiomyopathy. J Cardiovasc Transl Res 2016; 9(2): 153–161. Dostupné z DOI: <httP://dx.doi.org/10.1007/s12265–016–9681–5>.
- Alcalai R, Seidman JG, Seidman CE. Genetic basis of hypertrophic cardiomyopathy: from bench to the clinics. J Cardiovasc Electrophysiol 2008; 19(1): 104–110. Dostupné z DOI: <http://dx.doi.org/10.1111/j.1540–8167.2007.00965.x>.
- Mathew J, Zahavich L, Lafreniere-Roula M et al. Utility of genetics for risk stratification in pediatric hypertrophic cardiomyopathy. Clin Genet 2017; 93(2): 310–319. Dostupné z DOI: <http://dx.doi.org/10.1111/cge.13157>.
- Lopes LR, Brito D, Belo A et al. Genetic characterization and genotype-phenotype associations in a large cohort of patients with hypertrophic cardiomyopathy – An ancillary study of the Portuguese registry of hypertrophic cardiomyopathy. Int J Cardiol 2019; 278 : 173–179. Dostupné z DOI: <http://dx.doi.org/10.1016/j.ijcard.2018.12.012>.
- Blankenburg R, Hackert K, Wurster S et al. β-Myosin heavy chain variant Val606Met causes very mild hypertrophic cardiomyopathy in mice, but exacerbates HCM phenotypes in mice carrying other HCM mutations. Circ Res 2014; 115(2): 227–237. Dostupné z DOI: <http://dx.doi.org/10.1161/CIRCRESAHA.115.303178>.
- Dorn GW, McNally E. Two strikes and you’re out: gene-gene mutation interactions in HCM. Circ Res 2014; 115(2): 208–210. Dostupné z DOI: <http://dx.doi.org/10.1161/CIRCRESAHA.114.304383>.
- Ingles J, Doolan A, Chiu C et al. Compound and double mutations in patients with hypertrophic cardiomyopathy: implications for genetic testing and counselling. J Med Genet 2005; 42(10): e59. Dostupné z DOI: <http://dx.doi.org/10.1136/jmg.2005.033886>.
- Whiffin N, Minikel E, Walsh R et al. Using high-resolution variant frequencies to empower clinical genome interpretation. Genet Med 2017; 19(10): 1151–1158. Dostupné z DOI: <http://dx.doi.org/10.1038/gim.2017.26>.
- Rigopoulos AG, Ali M, Abate E et al. Advances in the diagnosis and treatment of transthyretin amyloidosis with cardiac involvement. Heart Fail Rev 2019; 24(4):521–533. Dostupné z DOI: <http://dx.doi.org/10.1007/s10741–019–09776–3>.
- Brosnan MJ, Rakhit D. Differentiating Athlete’s Heart From Cardiomyopathies – The Left Side. Hear Lung Circ 2018; 27(9): 1052–1062. Dostupné z DOI: <http://dx.doi.org/10.1016/j.hlc.2018.04.297>.
- Jiang J, Wakimoto H, Seidman JG et al. Allele-specific silencing of mutant Myh6 transcripts in mice suppresses hypertrophic cardiomyopathy. Science 2013; 342(6154): 111–114. Dostupné z DOI: <http://dx.doi.org/10.1126/science.1236921>.
- Cannon L, Yu ZY, Marciniec T et al. Irreversible Triggers for Hypertrophic Cardiomyopathy Are Established in the Early Postnatal Period. J Am Coll Cardiol 2015; 65(6): 560–569. Dostupné z DOI: <http://dx.doi.org/10.1016/j.jacc.2014.10.069>.
- Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell 2014; 157(6): 1262–1278. Dostupné z DOI: <http://dx.doi.org/10.1016/j.cell.2014.05.010>.
- Ma H, Marti-Gutierrez N, Park SW et al. Correction of a pathogenic gene mutation in human embryos. Nature 2017; 548(7668): 413–419. Dostupné z DOI: <http://dx.doi.org/10.1038/nature23305>.
Štítky
Diabetológia Endokrinológia Interné lekárstvo
Článok vyšiel v časopiseVnitřní lékařství
Najčítanejšie tento týždeň
2019 Číslo 10- Parazitičtí červi v terapii Crohnovy choroby a dalších zánětlivých autoimunitních onemocnění
- Intermitentní hladovění v prevenci a léčbě chorob
- Rizikové období v léčbě růstovým hormonem: přechod mladých pacientů k lékařům pro dospělé
- Statinová intolerance
- Monoklonální protilátky v léčbě hyperlipidemií
-
Všetky články tohto čísla
- Úvodník
- Interdisciplinární spolupráce k maximálnímu zrychlení dostupnosti moderní léčby ischemických iktů pro všechny nemocné, kteří potřebují endovaskulární trombektomii
- Novinky ve farmakologické léčbě srdečního selhání
- Nová evropská guidelines diagnostiky a léčby hypertenze 2018 – stručný komentář
- Akutní a rekurentní perikarditida
- Profylaxe infekční endokarditidy – nekonečný příběh s pestrou minulostí, neurčitou přítomností a nejasnou budoucností
- Zánětlivá onemocnění myokardu
- Současné a budoucí trendy v léčbě dyslipidemií
- Genetické vyšetření u pacientů s hypertrofickou kardiomyopatií
- Tako-tsubo syndrom
- Odešel profesor Marek, velký internista, endokrinolog, ale především velký člověk
- Z odborné literatury
- Vnitřní lékařství
- Archív čísel
- Aktuálne číslo
- Iba online
- Informácie o časopise
Najčítanejšie v tomto čísle- Akutní a rekurentní perikarditida
- Novinky ve farmakologické léčbě srdečního selhání
- Tako-tsubo syndrom
- Profylaxe infekční endokarditidy – nekonečný příběh s pestrou minulostí, neurčitou přítomností a nejasnou budoucností
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



