Dlouhé nekódující RNA a jejich význam u nádorových onemocnění
Long Non-Coding RNAs And Their Relevance in Cancer
A major portion of the eukaryotic genome is occupied by DNA sequences; transcripts of these sequences do not code for proteins. This part of the eukaryotic genome is transcribed in a developmentally regulated manner or as a response to external stimuli to produce large numbers of long non-coding RNAs (lncRNAs). Genome-wide studies indicate the existence of more than 3,300 lncRNAs. Long non-coding RNAs are tentatively defined as molecules of ncRNAs that are more than two hundred nucleotides long. Due to the complexity and diversity of their sequences, progress in the field of lncRNAs has been very slow. Nonetheless, lncRNAs have emerged as key molecules involved in the control of transcriptional and posttranscriptional gene regulatory pathways. Although limited numbers of functional lncRNAs have been identified so far, the immense regulatory potential of lncRNAs is already evident, emphasizing that a genome-wide characterization of functional lncRNAs is needed. The fact that many lncRNAs are deregulated in various human cancers, together with their functional characteristics, implies their eminent role in carcinogenesis. In this review, we summarize novel classes of lncRNAs, describe their biological functions emphasizing their roles in tumor biology and translational oncology research.
Key words:
long non-coding RNAs – lincRNA – pseudogenes – T-UCR – cancer
This study was supported by grants of Internal Grant Agency of the Czech Ministry of Health No. NT11214-4/2010, NT13514-4/2012, NT/13549, NT/13860 and NT/13585 and by Institutional Resources for Supporting the Research Organization provided by the Czech Ministry of Health in 2012.
The authors declare they have no potential conflicts of interest concerning drugs, products, or services used in the study.
The Editorial Board declares that the manuscript met the ICMJE “uniform requirements” for biomedical papers.
Submitted:
30. 6. 2012
Accepted:
3. 8. 2012
Authors:
J. Šána 1,2; P. Faltejsková 1,2; M. Svoboda 1,2,3
; O. Slabý 1,2
Authors‘ workplace:
CEITEC – Středoevropský technologický institut, MU Brno
1; Klinika komplexní onkologické péče, Masarykův onkologický ústav, Brno
2; Oddělení epidemiologie a genetiky nádorů, Masarykův onkologický ústav, Brno
3
Published in:
Klin Onkol 2012; 25(4): 246-254
Category:
Reviews
Overview
Většinu eukaryotického genomu představují DNA sekvence, které nekódují proteiny. Tyto sekvence jsou přepisovány buď podle vývojového programu daného organizmu nebo v rámci odpovědi na vnější signály. Výsledkem transkripce takových sekvencí je pak velké množství dlouhých nekódujících RNA (lncRNA). Celogenomové studie předpokládají existenci více než 3 300 lncRNA. Dlouhé nekódující RNA jsou definovány jako molekuly nekódujících RNA o délce více než 200 nukleotidů. Vzhledem k vysoké míře komplexnosti a rozmanitosti těchto sekvencí byl nárůst poznání v této oblasti relativně pomalý. Ačkoli bylo dosud funkčně charakterizováno pouze omezené množství lncRNA, jejich regulační potenciál je již dnes evidentní. LncRNA hrají klíčové role jak v transkripčních, tak v post-transkripčních regulačních drahách. U mnoha nádorových onemocnění dochází k deregulaci lncRNA, což společně s jejich funkčními vlastnostmi naznačuje jejich významný potenciál v procesech maligní transformace. Tento přehledový článek je zaměřen na shrnutí nedávno objevených skupin lncRNA, popis jejich biologických funkcí a zejména na jejich význam v nádorové biologii a translačním onkologickém výzkumu.
Klíčová slova:
dlouhé nekódující RNA – lincRNA –pseudogeny – T-UCR – nádorová onemocnění
Úvod
Centrální dogma molekulární biologie popisující vztah mezi genetickou informací a jejím fenotypovým vyjádřením pohlíží na RNA především jako na spojovací článek mezi DNA a proteinem. Tímto spojovacím článkem je primárně myšlena kódující mediátorová RNA (mRNA) určující pořadí aminokyselin v peptidovém řetězci. Avšak v samotném procesu translace jsou přímo zapojeny rovněž nekódující RNA (ribozomální a transferová RNA). Kromě toho je obecně akceptováno, že zásadní roli v biologii buňky hrají i další nekódující RNA, jako jsou například malé jaderné RNA účastnící se posttranskripčního sestřihu anebo malé jadérkové RNA zapojené do posttranskripční modifikace ribozomální RNA.
Na základě analýzy prvních zcela osekvenovaných prokaryotických genomů se předpokládalo, že většina genetické informace definující biologickou formu a fenotyp je exprimována ve formě proteinů, které mají nejen strukturální, ale také katalytické a regulační funkce [1]. Pokročilé čipové a sekvenační technologie umožňující vysokokapacitní celogenomové analýzy však odhalily, že tato skutečnost není zcela platná u eukaryotických organizmů. Jedním z největších překvapení moderní biologie byl objev, že lidský genom obsahuje pouze asi 20 000 protein-kódujících genů, což reprezentuje méně než 2 % jeho celkové kapacity (obr. 1). Neméně překvapivé bylo také zjištění, že nejméně 90 % genomu je transkripčně aktivních. Lidský transkriptom tedy zdaleka neobsahuje pouze sekvence protein-kódujících genů a jejich sestřihových variant, ale je v něm nepoměrně větší množství nekódujících RNA [1,2]. Obecně lze říci, že čím více je organizmus komplexní, tím větší počet nekódujících RNA jeho genom obsahuje. Nejprve byly tyto nekódující transkripty považovány za evolučně nahromaděný genetický odpad vzniklý při časném sestavování genů nebo inzercí mobilních genetických elementů. Nedávné výzkumy ovšem prokázaly, že tyto nekódující RNA hrají zásadní role v buněčném vývoji, fyziologii, ale také patologiích. Tuto domněnku podporuje mimo jiné fakt, že většina chromatin modifikujících komplexů postrádá schopnost vázat DNA. Na regulaci genové exprese a tedy kontrolách buněčných funkcí se tak musejí podílet další molekuly, mezi které nepochybně patří právě nekódující RNA. Příkladem takové nekódující RNA je Xist, která je nezbytná pro navázaní chromatin modelujících komplexů na chromozom X zajišťujících jeho inaktivaci v samičích somatických buňkách [4]. Obecně mohou nekódující RNA regulovat genovou expresi přímo, a nebo mohou zprostředkovávat zmíněnou vazbu mezi proteinovými komplexy a DNA, což je činí nenahraditelnou součástí sytému buněčných regulací [2–4].
![Zastoupení protein-kódujících sekvencí u vybraných eukaryotických a bakteriálních genomů. Upraveno podle [17].](https://www.prelekara.sk/media/cache/resolve/media_object_image_small/media/image/d420392fbe68664336ca4de7b4840cdd.jpg)
Dnešní vnímání regulačních funkcí dlouhých nekódujících RNA bylo významně ovlivněno pracemi Johna S. Mat-ticka z univerzity v Queenslandu, který patří mezi průkopníky myšlenky, že proteiny představují pouze minoritní část informačních výstupů eukaryotického genomu [3].
Nekódující RNA se dělí na základě své velikosti na dvě hlavní skupiny – krátké nekódující RNA a dlouhé nekódující RNA (lncRNA). Zatímco krátké nekódující RNA dosahují délky maximálně několika desítek bazí, lncRNA jsou transkripty podobné mRNA o délce od 200 nt, nicméně velmi často přesahující 100 kb. Avšak narozdíl od kódující RNA obsahují lncRNA pouze krátké otevřené čtecí rámce, nebo je zcela postrádají, což znemožňuje jejich účinný překlad do proteinu. Mnoho identifikovaných lncRNA je přepisováno RNA polymerázou II (RNAPII), následně sestřihováno, některé z nich mají polyadenylovány 3’ konce a obsahují 5’ čepičku [2]. Jiné lncRNA jsou však díky struktuře svých promotorových sekvencí přepisovány spíše RNA polymerázou III (RNAPIII) [5]. Z dosavadního výzkumu rovněž vyplývá, že lncRNA jsou velmi často komplementární ke kódujícím RNA řetězcům a jejich promotory jsou regulovány jak transkripčními faktory, tak epigeneticky pomocí specifických modifikací histonů [6]. Exprese lncRNA může být vývojově a tkáňově specifická a samotné molekuly jsou spojovány se širokým spektrem buněčných procesů, jako jsou alternativní sestřih, modulace proteinové aktivity, změny proteinové lokalizace a epigenetické regulace. LncRNA mohou být prekurzory krátkých nekódujících RNA nebo naopak součástí mechanizmu jejich umlčení [7–13]. Nicméně jejich hlavní úloha se zdá být přímo v regulaci genové exprese, a to jak na transkripriční, tak post-transkripční úrovni (obr. 2) [14]. Není proto překvapující, že deregulovaná exprese lncRNA byla pozorována u mnoha multifaktoriálně podmíněných chorob, včetně nádorových onemocnění, ischemické choroby srdeční [15] a Alzheimerovy choroby [16], a je se vznikem a progresí těchto onemocnění funkčně asociována [2]. LncRNA proto představují novou třídu potenciálních biomarkerů a terapeutických cílů u těchto onemocnění [17–20].
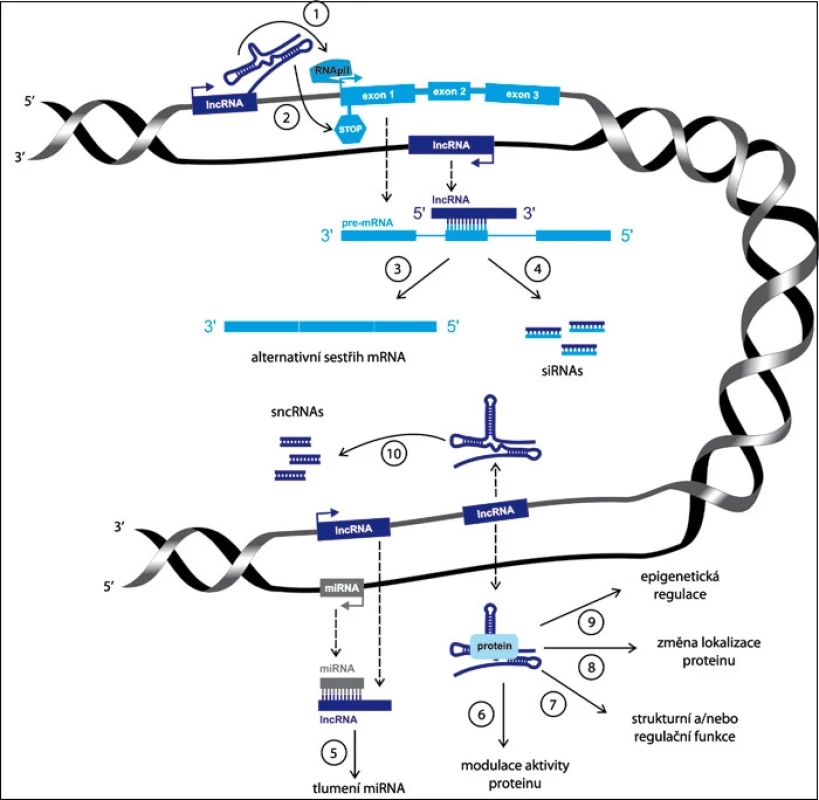
Tento článek je zaměřen na celkový přehled nedávno objevených skupin lncRNA, jejich biologické funkce a zejména na jejich význam v nádorové biologii a translačním onkologickém výzkumu (tab. 1).
![Hlavní třídy dlouhých nekódujících RNA. Upraveno podle [17].](https://www.prelekara.sk/media/cache/resolve/media_object_image_small/media/image/399ee5a79499e962b727c5987a78ea4b.png)
Dlouhé mezigenové nekódující RNA
Dlouhé mezigenové nekódující RNA (long non-coding RNA – lincRNA) jsou nově objevenou třídou lncRNA. RNA této třídy jsou dlouhé od několika stovek až po několik desítek tisíc bazí a v genomu se nacházejí vždy mezi dvěma geny. Doposud bylo identifikováno více než 3 000 lidských lincRNA, avšak méně než 1 % z nich bylo detailněji charakterizováno [6,21]. Nicméně bylo prokázáno, že lincRNA jsou zapojeny v biologických procesech, jako jsou např. imprinting nebo maligní transformace [11,20,21]. Z nedávných studií rovněž vyplývá, že tyto molekuly mění svůj expresní profil v průběhu fyziologického vývoje jedince, v odpovědi na různé druhy signalizací a vykazují významně odlišné expresní hladiny v primárních nádorech a metastázách ve srovnání s příslušnou nenádorovou tkání [6].
Jednou z prvních detailněji chrakterizovaných lincRNA je HULC (Highly Upregulated in Liver Cancer). Tato lncRNA je kódována na lokusu 6p24.3 a nese znaky typické pro mRNA, včetně jednoho intronu ohraničeného sestřihovými místy GT-AG, polyadenylovaného konce a jaderného exportního signálu, díky čemuž je HULC silně lokalizována v cytoplazmě. Ačkoli byla HULC purifikována společně s ribozomy, dosud nebyl detekován žádný její translační produkt [22]. HULC je významně zvýšena u jaterních metastáz kolorektálního karcinomu a v buněčných liniích hepatocelulárního karcinomu [23]. Na základě současných pozorování se zdá, že HULC může vyvazovat jak různé transkripční faktory a signální proteiny, tak některé mikroRNA, čímž zabraňuje jejich vazbě na cílovou molekulu, a reguluje tak její aktivitu a/nebo expresi. Mezi takto regulované mikroRNA patří také známá onkogenní miR-372, jež post-transkripčně reguluje PRKACB (protein kinase, cAMP-dependent, catalytic, beta), která indukuje fosforylaci proteinu CREB (cAMP responsive element binding protein). Aktivovaný protein CREB je schopný udržovat otevřenou chromatinovou strukturu v oblasti promotoru pro HULC, což následně vede ke zvýšené transkripci HULC, a vytváří tak pozitivní zpětnovazebnou smyčku [24].
Další dobře popsaná RNA patřící do podskupiny lincRNA je HOTAIR (HOX antisense intergenic RNA). HOTAIR je 2.2 kb dlouhý gen lokalizovaný u člověka uvnitř genového klastru HOXC na dlouhém raménku chromozomu 2. Bylo zjištěno, že tato lincRNA může prostřednictvím navázaného PRC2 (polycomb repressive complex 2) a následné trimetylace lyzinu 27 histonu H3 (H3K27) regulovat geny HOXD [20]. Obecně 5‘ oblast na HOTAIR váže protein PRC2 (polycomb repressive complex 2), který je zodpovědný za metylaci H3K27, zatímco její 3‘ oblast váže LSDS1, histon-lyzinovou demetylázu zprostředkovávající enzymatickou demetylaci H3K4Me2. HOTAIR se vyskytuje u savců, má málo konzervované sekvence, avšak vysoce konzervovanou strukturu a vyvinula se pravděpodobně dříve než okolní HOXC geny [25]. HOTAIR byla jednou z prvních lncRNA, u nichž byla popsána asociace s metastazováním. Hladina její exprese byla v porovnání s odpovídající nenádorovou tkání významně zvýšená u primárních i metastatických nádorů prsu a vysoké hladiny exprese HOTAIR navíc korelují se špatnou prognózou [11]. V případě hepatocelulárního karcinomu je zvýšená exprese HOTAIR kandidátním biomarkerem pro predikci časné rekurence u pacientů, kteří podstoupili transplantaci jater [26].
Cílená regulace nádorově specifických krátkých nekódujících RNA, především mikroRNA, se ukazuje být správným směrem v rámci moderních terapeutických strategií. Z toho důvodu je velmi žádoucí, aby byly navrhnuty molekuly, jež budou schopny inhibovat také lincRNA. Gupta et al ve své práci uklázali, že tyto molekuly mohou být degradovány pomocí siRNA, avšak tento způsob je z důvodu četných sekundárních struktur lincRNA komplikovaný a neefektivní [11,25]. Nicméně je evidentní, že nádorově asociované lincRNA představují slibnou skupinu lncRNA s potenciálem pro onkologickou diagnostiku a moderní terapie.
Dlouhé intronové nekódující RNA
Ačkoli není doposud biogeneze intronových lncRNA dostatečně popsána, výsledky některých studií nepřímo ukazují, že by v tomto procesu mohla být zapojena RNAPII. Svědčí o tom zejména shodně koregulované expresní profily mnoha intronových lncRNA s příslušnými protein-kódujícími transkripty, významné zapojení transkripčních faktorů asociovaných s RNAPII a v neposlední řadě přítomnost polyA konce [27–31]. Avšak po ovlivnění alfa-amanitinem, specifickým RNAPII inhibitorem, došlo ke zvýšené transkripci u 10 % dlouhých intronových poly(A+) nekódujících RNA oproti pouze 4 % protein-kódujícím genům [27,30,32]. Tato skutečnost tedy naznačuje, že některé tyto intronové lncRNA společně s některými protein-kódujícími RNA jsou z DNA přepisovány pomocí RNAP III nebo spRNAP IV (single-polypeptide nuclear RNA polymerase IV), jejíž aktivita se zdá být zesilována účinky právě zmíněného alfa-amanitinu [27,32–36].
Studie zkoumající evoluční konzervovanost intronových IncRNA napříč různými obratlovci odhalily, že délka konzervovaných sekvencí se pohybuje pouze okolo 100 bp. Ovšem při porovnání více příbuzných druhů, jako je člověk, myš, kráva a pes, vzrostla tato délka až na 750 bp [37,38]. Mezidruhový výskyt, tkáňová a subbuněčná expresní specificita, evoluční konzervovanost, schopnost reagovat na změny vnějšího prostředí a aberantní exprese u lidských nádorových onemocnění svědčí o zapojení intronových lncRNA do regulace genové exprese. Složitost těchto regulačních vztahů pak dokazuje zejména různá exprese intronových lncRNA a k nim korespondujících protein-kódujících sekvencí. Zatímco v některých případech je možné pozorovat vzájemně shodné expresní hladiny, u jiných byla naopak prokázána inverzní korelace [27,30,39,40]. Některé krátké nekódující RNA, jako jsou mikroRNA, bývají kódovány uvnitř intronových oblastí, především pak 5‘-orientovaných, přičemž většina intronových lncRNA je také součástí prvních intronů hostitelských genů. Z tohoto důvodu se předpokládá, že mnoho intronových lncRNA je zpracováváno do krátkých nekódujících RNA [27,41,42]. Podobně jako u mezigenových lncRNA byla popsána HOTAIR, mezi intronovými byla identifikována COLDAIR (cold assisted intronic noncoding RNA) zprostředkovávající při dlouhodobém působení nízkých teplot epigenetické umlčení genu FLC (FLOWERING LOCUS C) prostřednictvím již zmíněného proteinu PRC2 [43].
Velmi zajímavá studie ukázala, že intronové lncRNA jsou různě exprimovány v primárních a metastatických nádorech pankreatu. Navíc intronové lncRNA, které jsou odlišně exprimovány v metastázách duktálního adenokarcinomu pankreatu, se ve většině případů vyskytují uvnitř genů asociovaných s MAPK signální dráhou [44]. Tato pozorování tedy poukazují na potenciální význam této třídy nekódujících transkriptů v biologických procesech spojených s maligní transformací a metastazováním.
Nekódující RNA asociované s telomerami
Telomery chrání konce lineárních chromozomů před reparačním mechanizmem, který by je jinak rozpoznal a zpracoval jako dvouřetězcové zlomy na DNA. Tato ochranná funkce telomer je tedy nezbytná pro stabilitu chromozomů a až do nedávné doby panovala domněnka, že telomery jsou transkripčně neaktivní [45]. Nedávno ovšem několik výzkumných skupin nezávisle na sobě demonstrovalo, že silně metylované subtelomerické a telomerické oblasti, ačkoli postrádají protein-kódující geny, vykazují transkripční aktivitu a přepisují se do telomerových UUAGGG-repetic, čímž vzniká nekódující RNA (TERRA) [46–48]. Molekuly TERRA jsou sekvenčně konzervovány napříč eukaryoty, a byly tedy identifikovány i u člověka. Tyto transkripty jsou často syntetizovány z oblastí bohatých na konstitutivní heterochromatin, jsou polyadenylovány a jejich syntéza je senzitivní k působení alfa-amanitinu, což naznačuje, že jsou přepisovány RNAPII [45,49].
Délka TERRA se pohybuje v rozmezí od 100 bp až po více než 9 kb, jsou exprimovány v různých fázích buněčného cyklu a bylo zjištěno, že jejich expresi ovlivňuje několik faktorů, mezi než patří délka telomer, buněčný stres, stupeň buněčné diferenciace, struktura telomerického heterochromatinu, ale také například stadium nádorového onemocnění [45,48]. Bylo zjištěno, že poškození některých klíčových buněčných procesů, jako je např. dráha zajišťující odbourávání aberantních transkriptů u člověka nebo ztráta 5‘-3‘ exonukleázy Rat1p u Saccharomyces cerevisiae, je asociováno s významně zvýšenými hladinami TERRA. Vysoké hladiny TERRA pak korelují se ztrátou kompenzační kapacity telomer [46,48,49]. Je tedy velmi pravděpodobné, že TERRA jsou důležitými regulátory telomerázové aktivity [45,47,49]. Sampl et al zjistili, že expresní hladiny TERRA negativně korelují s malignitou gliomů [50]. Tyto výsledky navíc korespondují s pozorováním u nádorů hrtanu, tlustého střeva a lymfatických uzlin [47]. Molekuly TERRA se proto zdají být zajímavým směrem nejen ve výzkumu telomer, ale také nádorových onemocnění, u kterých je porucha na úrovni telomerických sekvencí velice častá [51].
Dlouhé nekódující RNA s dvojitou funkcí
Ještě do nedávné doby byly nekódující RNA striktně považovány za molekuly s regulačními funkcemi a nebyla jim přisuzována žádná protein-kódující kapacita typická pro mRNA. Ovšem nedávné studie identifikovaly a charakterizovaly bifunkční RNA transkripty vykazující jak funkci regulační, tak schopnost kódovat proteiny. Tato pozorování naznačují, že jednoznačná kategorizace některých molekul RNA nebude možná [52]. Mezi molekuly tohoto typu patří např. SRA (steroid receptor RNA activator), který vykazuje vlastnosti a funkce nekódující RNA, avšak začlenění specifické oblasti na jeho 5‘ konec vede k translaci a vzniku proteinu SRA (SRAP) [53–55]. SRA je známý RNA-koaktivátor mnoha jaderných receptorů, a je tedy schopný regulovat genovou expresi. Stejnou funkci plní i ve formě proteinu [52,53,56]. SRA byl identifikován v mnoha lidských tkáních. Jeho zvýšená exprese pak byla pozorována v játrech, kosterní svalovině, nadledvinkách a hypofýze. Nižší expresi naopak vykazovaly placenta, plíce, ledviny a slinivka [53]. Vysoká exprese SRA byla rovněž popsána u nádorů vaječníků a prsu [57–59]. Navíc serózní karcinomy vaječníku vykazují vyšší hladinu SRA než jejich granulózní formy. SRA tak nejenže představuje potenciální diagnostický biomarker, ale především je zcela novým fenoménem v oblasti biologie RNA, kdy jediný RNA transkript může vést jak ke vzniku funkčního proteinového produktu, tak setrvat na úrovni RNA a realizovat regulační funkce jako nekódující RNA [52,57].
Pseudogeny
Pseudogeny jsou kopiemi genů, které ztratily schopnost kódovat protein. Typicky to bývají poškozené nebo nekompletní protein-kódující sekvence, dlouho označované jako „junk“ DNA, tedy jako nepotřebná DNA vznikající během evoluce genomu [60,61]. Pseudogeny vznikají retrotranspozicí mRNA, většinou potom jako vedlejší produkty LINE-1 (long interspersed nuclear elements) retrotranspozice. Jsou tedy zpětně přepisovány a reintegrovány do genomické DNA. Proto původní „rodičovská“ sekvence nemusí být nutně na stejném chromozomu jako její retrotranspozonová kopie [62,63]. Takto reintegrovaná DNA může mít v zásadě tři funkce; jednak může být zcela neporušena, a zachovat si tak schopnost kódovat proteiny, a nebo může dojít k jejímu porušení a vzniku pseudogenu, který se v závislosti na míře poškození a umístění může i nemusí přepisovat do RNA [60]. Nedávná velmi zajímavá pozorování ukázala, že transkribované pseudogeny pravděpodobně mohou regulovat expresi jejich vývojově příbuzných protein-kódujících sekvencí [60,61]. Zajímavé rovněž je, že některé z těchto molekul vykazují tkáňově specifickou expresi.
Bylo zjištěno, že pseudogeny přepsané do lncRNA mohou být dále procesovány do krátkých RNA, které jsou schopny posttranskripčně regulovat genovou expresi prostřednictvím RNA interference. Kromě toho bylo zjištěno, že miR-220 a miR-492, mikroRNA specifické pro primáty, leží obě uvnitř pseudogenů [64]. Jiný a zcela odlišný mechanizmus regulace genové exprese pak spočívá v působení přepsaných pseudogenů jako „vychytávačů“ mikroRNA, které po navázání na tyto lncRNA nemohou dále regulovat své cílové sekvence [61,63].
Několik studií popisuje deregulaci exprese pseudogenů v průběhu nádorové progrese, což naznačuje možné funkční zapojení těchto molekul do procesu kancerogeneze a představuje je jako nové potenciální diagnostické nádorové biomarkery a terapeutické cíle. Jedním z takových pseudogenů je MYLKP1 (myosin light chain kinase pseudogen), který je částečně duplikován z původního genu MYLK (myosin light chain kinase). Funkční gen MYLK kóduje dvě izoformy, nmMLCK (nonmuscle myosin light chain kinase) a smMLCK (smooth muscle myosin light chain kinase) a reguluje buněčné kontrakce a cytokinezi. Navzdory významné homologii obou promotorů (~ 90 %) vykazuje promotor pro MYLKP1 ve zdravých bronchiálních epiteliálních buňkách oproti promotoru pro smMLCK minimální aktivitu. V buňkách adenokarcinomu plic však byl pozorován opačný jev, kdy promotor pro MYLKP1 vykazoval mnohonásobně vyšší aktivitu. Stejný trend byl pozorován rovněž ve tkáni kolorektálního karcinomu v porovnání s nenádorovou tkání tlustého střeva. Z funkčního hlediska se tedy zdá, že MYLKP1 inhibuje expresi smMLCK, což vede ke zvýšené buněčné proliferaci [65]. Kromě MYLKP1 byl u karcinomu prostaty popsán KLK31P, další pseudogen hrající pravděpodobně roli v kancerogenezi, avšak jeho detailnější charakterizace ještě nebyla provedena [66].
Tyto nálezy nejenže staví pseudogeny do role slibných diagnostických biomarkerů, ale především pseudogenům, které byly mnoho let považovány za pouhé evoluční relikty, přiřazují biologickou funkci.
Přepisované vysoce konzervované oblasti
V současné době je popsáno 481 sekvencí delších než 200 bp, které jsou vysoce konzervované mezi ortologními oblastmi lidského, krysího a myšího genomu. Tyto vysoce konzervované oblasti (ultraconserved regions – UCR) jsou lokalizovány jak v intra–, tak i v intergenových regionech. U člověka jsou pak některé z těchto UCR přepisovány do RNA (transcribed-ultraconserved regions – T-UCR) [67].
Z funkčního hlediska působí UCR jako zesilovače genové transkripce [68], zatímco T-UCR se na základě dosavadních poznatků jeví spíše jako inhibitory protein-kódujících genů, ale i některých nekódujících RNA, včetně mikroRNA. Vztah mezi T-UCR a mikroRNA ovšem není jednostranný a některé mikroRNA mohou pravděpodobně zpětně regulovat T-UCR. Tento předpoklad vyplývá především ze zjištění, že mnoho T-UCR obsahuje sekvence komplementární s některými mikroRNA. Mezi těmito navzájem komplementárními molekulami byla opakovaně pozorována negativní korelace jejich exprese [69,70].
Nedávné studie naznačily, že exprese T-UCR je u vybraných nádorových onemocnění, například chronické lymfatické leukémie, kolorektálního a hepatocelulárního karcinomu a neuroblastomu deregulována a koreluje s klinickopatologickými charakteristikami pacientů [70–72]. Navíc v případě neuroblastomu bylo zjištěno zapojení těchto molekul do procesů buněčné apoptózy a diferenciace [73]. U mnoha nádorových onemocnění je velmi častým jevem hypermetylace CpG ostrůvků v T-UCR genech, čímž dochází k jejich epigenetické inaktivaci. Tato skutečnost tedy podporuje obecný předpoklad, že jak genetické, tak i epigenetické změny u protein-kódujících, ale i nekódujících DNA sekvencí přispívají k maligní transformaci [69]. Význam UCR v kancerogenezi naznačil i vztah jednonukleotidových polymorfizmů (SNP) rs9572903 a rs2056116 nacházejících se v UCR oblastech se zvýšeným rizikem familiárního karcinomu prsu [74,75].
Závěr
Majoritní část transkriptomu tvoří nekódující RNA, které jsou významnými regulátory genové exprese. Významnou podskupinou těchto RNA jsou lncRNA o délce od 200 nt až po více než 100 kb. LncRNA jsou zapojeny do mnoha buněčných procesů, včetně alternativního sestřihu, modulace proteinové aktivity, změny proteinové lokalizace a především transkripční i post-transkripční regulace genové exprese. Díky těmto vlastnostem jsou lncRNA významnými molekulami v rámci buněčné biologie, nepostradatelné při udržování fyziologického stavu buňky. Alterace jejich hladin mohou mít zásadní důsledky a významně přispívat k procesu maligní transformace u mnoha nádorových onemocnění (tab. 2). LncRNA tak představují slibné diagnostické, prognostické a prediktivní biomarkery a také potenciální terapeutické cíle. Přestože v posledních letech bylo dosaženo významného pokroku v oblasti studia lncRNA, funkce a přesné mechanizmy, kterými se lncRNA zapojují do kancerogeneze jednotlivých nádorových onemocnění, nejsou stále uspokojivě objasněny.
![LncRNA deregulované u nádorových onemocnění. Upraveno podle [112].](https://www.prelekara.sk/media/cache/resolve/media_object_image_small/media/image/0f543fdb5c45ac319cdb0eff63cb1803.png)
Práce byla podpořena granty IGA MZČR NT11214-4/2010, NT13514-4/2012, NT/13549, NT/13860, NT/13585, a prostředky institucionální podpory výzkumné organizace poskytnuté Ministerstvem zdravotnictví ČR v roce 2012.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
RNDr. Ondřej Slabý, Ph.D.
Klinika komplexní onkologické péče
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: on.slaby@gmail.com
Obdrženo: 30. 6. 2012
Přijato: 3. 8. 2012
Sources
1. Stein LD. Human genome: end of the beginning. Nature 2004; 431(7011): 915–916.
2. Taft RJ, Pang KC, Mercer TR et al. Non-coding RNAs: regulators of disease. J Pathol 2010; 220(2): 126–139.
3. Mattick JS. Non-coding RNAs: the architects of eukaryotic complexity. EMBO Rep 2001; 2 : 986–991.
4. Zhang R, Zhang L, Yu W. Genome-wide expression of non-coding RNA and global chromatin modification. Acta Biochim Biophys Sin (Shanghai) 2012; 44(1): 40–47.
5. Pagano A, Castelnuovo M, Tortelli F et al. New small nuclear RNA gene-like transcriptional units as sources of regulatory transcripts. PLoS Genet 2007; 3(2): e1.
6. Guttman M, Amit I, Garber M et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature 2009; 458(7235): 223–237.
7. Costa FF. Non-coding RNAs: Meet thy masters. Bioessays 2010; 32(7): 599–608.
8. Chen Y, Song Y, Wang Z et al. Altered expression of MiR-148a and MiR-152 in gastrointestinal cancers and its clinical significance. J Gastrointest Surg 2010; 14(7): 1170–1179.
9. Lipovich L, Johnson R, Lin CY. MacroRNA underdogs in a mikroRNA world: evolutionary, regulatory, and biomedical significance of mammalian long non-protein-coding RNA. Biochim Biophys Acta 2010; 1799(9): 597–615.
10. Tripathi V, Ellis JD, Shen Z et al. The nuclear-retained noncoding RNA MALAT1 regulates alternative splicing by modulating SR splicing factor phosphorylation. Mol Cell 2010; 39(6): 925–938.
11. Gupta RA, Shah N, Wang KC et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature 2010; 464(7291): 1071–1076.
12. Orom UA, Derrien T, Beringer M et al. Long noncoding RNAs with enhancer-like function in human cells. Cell 2010; 143(1): 46–58.
13. Iacoangeli A, Lin Y, Morley EJ et al. BC200 RNA in invasive and preinvasive breast cancer. Carcinogenesis 2004; 25(11): 2125–2133.
14. Mattick JS, Amaral PP, Dinger ME et al. RNA regulation of epigenetic processes. Bioessays 2009; 31(1): 51–59.
15. Ishii N, Ozaki K, Sato H et al. Identification of a novel non-coding RNA, MIAT, that confers risk of myocardial infarction. J Hum Genet 2006; 51(12): 1087–1099.
16. Faghihi MA, Modarresi F, Khalil AM et al. Expression of a noncoding RNA is elevated in Alzheimer’s disease and drives rapid feed-forward regulation of beta-secretase. Nat Med 2008; 14(7): 723–730.
17. Sana J, Faltejskova P, Svoboda M et al. Novel classes of non-coding RNAs and cancer. J Transl Med 2012; 10(1): 103.
18. Costa FF. Non-coding RNAs: new players in eukaryotic biology. Gene 2005; 357(2): 83–94.
19. Okamura K, Chung WJ, Ruby JG et al. The Drosophila hairpin RNA pathway generates endogenous short interfering RNAs. Nature 2008; 453(7196): 803–806.
20. Rinn JL, Kertesz M, Wang JK et al. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell 2007; 129(7): 1311–1323.
21. Khalil AM, Guttman M, Huarte M et al. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc Natl Acad Sci U S A 2009; 106(28): 11667–11672.
22. Brannan CI, Dees EC, Ingram RS et al. The product of the H19 gene may function as an RNA. Mol Cell Biol 1990; 10(1): 28–36.
23. Gabory A, Jammes H, Dandolo L. The H19 locus: role of an imprinted non-coding RNA in growth and development. Bioessays 2010; 32(6): 473–480.
24. Hibi K, Nakamura H, Hirai A et al. Loss of H19 imprinting in esophageal cancer. Cancer Res 1996; 56(3): 480–482.
25. Tsai MC, Manor O, Wan Y et al. Long noncoding RNA as modular scaffold of histone modification complexes. Science 2010; 329(5992): 689–693.
26. Yang Z, Zhou L, Wu LM et al. Overexpression of long non-coding RNA HOTAIR predicts tumor recurrence in hepatocellular carcinoma patients following liver transplantation. Ann Surg Oncol 2011; 18(5): 1243–1250.
27. Louro R, Smirnova AS, Verjovski-Almeida S. Long intronic noncoding RNA transcription: expression noise or expression choice? Genomics 2009; 93(4): 291–298.
28. Louro R, Nakaya HI, Amaral PP et al. Androgen responsive intronic non-coding RNAs. BMC Biol 2007; 5 : 4.
29. Cawley S, Bekiranov S, Ng HH et al. Unbiased mapping of transcription factor binding sites along human chromosomes 21 and 22 points to widespread regulation of noncoding RNAs. Cell 2004; 116(4): 499–509.
30. Nakaya HI, Amaral PP, Louro R et al. Genome mapping and expression analyses of human intronic noncoding RNAs reveal tissue-specific patterns and enrichment in genes related to regulation of transcription. Genome Biol 2007; 8(3): R43.
31. Dinger ME, Amaral PP, Mercer TR et al. Long noncoding RNAs in mouse embryonic stem cell pluripotency and differentiation. Genome Res 2008; 18(9): 1433–1445.
32. Kravchenko JE, Rogozin IB, Koonin EV et al. Transcription of mammalian messenger RNAs by a nuclear RNA polymerase of mitochondrial origin. Nature 2005; 436(7051): 735–739.
33. Li SC, Tang P, Lin WC. Intronic mikroRNA: discovery and biological implications. DNA Cell Biol 2007; 26(4): 195–207.
34. Borchert GM, Lanier W, Davidson BL. RNA polymerase III transcribes human mikroRNAs. Nat Struct Mol Biol 2006; 13(12): 1097–1101.
35. Massone S, Vassallo I, Castelnuovo M et al. RNA polymerase III drives alternative splicing of the potassium channel-interacting protein contributing to brain complexity and neurodegeneration. J Cell Biol 2011; 193(5): 851–866.
36. Massone S, Vassallo I, Fiorino G et al. 17A, a novel non-coding RNA, regulates GABA B alternative splicing and signaling in response to inflammatory stimuli and in Alzheimer disease. Neurobiol Dis 2011; 41(2): 308–317.
37. Louro R, El-Jundi T, Nakaya HI et al. Conserved tissue expression signatures of intronic noncoding RNAs transcribed from human and mouse loci. Genomics 2008; 92(1): 18–25.
38. Rearick D, Prakash A, McSweeny A et al. Critical association of ncRNA with introns. Nucleic Acids Res 2011; 39(6): 2357–2366.
39. Mercer TR, Dinger ME, Sunkin SM et al. Specific expression of long noncoding RNAs in the mouse brain. Proc Natl Acad Sci U S A 2008; 105(2): 716–721.
40. Katayama S, Tomaru Y, Kasukawa T et al. Antisense transcription in the mammalian transcriptome. Science 2005; 309(5740): 1564–1566.
41. Filipowicz W, Pogacic V. Biogenesis of small nucleolar ribonucleoproteins. Curr Opin Cell Biol 2002; 14(3): 319–327.
42. Hirose T, Ideue T, Nagai M et al. A spliceosomal intron binding protein, IBP160, links position-dependent assembly of intron-encoded box C/D snoRNP to pre-mRNA splicing. Mol Cell 2006; 23(5): 673–684.
43. Heo JB, Sung S. Vernalization-mediated epigenetic silencing by a long intronic noncoding RNA. Science 2011; 331(6013): 76–79.
44. Tahira AC, Kubrusly MS, Faria MF et al. Long noncoding intronic RNAs are differentially expressed in primary and metastatic pancreatic cancer. Mol Cancer 2011; 10 : 141.
45. Isken O, Maquat LE. Telomeric RNAs as a novel player in telomeric integrity. F1000 Biol Rep 2009; 1 : 90.
46. Azzalin CM, Reichenbach P, Khoriauli L et al. Telomeric repeat containing RNA and RNA surveillance factors at mammalian chromosome ends. Science 2007; 318(5851): 798–801.
47. Schoeftner S, Blasco MA. Developmentally regulated transcription of mammalian telomeres by DNA-dependent RNA polymerase II. Nat Cell Biol 2008; 10(2): 228–236.
48. Schoeftner S, Blasco MA. A ‚higher order‘ of telomere regulation: telomere heterochromatin and telomeric RNAs. EMBO J 2009; 28(16): 2323–2336.
49. Luke B, Panza A, Redon S et al. The Rat1p 5‘ to 3‘ exonuclease degrades telomeric repeat-containing RNA and promotes telomere elongation in Saccharomyces cerevisiae. Mol Cell 2008; 32(4): 465–477.
50. Sampl S, Pramhas S, Stern C et al. Expression of Telomeres in Astrocytoma WHO Grade 2 to 4: TERRA Level Correlates with Telomere Length, Telomerase Activity, and Advanced Clinical Grade. Transl Oncol 2012; 5(1): 56–65.
51. Schoeftner S, Blasco MA. Chromatin regulation and non-coding RNAs at mammalian telomeres. Semin Cell Dev Biol 2010; 21(2): 186–193.
52. Ulveling D, Francastel C, Hube F. When one is better than two: RNA with dual functions. Biochimie 2011; 93(4): 633–644.
53. Lanz RB, McKenna NJ, Onate SA et al. A steroid receptor coactivator, SRA, functions as an RNA and is present in an SRC-1 complex. Cell 1999; 97(1): 17–27.
54. Kawashima H, Takano H, Sugita S et al. A novel steroid receptor co-activator protein (SRAP) as an alternative form of steroid receptor RNA-activator gene: expression in prostate cancer cells and enhancement of androgen receptor activity. Biochem J 2003; 369(Pt 1): 163–171.
55. Chooniedass-Kothari S, Emberley E, Hamedani MK, et al. The steroid receptor RNA activator is the first functional RNA encoding a protein. FEBS Lett 2004; 566 (1–3): 43–47.
56. Xu K, Liang X, Cui D et al. miR-1915 inhibits Bcl-2 to modulate multidrug resistance by increasing drug-sensitivity in human colorectal carcinoma cells. Mol Carcinog 2011. [Epub ahead of print].
57. Hussein-Fikret S, Fuller PJ. Expression of nuclear receptor coregulators in ovarian stromal and epithelial tumours. Mol Cell Endocrinol 2005; 229(1–2): 149–160.
58. Lanz RB, Chua SS, Barron N et al. Steroid receptor RNA activator stimulates proliferation as well as apoptosis in vivo. Mol Cell Biol 2003; 23(20): 7163–7176.
59. Leygue E, Dotzlaw H, Watson PH et al. Expression of the steroid receptor RNA activator in human breast tumors. Cancer Res 1999; 59(17): 4190–4193.
60. Harrison PM, Zheng D, Zhang Z et al. Transcribed processed pseudogenes in the human genome: an intermediate form of expressed retrosequence lacking protein-coding ability. Nucleic Acids Res 2005; 33(8): 2374–2383.
61. Pink RC, Wicks K, Caley DP et al. Pseudogenes: pseudo-functional or key regulators in health and disease? RNA 2011; 17(5): 792–798.
62. Esnault C, Maestre J, Heidmann T. Human LINE retrotransposons generate processed pseudogenes. Nat Genet 2000; 24(4): 363–367.
63. Terai G, Yoshizawa A, Okida H et al. Discovery of short pseudogenes derived from messenger RNAs. Nucleic Acids Res 2010; 38(4): 1163–1171.
64. Devor EJ. Primate mikroRNAs miR-220 and miR-492 lie within processed pseudogenes. J Hered 2006; 97(2): 186–190.
65. Han YJ, Ma SF, Yourek G et al. A transcribed pseudogene of MYLK promotes cell proliferation. FASEB J 2011; 25(7): 2305–2312.
66. Lu W, Zhou D, Glusman G et al. KLK31P is a novel androgen regulated and transcribed pseudogene of kallikreins that is expressed at lower levels in prostate cancer cells than in normal prostate cells. Prostate 2006; 66(9): 936–944.
67. Bejerano G, Pheasant M, Makunin I et al. Ultraconserved elements in the human genome. Science 2004; 304(5675): 1321–1325.
68. Nobrega MA, Ovcharenko I, Afzal V et al. Scanning human gene deserts for long-range enhancers. Science 2003; 302(5 644): 413.
69. Lujambio A, Portela A, Liz J et al. CpG island hypermethylation-associated silencing of non-coding RNAs transcribed from ultraconserved regions in human cancer. Oncogene 2010; 29(48): 6390–63401.
70. Calin GA, Liu CG, Ferracin M et al. Ultraconserved regions encoding ncRNAs are altered in human leukemias and carcinomas. Cancer Cell 2007; 12(3): 215–229.
71. Scaruffi P, Stigliani S, Moretti S et al. Transcribed-Ultra Conserved Region expression is associated with outcome in high-risk neuroblastoma. BMC Cancer 2009; 9 : 441.
72. Sana J, Hankeova S, Svoboda M et al. Expression levels of transcribed ultraconserved regions uc.73 and uc.388 are altered in colorectal cancer. Oncology 2012; 82(2): 114–118.
73. Mestdagh P, Fredlund E, Pattyn F et al. An integrative genomics screen uncovers ncRNA T-UCR functions in neuroblastoma tumours. Oncogene 2010; 29(24): 3583–3592.
74. Catucci I, Verderio P, Pizzamiglio S et al. SNPs in ultraconserved elements and familial breast cancer risk. Carcinogenesis 2009; 30(3): 544–545; author reply 6.
75. Yang R, Frank B, Hemminki K et al. SNPs in ultraconserved elements and familial breast cancer risk. Carcinogenesis 2008; 29(2): 351–355.
76. Ji P, Diederichs S, Wang W et al. MALAT-1, a novel noncoding RNA, and thymosin beta4 predict metastasis and survival in early-stage non-small cell lung cancer. Oncogene 2003; 22(39): 8031–8041.
77. Lin R, Maeda S, Liu C et al. A large noncoding RNA is a marker for murine hepatocellular carcinomas and a spectrum of human carcinomas. Oncogene 2007; 26(6): 851–858.
78. Davis IJ, Hsi BL, Arroyo JD et al. Cloning of an Alpha-TFEB fusion in renal tumors harboring the t(6;11)(p21;q13) chromosome translocation. Proc Natl Acad Sci U S A 2003; 100(10): 6051–6056.
79. Guo F, Li Y, Liu Y et al. Inhibition of metastasis-associated lung adenocarcinoma transcript 1 in CaSki human cervical cancer cells suppresses cell proliferation and invasion. Acta Biochim Biophys Sin (Shanghai) 2010; 42(3): 224–229.
80. Fellenberg J, Bernd L, Delling G et al. Prognostic significance of drug-regulated genes in high-grade osteosarcoma. Mod Pathol 2007; 20(10): 1085–1094.
81. Koshimizu TA, Fujiwara Y, Sakai N et al. Oxytocin stimulates expression of a noncoding RNA tumor marker in a human neuroblastoma cell line. Life Sci 2010; 86 (11–12): 455–460.
82. Panzitt K, Tschernatsch MM, Guelly C et al. Characterization of HULC, a novel gene with striking up-regulation in hepatocellular carcinoma, as noncoding RNA. Gastroenterology 2007; 132(1): 330–342.
83. Matouk IJ, Abbasi I, Hochberg A et al. Highly upregulated in liver cancer noncoding RNA is overexpressed in hepatic colorectal metastasis. Eur J Gastroenterol Hepatol 2009; 21(6): 688–692.
84. Chen W, Bocker W, Brosius J et al. Expression of neural BC200 RNA in human tumours. J Pathol 1997; 183(3): 345–351.
85. Berteaux N, Lottin S, Adriaenssens E et al. Hormonal regulation of H19 gene expression in prostate epithelial cells. J Endocrinol 2004; 183(1): 69–78.
86. Eis PS, Tam W, Sun L et al. Accumulation of miR-155 and BIC RNA in human B cell lymphomas. Proc Natl Acad Sci U S A 2005; 102(10): 3627–3632.
87. Chung S, Nakagawa H, Uemura M et al. Association of a novel long non-coding RNA in 8q24 with prostate cancer susceptibility. Cancer Sci 2011; 102(1): 245–252.
88. Pasic I, Shlien A, Durbin AD et al. Recurrent focal copy-number changes and loss of heterozygosity implicate two noncoding RNAs and one tumor suppressor gene at chromosome 3q13.31 in osteosarcoma. Cancer Res 2010; 70(1): 160–171.
89. Petrovics G, Zhang W, Makarem M et al. Elevated expression of PCGEM1, a prostate-specific gene with cell growth-promoting function, is associated with high-risk prostate cancer patients. Oncogene 2004; 23(2): 605–611.
90. Srikantan V, Zou Z, Petrovics G et al. PCGEM1, a prostate-specific gene, is overexpressed in prostate cancer. Proc Natl Acad Sci U S A 2000; 97(22): 12216–12221.
91. Fu X, Ravindranath L, Tran N et al. Regulation of apoptosis by a prostate-specific and prostate cancer-associated noncoding gene, PCGEM1. DNA Cell Biol 2006; 25(3): 135–141.
92. Wang XS, Zhang Z, Wang HC et al. Rapid identification of UCA1 as a very sensitive and specific unique marker for human bladder carcinoma. Clin Cancer Res 2006; 12(16): 4851–4858.
93. Wang F, Li X, Xie X et al. UCA1, a non-protein-coding RNA up-regulated in bladder carcinoma and embryo, influencing cell growth and promoting invasion. FEBS Lett 2008; 582(13): 1919–1927.
94. Bussemakers MJ, van Bokhoven A, Verhaegh GW et al. DD3: a new prostate-specific gene, highly overexpressed in prostate cancer. Cancer Res 1999; 59(23): 5975–5979.
95. de Kok JB, Verhaegh GW, Roelofs RW et al. DD3(PCA3), a very sensitive and specific marker to detect prostate tumors. Cancer Res 2002; 62(9): 2695–2698.
96. Korneev SA, Korneeva EI, Lagarkova MA et al. Novel noncoding antisense RNA transcribed from human anti-NOS2A locus is differentially regulated during neuronal differentiation of embryonic stem cells. RNA 2008; 14(10): 2030–2037.
97. Braconi C, Valeri N, Kogure T et al. Expression and functional role of a transcribed noncoding RNA with an ultraconserved element in hepatocellular carcinoma. Proc Natl Acad Sci U S A 2011; 108(2): 786–791.
98. Yu W, Gius D, Onyango P et al. Epigenetic silencing of tumour suppressor gene p15 by its antisense RNA. Nature 2008; 451(7175): 202–206.
99. Folkersen L, Kyriakou T, Goel A et al. Relationship between CAD risk genotype in the chromosome 9p21 locus and gene expression. Identification of eight new ANRIL splice variants. PLoS One 2009; 4(11): e7677.
100. Yap KL, Li S, Munoz-Cabello AM et al. Molecular interplay of the noncoding RNA ANRIL and methylated histone H3 lysine 27 by polycomb CBX7 in transcriptional silencing of INK4a. Mol Cell 2010; 38(5): 662–674.
101. Pasmant E, Laurendeau I, Heron D et al. Characterization of a germ-line deletion, including the entire INK4//ARF locus, in a melanoma-neural system tumor family: identification of ANRIL, an antisense noncoding RNA whose expression coclusters with ARF. Cancer Res 2007; 67(8): 3963–3969.
102. Miyoshi N, Wagatsuma H, Wakana S et al. Identification of an imprinted gene, Meg3/Gtl2 and its human homologue MEG3, first mapped on mouse distal chromosome 12 and human chromosome 14q. Genes Cells 2000; 5(3): 211–220.
103. Zhang X, Zhou Y, Mehta KR et al. A pituitary-derived MEG3 isoform functions as a growth suppressor in tumor cells. J Clin Endocrinol Metab 2003; 88(11): 5119–5126.
104. Zhang X, Rice K, Wang Y et al. Maternally expressed gene 3 (MEG3) noncoding ribonucleic acid: isoform structure, expression, and functions. Endocrinology 2010; 151(3): 939–947.
105. Mourtada-Maarabouni M, Pickard MR, Hedge VL et al. GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer. Oncogene 2009; 28(2): 195–208.
106. Chooniedass-Kothari S, Hamedani MK, Troup S et al. The steroid receptor RNA activator protein is expressed in breast tumor tissues. Int J Cancer 2006; 118(4): 1054–1059.
107. Poliseno L, Salmena L, Zhang J et al. A coding-independent function of gene and pseudogene mRNAs regulates tumour biology. Nature 2010; 465(7301): 1033–1038.
108. Alimonti A, Carracedo A, Clohessy JG et al. Subtle variations in Pten dose determine cancer susceptibility. Nat Genet 2010; 42(5): 454–458.
109. Zhu Y, Yu M, Li Z et al. ncRAN, a newly identified long noncoding RNA, enhances human bladder tumor growth, invasion, and survival. Urology 2011; 77(2): 510e1–510e5.
110. Yu M, Ohira M, Li Y et al. High expression of ncRAN, a novel non-coding RNA mapped to chromosome 17q25.1, is associated with poor prognosis in neuroblastoma. Int J Oncol 2009; 34(4): 931–938.
111. Silva JM, Boczek NJ, Berres MW et al. LSINCT5 is over expressed in breast and ovarian cancer and affects cellular proliferation. RNA Biol 2011; 8(3): 496–505.
112. Gibb EA, Brown CJ, Lam WL. The functional role of long non-coding RNA in human carcinomas. Mol Cancer 2011; 10 : 38.
Labels
Paediatric clinical oncology Surgery Clinical oncologyArticle was published in
Clinical Oncology

2012 Issue 4
- Possibilities of Using Metamizole in the Treatment of Acute Primary Headaches
- Metamizole at a Glance and in Practice – Effective Non-Opioid Analgesic for All Ages
- Metamizole vs. Tramadol in Postoperative Analgesia
- Spasmolytic Effect of Metamizole
- Safety and Tolerance of Metamizole in Postoperative Analgesia in Children
-
All articles in this issue
- Význam mikroprostředí kostní dřeně v patogenezi mnohočetného myelomu
- CT kolonografie – přehled vývoje metodiky a indikací
- Dlouhé nekódující RNA a jejich význam u nádorových onemocnění
- Léčba histiocytózy z Langerhansových buněk kladribinem dosáhla u 9 z 10 dospělých pacientů dlouhodobé kompletní remise
- Mutace genu EGFR u pacientů s pokročilým NSCLC
- Projekt HARDROCK: parametrický sběr a analýza dat pacientů s nádory hlavy a krku na Klinice onkologické FN Ostrava – důraz na význam frakcionace a definice cílového objemu v radioterapii
- Jaký je prognostický význam molekulárně genetických faktorů u karcinomu endometria?
- Hepatocelulární karcinom – dlouhodobě léčitelné onemocnění
- Léčba nízkostupňového gliomu supratentoriální oblasti mozku po neurochirurgické subtotální resekci s projevy objemového up-gradingu
- Porovnávanie histologických typov primárnych a korešpondujúcich recidivujúcich bazocelulárnych karcinómov kože
- Paratestikulárny mezotelióm v mladom veku. Kazuistika
- Clinical Oncology
- Journal archive
- Current issue
- About the journal
Most read in this issue
- Hepatocelulární karcinom – dlouhodobě léčitelné onemocnění
- Léčba nízkostupňového gliomu supratentoriální oblasti mozku po neurochirurgické subtotální resekci s projevy objemového up-gradingu
- Mutace genu EGFR u pacientů s pokročilým NSCLC
- CT kolonografie – přehled vývoje metodiky a indikací
