-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Detekce DNA Neisseria meningitidis, Haemophilus influenzae a Streptococcus pneumoniae v klinickém materiálu metodou real-time PCR
Real-time PCR detection of Neisseria meningitidis, Haemophilus influenzae, and Streptococcus pneumoniae DNA in clinical specimens
The study aim was to implement a molecular real-time polymerase chain reaction (PCR) assay recommended by the CDC (Centers for Disease Control and Prevention) for the detection of Neisseria meningitidis, Haemophilus influenzae, and Streptococcus pneumoniae in clinical (culture negative) specimens from patients with suspected invasive bacterial disease. Clinical specimens are referred to the National Reference Laboratory (NRL) for Meningococcal Infections, Unit for Airborne Bacterial Infections, Centre for Epidemiology and Microbiology, National Institute of Public Health from various regions of the Czech Republic. Clinical specimens are, in particular, cerebrospinal fluid, anti-coagulated blood or serum and, exceptionally, post-mortem specimens. The NRL has implemented molecular diagnosis of these bacterial pathogens involved in meningitis and sepsis from clinical specimens since 1999. The first diagnostic method was semi-nested PCR followed by electrophoretic analysis. In 2014, a molecular qualitative real-time PCR assay was implemented.
Keywords:
rt-PCR detection of Neisseria meningitidis, Haemophilus influenzae, and Streptococcus pneumoniae – rt-PCR detection of the agents of bacterial meningitis and sepsis
Autoři: Z. Vacková 1,2; D. Lžičařová 1,3; N. K. Stock 1,4,5; J. Kozáková 1
Působiště autorů: Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha 1; 3. lékařská fakulta Univerzity Karlovy v Praze 2; Ústav lékařské mikrobiologie 2. LF UK a FN Motol 3; European Program for Public Health Microbiology (EUPHEM), ECDC, Stockholm, Sweden 4; Institute of Hygiene and Microbiology, Julius-Maximilians-University, Würzburg, Germany 5
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 64, 2015, č. 4, s. 222-230
Kategorie: Původní práce
Souhrn
Cílem práce bylo zavedení molekulární real-time PCR metodiky doporučené CDC (Centers for Disease Control and Prevention) pro účely detekce bakterií Neisseria meningitidis, Haemophilus influenzae a Streptococcus pneumoniae v klinických (kultivačně negativních) vzorcích při podezření na invazivní bakteriální onemocnění. Klinické vzorky jsou zasílány do NRL pro meningokokové nákazy Oddělení bakteriálních vzdušných nákaz CEM SZÚ z různých regionů České republiky. Jedná se především o likvor, nesrážlivou krev či sérum, výjimečně sekční materiál. NRL má zavedenou molekulární diagnostiku těchto bakteriálních původců menigitid a sepsí z klinických vzorků již od roku 1999. Původně se jednalo o seminested polymerázovou řetězovou reakci s elektroforetickým vyhodnocením. V roce 2014 byla implementována molekulární metoda real-time PCR s kvalitativním hodnocením.
Klíčová slova:
rt-PCR detekce Neisseria meningitidis, Haemophilus influenzae, Streptococcus pneumoniae – rt-PCR detekce původců bakteriálních menigitid a sepsíÚVOD
Neisseria meningitidis, Streptococcus pneumoniae i Haemophilus influenzae patří celosvětově k velice významným patogenům. Jedná se o bakterie evolučně blízce nepříbuzné, nicméně sdílející některé vlastnosti významné pro ekologii a chování v organismu hostitele. Především jde o antigenní variabilitu určující velké množství séroskupin a sérotypů těchto bakterií, celosvětové rozšíření s odlišnou distribucí séroskupin či sérotypů, nebo schopnost vyvolávat různé formy interakcí s lidským organismem. Často představují běžnou bakteriální flóru, jindy vyvolávají lehká respirační onemocnění. Jsou však schopny vyvolat i život ohrožující invazivní onemocnění, jakými jsou meningitidy, bakterémie, sepse či pneumonie.
Implementovaná molekulární metoda je určena pro diagnostiku invazivních bakteriálních onemocnění. Umožňuje detekci agens v kratším časovém intervalu, detekuje i nízkou koncentraci DNA ve vzorku (včetně kultivačně negativních, i několik dní po zahájení antibiotické terapie).
Národní referenční laboratoř pro meningokokové nákazy (NRL MEN) Oddělení vzdušných bakteriálních nákaz Centra epidemiologie a mikrobiologie (CEM) Státního zdravotního ústavu (SZÚ), má již od roku 1999 zavedenu seminested polymerázovou řetězovou reakci (PCR) pro detekci Neisseria meningitidis se sérotypizací B a C [1]. Roku 2003 byla metodika rozšířena o detekci Streptococcus pneumoniae, Haemophilus influenzae a sérotypizaci Neisseria meningitidis o séroskupiny A, W, Y [2]. Implementovaná metoda real-time PCR (rt-PCR) v rámci Oddělení vzdušných bakteriálních nákaz CEM SZÚ nahradí původní metodiku identifikace Neisseria meningitidis, Streptococcus pneumoniae a Haemophillus influenzae přímo z klinických vzorků při podezření na invazivní bakteriální onemocnění. Tato metodika je společně s ostatními nabízena lékařům. Doporučení na typ, odběr a transport materiálu viz http://www.szu.cz/odd-vzdusne-nakazy-doporucene-postupy.
Principem této kvalitativní metody rt-PCR je amplifikace úseku DNA obsahujícího cílovou sekvenci a detekce této cílové sekvence pomocí hybridizace oligonukleotidu značeného fluorescenčním barvivem. Intenzita fluorescence je úměrná množství amplifikovaného úseku v reakci, měří se průběžně po dobu trvání reakce za použití optického detekčního systému rt-PCR cykléru. Jako cílové sekvence pro PCR identifikaci Neisseria meningitidis, Streptococcus pneumoniae i Haemophillus influenzae mohou být použity různé geny, jenž dané bakteriální genomy specificky obsahují. Jedná se především o geny ctrA, crgA, sodC, ply, psaA, lytA, ompP2, hpd, bexA [3-8]. Pro každé bakteriální agens byl pro detekci vybrán jeden specifický gen, jednalo se vždy o gen vyskytující se v ideálním případě specificky, vždy pouze u daného druhu napříč séroskupinami či sérotypy a nezávisle na přítomnosti či nepřítomnosti pouzdra. Metoda je navržena na detekci cílové sekvence genu sodC u Neisseria meningitidis. Tento gen je obsažen v genomu opouzdřených i neopouzdřených kmenů. SodC gen kóduje Cu, Zn superoxiddismutázu. Detekce Haemophilus influenzae je namířena na cílovou sekvenci genu hpd kódujícího protein D. Jedná se o gen přítomný v genomu opouzdřených i neopouzdřených kmenů. Identifikace Streptococcus pneumoniae probíhá detekcí cílové sekvence genu lytA kódujícího autolysin. Sekvence jsou vysoce specifické pro příslušné bakteriální druhy. Použití vybraných genů při PCR identifikaci Neisseria meningitidis, Streptococcus pneumoniae i Haemophilus influenzae je celoevropsky doporučeno CDC [9].
MATERIÁL A METODIKA
Kmeny
Pro potřeby testování metodiky, možných nespecifických reakcí i velice důležitou přípravu pozitivních interních kontrol pro rt-PCR se použily kmeny ze sbírek NRL pro meningokokové nákazy, NRL pro streptokokové nákazy, NRL pro hemofilové nákazy, vše Oddělení bakteriálních vzdušných nákaz CEM SZÚ v Praze a České národní sbírky typových kultur (CNCTC) SZÚ v Praze. Testování bylo zaměřeno na kmeny rodů Neisseria, Haemophilus a Streptococcus společně s kmeny rodově příbuzných bakterií či kmeny představující riziko záměny při klinických projevech. Celkově bylo testováno 70 kmenů (tab. 1).
Tab. 1. Souhrnný přehled testovaných kmenů a klinických vzorků Table 1. Summary review of strains and clinical specimens tested 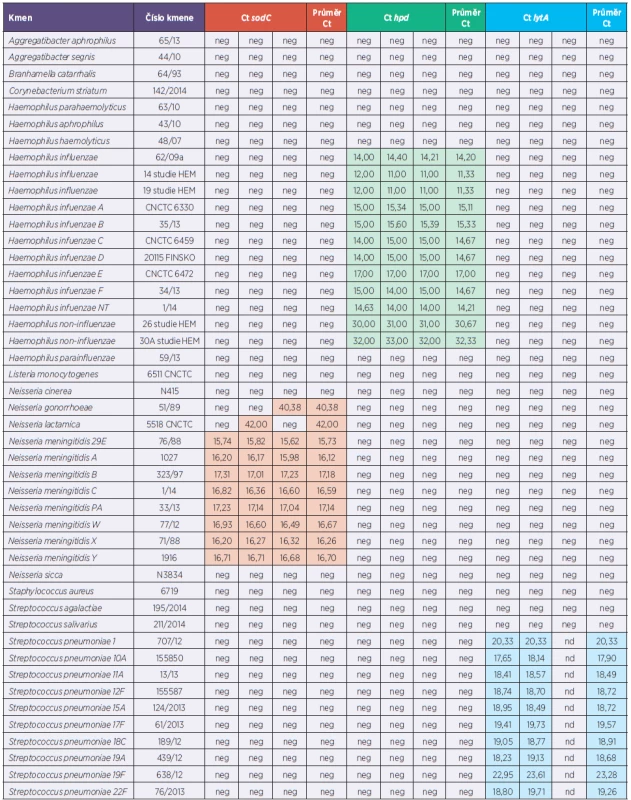

Barevně označeny pozitivitní záchyty (červená – N. meningitidis, zelená – H. influenzae, modrá – S. pneumoniae) neg* v těchto případech se liší výsledek seminested PCR a rt-PCR (seminested PCR pozitivní, rt-PCR negativní) Positive capturing – colour marks (red – N. meningitidis, green – H. influenzae, blue – S. pneumoniae) neg* in these cases the results seminested PCR and rt-PCR are different (seminested PCR positive, rt-PCR negative) Klinické vzorky
Při procesu implementace rt-PCR metody určené k detekci DNA Neisseria meningitidis, Haemophilus influenzae a Streptococcus pneumoniae v klinickém materiálu byly použity primárně sterilní klinické kultivačně negativní vzorky zasílané do NRL pro meningokokové nákazy s požadavkem identifikace jednoho či všech tří zmiňovaných patogenů při podezření na invazivní bakteriální onemocnění.
Druhy testovaného materiálu: mozkomíšní mok, krev nesrážlivá, krev srážlivá – sérum, pleurální tekutina, hemokultivační nádobky Bact/Alert, pitevní materiál (petechie). Metodika je však validována pro mozkomíšní mok a nesrážlivou krev, ostatní typy materiálu byly testovány jen v minimálním počtu vzorků. Celkově bylo testováno 55 klinických vzorků (viz tab. 1).
Postup izolace DNA
Postup je jednotný pro izolaci z klinických materiálů i bakteriálních kultur. Postup izolace DNA je prováděn pomocí izolačního kitu QIAmp DNA Mini Kit (QIAGEN), po předchozí enzymatické preinkubaci. Využívají se enzymy lyzozym (Sigma Aldrich), mutanolyzin (Sigma Aldrich) a proteináza K (QIAGEN) [10].
Izolovaná DNA se skladuje krátkodobě po dobu vyšetřování vzorku při 2–8 °C, za účelem dlouhodobé archivace vyšetřeného materiálu při -20 °C.
Postup provedení rt-PCR reakce
Reakční směs použitá pro PCR má následující složení: TaqMan Universal Master Mix kit (ABI), specifické primery a značená sonda (Generi Biotech). Sekvence a finální koncentrace použitých primerů a sond byly přijaty podle CDC doporučení [9] a jsou uvedeny v tabulce 2. Sondy jsou značené FAM-BHQ1. K 16 µl reakční směsi (objemové složení reakční směsi: 10 µl master mixu + 2 µl forward primeru + 2 µl reverse primeru + 2 µl sondy) se přidávají 4 µl izolované DNA vzorku či kontroly. Amplifikace byla prováděná v Rt-PCR termocykléru CFX 96 (BIO-RAD) s detekčním systémem tvořeným 6 fotodiodami LED (rozmezí vlnových délek pro oblast excitace 450–684 nm, rozmezí vlnových délek pro oblast emise 515–730 nm). Byl použit následující amplifikční program s detekcí v kroku hybridizace: počáteční denaturace 50 °C/2 min, 95 °C/10 min, následně opakování 45 cyklů 95 °C/15 sec, 60 °C/1 min (detekce) a nakonec ochlazení na 4 °C do zastavení.
Tab. 2. Primery a sondy pro rt-PCR Table 2. Primers and probes for rt-PCR 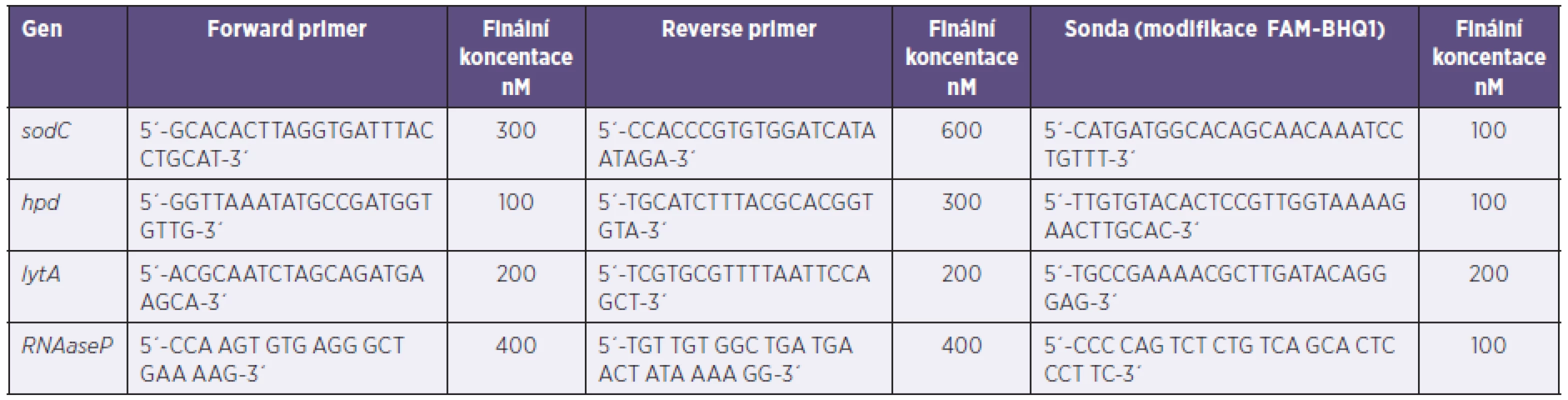
Každý vzorek je pro každou sekvenci testován v tripletu. Pro každou sekvenci jsou zařazeny tři kontroly (negativní, izolační, pozitivní) testované v tripletu. Při každém pokusu se tedy používá neznámý klinický vzorek (izolovaná DNA z neznámého klinického vzorku), negativní izolační kontrola (NIK – vzorek PCR H2O, která prošla postupem izolace DNA souběžně s klinickým vzorkem), negativní kontrola (NTC – vzorek PCR H2O, kontrola která neobsahuje templátovou DNA), pozitivní kontrola (PK – izolovaná DNA referenčních bakteriálních kmenů Neisseria meningitidis, Haemophilus influenzae či Streptococcus pneumoniae). Kontrola extrakce a inhibice se provádí detekcí humánní DNA v každém klinickém vzorku. Kontrola je zařazena jako čtvrtá cílová sekvence RNAsaP [11] a testuje se ve stejném režimu (nutnost zařazení tří typů kontrol, pozitivní kontrolou je izolovaná DNA ze známého humánního klinického vzorku).
Přístupová čísla cílových sekvencí v GenBank
sodC – GenBank AE002098.2, hpd – Z35661.1, lytA – GenBank AE005672, RNAsaP – GenBank NM_006413
VALIDAČNÍ PARAMETRY METODY
Vytvoření standardní křivky a stanovení odvozených charakteristik rt-PCR testu
Pro vytvoření standardní křivky byla provedena rt-PCR měření podle popsaného protokolu v sérii ředění DNA o známé počáteční koncentraci (měřeno spektrofotometrem Nanodrop 1000) s koeficientem 10. Standardní křivka byla vytvořena v programu BIO-RAD CFX Manager (v grafu na ose x vyneseny hodnoty dekadického logaritmu jednotlivých ředění, počáteční koncentrace označena jako 1, na ose y hodnoty Ct (cycle treshold) pro jednotlivá měření). Oblast linearity, směrnice přímky v oblasti linearity (slope), efektivita E a korelační koeficient R2 byly též stanoveny pomocí programu BIO-RAD CFX Manager.
Stanovení dolního limitu detekce (LLOD)
- Stanovení limitu detekce jako počtu kopií DNA v reakci a v objemové jednotce:
Rt-PCR detekce DNA podle popsaného protokolu v sérii ředění DNA s koeficientem 10. Limit detekce byl odvozen od nejvyššího ředění, ve kterém byla DNA spolehlivě detekována (pozitivní amplifikace ve 2 ze 3 replikátů a zároveň ve 2 oddělených rt-PCR pokusech). Měření koncentrace DNA v neředěném vzorku bylo provedeno pomocí spektrofotometru (Nanodrop 1000), koncentrace v ředěných vzorcích byla odvozena výpočtem. Počet kopií DNA v objemové jednotce příslušného ředění byl odvozen výpočtem podle vzorce
CDNA = koncentrace DNA v příslušném ředění v jednotkách g/µl
6,023 . 1023 = Avogadrova konstanta udávající počet částic v 1 molu látky
650 = průměrná molární hmotnost 1 páru bazí v molekule DNA (g/mol)
délka genomu = vyjádřená jako počet párů bazí (bp), délka genomu Neisseria meningitidis séroskupina B [12], délka genomu Haemophilus influenzae NT [13], délka genomu Streptococcus pneumoniae sérotyp 3 [14].
Počet kopií DNA v reakci pro příslušné ředění byl získán vynásobením počtu kopií DNA v objemové jednotce a počtu objemových jednotek v reakci podle uvedeného protokolu (4x).
- Stanovení limitu detekce v CFU (colony forming unit)/ml:
Detekce rt-PCR DNA podle popsaného protokolu v sérii ředění (koeficient 10) živé bujónové kultury N. meningitidis, H. influenzae a S. pneumoniae. Limit detekce je odvozen od nejvyššího ředění, ve kterém byla spolehlivě detekována DNA, a od nejvyššího ředění, pro které byl pozorován pozitivní kultivační nález.
Neisseria meningitidis – použit kmen 58/14, séroskupina B, ze sbírky NRL MEN. Z čerstvé kultury (Čokoládový Müller Hinton agar – ČMH (BIO-RAD), inkubace v atmosféře se zvýšenou tenzí 5% CO2, teplota 37 °C, 24 hod) připravena suspenze v Brain Heart Infusion – BHI (OXOID), stupeň zákalu 2 podle McFarlanda. Alikvóty po 1 ml suspenze byly zmrazeny při -70 °C. Z 1 alikvóty bylo připraveno 12–15 ředění (koeficient 10) ve sterilním BHI, z každého ředění vyočkováno 100 µl suspenze na ČMH agar, rozočkováno po celé ploše půdy a inkubováno za výše uvedených podmínek. Ve zbylé suspenzi byly bakterie usmrceny 10minutovou inkubací při teplotě 100 °C a následovala extrakce a rt-PCR detekce DNA podle výše popsaného protokolu.
Po proběhlé inkubaci byly pro jednotlivá ředění spočteny vyrostlé kolonie a podle výsledku rt-PCR pokusu bylo určeno nejvyšší ředění, ve kterém byla spolehlivě detekována bakteriální DNA (kritéria uvedena výše). Limit detekce byl vyjádřen jako počet CFU/ml v nejvyšším ředění, v němž byla spolehlivě detekována bakteriální DNA ve dvou nezávislých pokusech.
Haemophilus influenzae – použit kmen 1/14, netypovatelný NT, ze sbírky NRL HEM. Z čerstvé kultury (čokoládový selektivní agar pro hemofily (OXOID), inkubace v atmosféře se zvýšenou tenzí 5% CO2, teplota 35 °C, 24 hod) připravena suspenze v Levinthalově bujónu, stupeň zákalu 0,5 podle McFarlanda, naředěna 10krát. Alikvóty po 1 ml naředěné suspenze zmrazeny při -70 °C. Postup při přípravě a zpracování série ředění viz N. meningitidis, použitá kultivační média a podmínky inkubace viz H. influenzae.
Streptococcus pneumoniae – použit kmen 199/12, sérotyp 4, ze sbírky NRL STR. Z čerstvé kultury (Columbia krevní agar (OXOID), 7 % beraních erytrocytů, inkubace v atmosféře se zvýšenou tenzí 5% CO2 , teplota 35 °C, 24 hod) připravena suspenze v BHI, stupeň zákalu 0,5 podle McFarlanda, naředěna 10krát. Alikvóty po 1 ml naředěné suspenze zmrazeny při -70 °C. Postup při přípravě a zpracování série ředění viz N. meningitidis, použitá kultivační média a podmínky inkubace viz S. pneumoniae.
Stanovení přesnosti (opakovatelnosti, repeatability) v oblasti dolního limitu detekce
Pro stanovení přesnosti v oblasti dolního limitu detekce bylo provedeno měření nejnižšího ředění, v němž byla spolehlivě detekována DNA příslušného bakteriálního původce, v 10 opakováních. Z naměřených hodnot byl vypočten variační koeficient.
Stanovení mezilehlé přesnosti (reprodukovatelnosti, reproducibility)
Pro stanovení mezilehlé přesnosti byly využity hodnoty Ct pozitivních kontrol téže šarže získané při 20 různých měřeních. Pozitivní kontroly se stejně jako vzorek měří v tripletu, proto byl pro každé měření nejprve spočten aritmetický průměr tří získaných hodnot Ct, variační koeficient byl pak vypočten z 20 průměrných hodnot Ct spočtených pro jednotlivá měření.
Ověření senzitivity a specificity
Ověření senzitivity: Testováno s kulturami N. meningitidis, H. influenzae a S. pneumoniae (viz tab 1). Ověření specificity in-silico: Provedeno pomocí NCBI BLAST.
Experimentální ověření specificity: Ověřeno s kulturami příbuzných bakteriálních druhů a druhů, které mohou být původci invazivních bakteriálních onemocnění (viz tab. 1).
Interpretace výsledků rt-PCR
Probíhá hodnocení hodnot Ct (cycle treshold). Vyšetření se považuje za validní, pokud hodnota Ct pozitivních kontrol odpovídá hodnotám naměřeným při jejich testování (používáno v rozmezí 30–36), NTC kontrola a negativní izolační kontrola jsou analytickým systémem vyhodnoceny jako negativní (NT – no template) a ve vzorku je detekována humánní DNA (pozitivita pro cílovou sekvenci RNAsaP).
Výsledek vyšetření vzorku se hodnotí jako pozitivní pro příslušnou cílovou sekvenci, pokud hodnota Ct je menší než 40 alespoň ve dvou ze tří replikátů.
Výsledek vyšetření vzorku se hodnotí jako negativní pro příslušnou cílovou sekvenci, pokud je vzorek analytickým systémem vyhodnocen jako negativní (NT – no template) nejméně ve dvou ze tří replikátů a pokud se při kontrole extrakce a inhibice ve vzorku prokáže přítomnost humánní DNA (cílová sekvence RNAsaP).
VÝSLEDKY VALIDACE A TESTOVÁNÍ
Stanovení dolního limitu detekce – počet kopií DNA v reakci a v objemové jednotce
Pro stanovení LLOD pro druh Neisseria meningitidis (sodC) byl použit kmen 323/97, sérotyp B. Hodnota LLOD byla stanovena s výsledkem 1,21 . 102 kopií/µl, což odpovídá 4,8 . 102 kopií v reakci (podle protokolu – 4 µl templátu v reakčním objemu 20 µl).
Pro stanovení LLOD pro druh Haemophilus influenzae (hpd) byl použit kmen 1/14, netypovatelný kmen. Hodnota LLOD byla 52,2 kopií/µl, to odpovídá 208,8 kopií v reakci.
Pro stanovení LLOD pro druh Streptococcus pneumoniae (lytA) byl použit kmen 702/14, sérotyp 3. Hodnota LLOD byla 59,2 kopií/µl, to odpovídá 236,8 kopií v reakci.
Stanovení dolního limitu detekce – CFU/ml
Neisseria meningitidis 58/14, sérotyp B – v pokusu č. 1 LLOD 1 CFU/ml, v pokusu č. 2 LLOD 1 CFU/ml.
Haemophilus influenzae 1/14, netypovatelný kmen – v pokusu č. 1 LLOD 40 CFU/ml, v pokusu č. 2 LLOD 30 CFU/ml.
Streptococcus pneumoniae 199/12, sérotyp 4 – v pokusu č. 1 LLOD 10 CFU/ml, v pokusu č. 2 LLOD 5 CFU/ml.
Stanovení přesnosti (opakovatelnosti, repeatability) v oblasti dolního limitu detekce (počet kopií DNA v objemové jednotce)
Neisseria meningitidis B, 323/97 – aritmetický průměr z 10 naměřených hodnot Ct – 38,184; variační koeficient 0,026.
Haemophilus influenzae NT, 1/14 – aritmetický průměr z 10 naměřených hodnot Ct – 37,418; variační koeficient 0,024.
Streptococcus pneumoniae 3, 702/14 – aritmetický průměr z 10 naměřených hodnot Ct – 38,872; variační koeficient 0,026.
Stanovení mezilehlé přesnosti (reprodukovatelnosti, reproducibility)
Neisseria meningitidis PA, 33/14 – 20 měření, variační koeficient 0,030.
Haemophilus influenzae NT, 1/14 – 20 měření, variační koeficient 0,037.
Streptococcus pneumoniae 6C, 105/14 – 20 měření, variační koeficient 0,041.
Charakteristiky rt-PCR testu odvozené od standardní křivky
- Detekce DNA Neisseria meningitidis, cílová sekvence sodC
Oblast linearity: Standardní křivka je lineární pro měření koncentrací DNA v rozsahu
1,21 . 102 – 1,21 . 106 kopií/µl.
Odvozené charakteristiky: směrnice přímky (slope) = -3,918; efektivita E = 80 %; korelační koeficient R2 = 0,998
- Detekce DNA Haemophilus influenzae, cílová sekvence hpd
Oblast linearity: Standardní křivka je lineární pro měření koncentrací DNA v rozsahu
52,2–5,22 . 106 kopií/µl.
Odvozené charakteristiky: směrnice přímky (slope) = -4,046; efektivita E = 76,7 %; korelační koeficient R2 = 0,998
- Detekce DNA Streptococcus pneumoniae, cílová sekvence lytA
Oblast linearity: Standardní křivka je lineární pro měření koncentrací DNA v rozsahu
59,2–5,92 . 106 kopií/µl.
Odvozené charakteristiky: směrnice přímky (slope) = -3,834; efektivita E = 82,3 %; korelační koeficient R2 = 0,991
Testování analytické specificity
Výsledky přehledně shrnuje tabulka 1.
Testování klinického materiálu
Výsledky přehledně shrnuje tabulka 1.
DISKUSE
Výsledky testování zvolené doporučené metody rt-PCR detekce DNA tří potenciálních původců invazivního bakteriálního onemocnění byly zhodnoceny z hlediska požadavků kladených na validaci tohoto typu molekulárně genetických metod a konečná podoba testu prováděného v rutinní praxi byla zvolena též s ohledem na patogenezi onemocnění.
Metoda byla zaváděna pro diagnostiku invazivního bakteriálního onemocnění a druhy vyšetřovaného materiálu byly zvoleny s přihlédnutím k evropským „case“ definicím invazivních meningokokových, hemofilových a pneumokokových onemocnění. Primárně se vyšetřuje sterilní klinický materiál, průkaz infekčního agens je proto bez ohledu na kvantitu jednoznačně podpůrnou informací pro diagnózu invazivního onemocnění. Byla proto zvolena metoda kvalitativní detekce bakteriální DNA, kde se předpokládá pro každou detekovanou sekvenci dvojí možný závěr vyšetření – pozitivní nebo negativní nález DNA. Množství detekované DNA ve vzorku nekoreluje spolehlivě se závažností klinického stavu. Infekce více než jedním původcem je u invazivních bakteriálních onemocnění velmi vysoce nepravděpodobná a při testování nebyla tato možnost brána v úvahu.
Stanovení limitu detekce jako koncentrace CFU v objemové jednotce bylo provedeno pro ověření celého postupu včetně extrakce DNA.
Specificita testů pro jednotlivá detekovaná agens byla ověřena orientačně vyhledáním sekvencí jednotlivých oligonukleotidů ve vyhledávači NCBI (BLAST). Pro všechny oligonukleotidy byla potvrzena 100% shoda v sekvenci pouze v hledané oblasti genomu příslušných agens. Při testování specificity testu s použitím vzorků DNA izolované z jiných bakteriálních druhů byla pro sodC naměřena hodnota Ct pro 1 kmen Neisseria gonorrhoeae a 1 kmen Neisseria lactamica. V obou případech byly hodnoty Ct vyšší než arbitrárně stanovená hodnota cut-off, výsledek tedy může být hodnocen jako negativní. Pro druh Neisseria meningitidis byly při stanovení LLOD v počtu kopií DNA v objemové jednotce naměřeny pro nejnižší spolehlivě detekovanou koncentaci DNA 1,21 . 102 kopií/µl naměřeny hodnoty Ct v rozmezí 36,00–39,00. Oba zmíněné druhy jsou geneticky blízce příbuzné druhu Neisseria meningitidis, navíc mezi N. lactamica a N. meningitidis, blízce příbuznými druhy osidlujícími shodně sliznice horních dýchacích cest, dochází k horizontálnímu transferu genů [15]. Druh Neisseria gonorrhoeae je primárním patogenem a může být původcem invazivních onemocnění, Neisseria lactamica je součástí běžné orofaryngeální flóry a nepovažuje se za možného původce invazivního onemocnění v běžné populaci. U všech testovaných kmenů Neisseria meningitidis i klinických materiálů invazivních meningokokových onemocnění byla u sodC pozitivní reakce, což potvrdilo vhodnost výběru daného genu oproti genu ctrA, u něhož je popisována negativita až u 16 % kmenů Neisseria meningitidis [16]. Pro hpd byly hodnoty Ct naměřeny pouze pro kmeny náležející k druhu Haemophilus influenzae, což ukazuje na vysokou druhovou specificitu genu. Pro lytA byly hodnoty Ct naměřeny u 2 z 9 testovaných kmenů Streptococcus mitis. Streptococcus mitis je geneticky blízce příbuzný druhu Streptococcus pneumoniae, v jeho genomu může být přítomen funkční gen pro autolyzin. Whatmore et al. testovali různé cílové sekvence z oblasti genu pro autolyzin na velkém souboru kmenů patřících k druhům streptokoků ze skupiny „mitis group“, pro některé z testovaných kmenů byla zjištěna pozitivita i pro sekvenci používanou v implementovaném testu [17].
Pro testování metody na vzorcích klinického materiálu, kultivačně negativního, byly vybrány vzorky testované též seminested PCR metodou s elektroforetickou vizualizací produktů používanou původně v NRL MEN. Výsledky obou metod byly porovnány. Rozdílné výsledky byly pozorovány u 4 vzorků ze 55 vzorků testovaných. Jednalo se o dva vzorky séra a dva vzorky likvoru. U všech rozdílných případů byl výsledek seminested PCR pozitivní a rt-PCR negativní. Vzorky likvoru byly více než deset let skladovány v mrazu, což pravděpodobně ovlivnilo výsledky. Vzorky séra byly krátkodobě skladovány v mrazu a tato skutečnost mohla výsledky taktéž ovlivnit, avšak i podle jiných autorů je rt-PCR detekce DNA ve vzorcích séra méně spolehlivá. V našem souboru byl testován příliš malý počet vzorků séra k tomu, aby bylo možno vyvodit závěr, že nejde o materiál vhodný k testování. Kromě mozkomíšního moku byly testovány taktéž další druhy materiálu, byly však k dispozici ve velmi malém počtu vzorků. Tyto druhy materiálu budou k vyšetření přijímány, metoda však pro ně zatím nemohla být validována. Předchozí testování simulovaných vzorků nesrážlivé krve (data nejsou prezentována) ukázalo, že nesrážlivá krev je též vhodným materiálem pro testovanou metodu.
Použitím rt-PCR metodiky se podařilo velmi zkrátit časovou náročnost diagnostiky závažných invazivních bakteriálních meningokokových, hemofilových a pneumokokových onemocnění až na hranici pěti hodin. Dřívější diagnostika založená na metodě seminested PCR vyžadovala průměrně 9 hodin a kultivační vyšetření 24 hodin.
ZÁVĚR
Metoda rt-PCR detekce DNA Neisseria meningitidis, Haemophilus influenzae a Streptococcus pneumoniae byla prověřena testováním sbírkových bakteriálních kmenů i klinického kultivačně negativního materiálu v širokém rozsahu. Byly stanoveny validační parametry metody. Podle výsledů je metoda vhodná pro testování vzorků primárně sterilního klinického materiálu, především mozkomíšního moku a nesrážlivé krve. Implementovaná metoda je určena pro diagnostiku invazivních bakteriálních onemocnění. Molekulární metoda umožňuje detekci agens v kratším časovém intervalu a detekuje i nízkou koncentraci DNA ve vzorku (včetně kultivačně negativních, i několik dní po zahájení antibiotické terapie) a je doporučována k rutinnímu užívání. Provádění těchto molekulárních vyšetření má velký význam, vzhledem ke stále se zvyšujícímu procentu invazivních bakteriálních onemocnění kultivačně negativních, laboratorně diagnostikovaných pouze metodami PCR.
Podpořeno MZ ČR - RVO („Státní zdravotní ústav - SZU, 75010330“).
Do redakce došlo dne 3. 11. 2014.
Adresa pro korespondenci:
Mgr. Zuzana Vacková
SZÚ Praha
Šrobárova 48
100 42 Praha 10
e-mail: zuzana.vackova@szu.cz
Zdroje
1. Kalmusová J, Pavlíková V, Křížová P, et al. První výsledky PCR diagnostiky invazivního meningokokového onemocnění. Klinická mikrobiologie a infekční lékařství, 2001;7(1):15–19. ISSN 1211-264X.
2. Kalmusová J, Bronská E, Křížová P. Diagnostika invazivního meningokokového, hemofilového a pneumokokového onemocnění PCR metodou. Klinická mikrobiologie a infekční lékařství, 2004;10 : 130–133. ISSN 1211-264X.
3. Carvalho MG, Tondella ML, McCaustland K, Weidlich L, McGee L, Mayer LW, Steigerwalt A, Whaley M, Facklam RR, Fields B, Carlone G, Ades EW, Dagan R, Sampson JS. Evaluation and improvement of real-time PCR assays targeting lytA, ply, and psaA genes for detection of pneumococcal DNA. Journal of Clinical Microbiology, 2007; 45(8):2460–2466. ISSN 0095-1137.
4. Messmer TO, Sampson JS, Stinson A, Wong B, Carlone GM, Facklam RR. Comparison of four polymerase chain reaction assays for specificity in the identification of Streptococcus pneumoniae. Diagnostic Microbiology and Infectious Disease, 2004;49 : 249–254. ISSN 0732-8893.
5. Mothershed EA, Sacchi CT, Whitney AM, Barnett GA, Ajello GW, Schmink S, Mayer LW, Phelan M, Taylor TH Jr, Bernhardt SA, Rosenstein NE, Popovic T. Use of real-time PCR to resolve slide agglutination discrepancies in serogroup identification of Neisseria meningitidis. Journal of Clinical Microbiology, 2004;42(1): 320–328. ISSN 0095-1137.
6. Taha MK, Alonso JM, Cafferkey M, Caugant DA, Clarke SC, Diggle MA, Fox A, Frosch M, Gray SJ, Guiver M, Heuberger S, Kalmusova J, Kesanopoulos K, Klem AM, Kriz P, Marsh J, Mölling P, Murphy K, Olcén P, Sanou O, Tzanakaki G, Vogel U. Interlaboratory comparison of PCR-based identification and genogrouping of Neisseria meningitidis. Journal of Clinical Microbiology, 2005;43(1):144–149. ISSN 0095-1137.
7. Wang X, Mair R, Hatcher C, Theodore MJ, Edmond K, Wu HM, Harcourt BH, Carvalho MG, Pimenta F, Nymadawa P, Altantsetseg D, Kirsch M, Satola SW, Cohn A, Messonnier NE, Mayer LW. Detection of bacterial pathogens in Mongolia meningitis surveillance with a new real-time PCR assay to detect Haemophilus influenzae. International Journal of Medical Microbiology, 2011;301 : 303–309. ISSN 1438-4221.
8. Corless CE, Guiver M, Borrow R, Edwards-Jones V, Fox AJ, Kaczmarski EB. Simultaneous Detection of Neisseria meningitidis, Haemophilus influenzae, and Streptococcus pneumoniae in Suspected Cases of Meningitis and Septicemia Using Real-Time PCR. Journal of Clinical Microbiology, 2001;39(4):1553–1558. ISSN 0095-1137.
9. WHO. PCR for Detection and Characterization of Bacterial Meningitis Pathogens: Neisseria meningitidis, Haemophilus influenzae, and Streptococcus pneumoniae, in Laboratory Methods for the Diagnosis of Meningitis caused by Neisseria meningitidis, Streptococcus pneumoniae and Haemophilus influenzae. WHO manual [online]. 2011, 2nd edition, Chapter 10. Dostupné na www: http://www.cdc.gov/meningitis/bacterial.html
10. CDC. Blood and Body Fluid DNA Extraction for Streptococci. CDC doporučení [online]. 2014, Dostupné na www: http://www.cdc.gov/streplab/downloads/pcr-body-fluid-DNA-extract-strep.pdf.
11. Emery SL, Erdman DD, Bowen MD, et al. Real-Time Reverse Transcriptiom-Polymerase Chain Reaction Assay for SARS-associated Coronavirus. Emerging Infectious Diseases, 2004;10(2):311–316. ISSN 1080-6040.
12. Piet JR, Huis in ’t Veld RAG, van Schaik BDC, et al. Genome Sequence of Neisseria meningitidis Serogroup B Strain H44/76. Journal of Bacteriology, 2011;193(9):2371–2372. ISSN 1098-5530.
13. Harrison A, Dyer DW, Gillaspy A, et al. Genomic Sequence of an Otitis Media Isolate of Nontypeable Haemophilus influenzae: Comparative Study with H. influenzae Serotype d, Strain KW20. Journal of Bacteriology, 2005;187(13): 4627–4636. ISSN 1098-5530.
14. Hiller NL, Janto B, Hogg JS, et al. Comparative Genomic Analyses of Seventeen Streptococcus pneumoniae Strains: Insights into the Pneumococcal Supragenome. Journal of Bacteriology, 2007;189(22):8186–8195. ISSN 1098-5530.
15. Hanage WP, Fraser C, Spratt BG. Fuzzy species among recombinogenic bacteria. BMC Biology, 2005;3 : 3–6. ISSN 1741-7007.
16. Dolan TJ, Hatcher CP, Satterfield DA, et al. sodC-Based Real-Time PCR for Detection of Neisseria meningitidis. PLoS ONE, 2011;6(5): e19361. ISSN 1932-6203.
17. Whatmore AW, Efstratiou A, Pickerill AP, et al. Genetic Relationships between Clinical Isolates of Streptococcus pneumoniae, Streptococcus oralis, and Streptococcus mitis: Characterization of “Atypical” Pneumococci and Organisms Allied to S. mitis Harboring S. pneumoniae Virulence Factor-Encoding Genes. Infection and Immunity, 2000;68(3):1374–1382. ISSN 1098-5522.
Štítky
Hygiena a epidemiológia Infekčné lekárstvo Mikrobiológia
Článek Syndrom toxického šokuČlánek Rejstříky
Článok vyšiel v časopiseEpidemiologie, mikrobiologie, imunologie
Najčítanejšie tento týždeň
2015 Číslo 4- Parazitičtí červi v terapii Crohnovy choroby a dalších zánětlivých autoimunitních onemocnění
- Očkování proti virové hemoragické horečce Ebola experimentální vakcínou rVSVDG-ZEBOV-GP
- Koronavirus hýbe světem: Víte jak se chránit a jak postupovat v případě podezření?
-
Všetky články tohto čísla
- Úloha Streptococcus mutans v orálnom biofilme
- Onemocnění člověka způsobená hantaviry – stále opomíjené zoonózy?
- Imunitní odpověď v patogenezi infekce virem hepatitidy C
- Patogeneze klíšťové encefalitidy a možnosti antivirové terapie
- Syndrom toxického šoku
- Detekce DNA Neisseria meningitidis, Haemophilus influenzae a Streptococcus pneumoniae v klinickém materiálu metodou real-time PCR
- Fekální bakterioterapie v léčbě rekurentní kolitidy způsobené Clostridium difficile na Klinice infekčních chorob Fakultní nemocnice Brno v letech 2010–2014 – prospektivní studie
- Benigní akutní myozitidy u dětí jako komplikace chřipky B a jejich diferenciální diagnostika
- Epidemie příušnic v Plzeňském kraji v roce 2011
- Daniel Růžek a kolektiv: Klíšťová encefalitida
- Rejstříky
- Epidemiologie, mikrobiologie, imunologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Benigní akutní myozitidy u dětí jako komplikace chřipky B a jejich diferenciální diagnostika
- Onemocnění člověka způsobená hantaviry – stále opomíjené zoonózy?
- Syndrom toxického šoku
- Úloha Streptococcus mutans v orálnom biofilme
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy





