-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Identifikace nových molekulárních markerů pro sledování minimální reziduální nemoci u akutních leukemií
Identification of new molecular markers for minimal residual disease assessment in acute leukemia patients
Acute leukaemia (AL) comprises a heterogeneous group of haematological malignancies with varying prognoses. In light of this heterogeneity, individual patient response to treatment can be difficult to predict. Sensitive monitoring of residual leukemic cell populations (minimal residual disease – MRD) is thus very important and holds great potential for improving treatment strategies. Commonly used MRD targets include recurrent cytogenetic abnormalities and mutations in important haematological genes. Unfortunately, such targets are identified in a majority of adult ALL patients but only in about 50% of adult AML patients. Identification of new specific leukemic blast molecular markers for MRD assessment is therefore highly desirable. Our goal was to develop a unique technical approach for the identification and mapping of clone-specific chromosomal abnormalities down to the single nucleotide level using current molecular cytogenetic techniques, particularly multicolour fluorescence in situ hybridization, multicolour chromosome banding (mFISH, mBAND) and multiplex hybridization of fluorescently labelled BAC clones (BAC-FISH). Higher resolution was achieved by hybridization of fluorescent probes to combed DNA fibres (molecular combing, fibre-FISH). Another approach used for the precise identification of chromosomal breakpoints was chromosome micro dissection followed by next-generation sequencing (NGS) of the dissected material. Finally, a specific Real-Time PCR assay to monitor MRD was designed. Modern technologies open new vistas for the detection and identification of unique clone-specific abnormalities in AL patients. Our work clearly suggests that mapping from the chromosomal level down to the nucleotide level is feasible and readily applicable in eligible AL patients, allowing its´ use in standard clinical practice and as a tool for personalized „tailor-made“ medicine.
Key words:
acute leukaemia, minimal residual disease, cytogenetics, molecular cytogenetics, chromosome microdissection, next-generation sequencing, personalized medicine
Autoři: T. Jančušková 1; R. Plachý 1; J. Štika 1; L. Krutílková 1; Hardekopf Dw 1; T. Liehr 2; N. Kosyakova 2; R. Čmejla 1; L. Žejšková 1; T. Kozák 3; P. Žák 4; M. Karas 5; S. Peková 1
Působiště autorů: Chambon s. r. o., Laboratoř molekulární diagnostiky, Praha, 2Friedrich-Schiller-Universität, Institut für Human Genetik, Jena, 3FN Královské Vinohrady, Interní hematologická klinika, Praha, 4FN Hradec Králové, 2. interní klinika – oddělení klinické hemato 1
Vyšlo v časopise: Transfuze Hematol. dnes,19, 2013, No. 1, p. 8-21.
Kategorie: Souhrnné práce, původní práce, kazuistiky
Souhrn
Akutní leukemie (AL) představují heterogenní skupinu maligních onemocnění krvetvorby s různou prognózou. Díky této různorodosti AL nelze s jistotou predikovat odpověď pacienta na léčbu. Proto je velmi důležité sledovat množství zbytkové maligní populace buněk po léčbě, tzv. minimální reziduální nemoc (MRN). K detekci MRN se běžně využívají rekurentní cytogenetické abnormality a mutace v hematologicky významných genech, které jsou rutinním screeningem identifikovány u naprosté většiny dospělých pacientů s akutní lymfoblastickou leukemií a přibližně u poloviny dospělých pacientů s akutní myeloidní leukemií. Z tohoto důvodu je velmi žádoucí identifikovat nové specifické markery leukemických blastů, které budou sloužit ke sledování MRN u pacientů, u nichž nebyla nalezena žádná ze standardně vyšetřovaných aberací.
Naším cílem bylo vyvinout zcela nový technický přístup k identifikaci a mapování unikátních klonálně specifických chromozomových abnormalit až na úroveň jednotlivých nukleotidů, a to pomocí moderních technik molekulární cytogenetiky, zejména mnohobarevné fluorescenční in situ hybridizace, mnohobarevného pruhování chromozomů (mFISH, mBAND) a multiplexní hybridizace fluorescenčně značených sond (BAC-FISH). Pro vyšší rozlišení byly fluorescenčně značené sondy aplikovány na linearizovaná vlákna DNA (molecular combing, fiber-FISH). Dalším nástrojem, který byl využit k přesné identifikaci zlomových míst aberovaných chromozomů byla mikrodisekce derivovaných chromozomů a následné sekvenování disekovaného materiálu pomocí technologie sekvenování nové generace (NGS). Posledním krokem byla konstrukce specifické molekulární PCR eseje v reálném čase pro monitorování MRN, která umožňuje sledovat odpověď pacienta na léčbu, příp. včas zachytit počínající molekulární relaps onemocnění.
Moderní technologie umožňují detekovat a identifikovat unikátní klonálně specifické abnormality u pacientů s AL. Předložená práce jasně ukazuje, že mapování chromozomových aberací až na úroveň nukleotidů je pro vybrané pacienty s AL realizovatelné a vhodné pro standardní klinickou praxi. Jedná se o laboratorní přístup „šitý na míru“ nemocných s AL, který naplňuje naši představu o personalizované medicíně.Klíčová slova:
akutní leukemie, minimální reziduální nemoc, molekulární cytogenetika, chromozomová mikrodisekce, sekvenování nové generace, personalizovaná medicínaÚvod
Akutní leukemie (AL) patří mezi závažná hematoonkologická onemocnění s různou prognózou, která závisí na včasné a správné diagnóze. Díky velké biologické diverzitě AL bohužel nelze zcela predikovat odpověď pacienta na léčbu. Proto je nutné využívat laboratorních technik, které pomocí klonálně specifických znaků umožní správně diagnostikovat a stratifikovat pacienty s AL.
Cytogenetická analýza buněk kostní dřeně, případně periferní krve, je nedílnou součástí vyšetření při záchytu onemocnění. Umožňuje nejen upřesnění diagnózy, určení stadia nemoci, ale je také důležitým nezávislým prognostickým ukazatelem. Abnormality karyotypu u de novo zjištěných akutních leukemií jsou sice prokazatelné přibližně u 50 % dospělých pacientů s AML, resp. u 70 % dospělých pacientů s ALL (1), ale jen některé z nich mají za následek vznik fúzního produktu, jenž je možné detekovat na molekulární úrovni, a tedy s vyšší citlivostí než je tomu u cytogenetického vyšetření, případně vyšetření pomocí průtokové cytometrie (2). K rutinně vyšetřovaným fúzním transkriptům, které lze monitorovat na RNA úrovni patří u pacientů s akutní myeloidní leukemií (AML) zejména AML1-ETO (RUNX1-RUNX1T1), PML-RARΑ, DEK-CAN (DEK-NUP214), CBFβ-MYH11 (3). U pacientů s akutní lymfoblastickou leukemií (ALL) jsou to fúzní transkripty BCR-ABL, MLL-AF4 (MLL-AFF1), E2A-PBX1 (TCF3-PBX1), TEL-AML1 (ETV6-RUNX1) (4, 5). U některých pacientů jsou pomocí metod molekulární biologie identifikovány nejrůznější mutace v hematologicky významných genech – NPM1, CEBPα, FLT3, c-KIT, WT1 (6, 7, 8). U pacientů s ALL jsou časté přestavby těžkých řetězců imunoglobulinu (IGH) a T-buněčného receptoru (TCR) (9).
Zmíněné chromozomové abnormality, genové mutace a přestavby mohou být v průběhu léčby pacientů s AL použity jako molekulární markery k monitorování minimální reziduální nemoci (MRN). Detekce MRN je v současné době rozhodujícím nástrojem ke zhodnocení kvality odpovědi pacienta na léčbu, jelikož umožňuje predikovat její selhání, případně u pacientů po transplantaci kostní dřeně dovoluje včas předpovědět blížící se relaps onemocnění. Molekulární technologie, založené na PCR v reálném čase (Real-Time PCR), využívající specifických genetických znaků maligních buněk, mají vysokou senzitivitu i specifitu, a jsou proto v moderní medicíně považovány za zlatý standard v diagnostice AL a sledování MRN. Proto je velmi žádoucí identifikovat nové klonálně specifické znaky leukemických buněk, a umožnit tak monitorování MRN i pacientům, u nichž nebyla nalezena žádná ze standardně vyšetřovaných aberací.
Cílem naší současné práce bylo vyvinout postup, který slouží nejen k identifikaci nových unikátních klonálně specifických markerů, ale také k jejich převedení z cytogenetické úrovně (chromozomové) na molekulární úroveň (nukleotidovou). Finálním krokem je příprava PCR eseje v reálném čase pro specifické sledování MRN, která umožní s vysokou specifitou a citlivostí sledovat dynamiku maligní populace buněk v odpovědi na léčbu pacienta.
Materiál a metodika
Pilotní studie
V rámci pilotní studie byly testovány dva přístupy identifikace nukleotidové sekvence zlomu derivovaných chromozomů. Pro oba přístupy byla nutná identifikace derivovaného chromozomu pomocí molekulárně cytogenetických metod. První způsob spočíval v mapování aberovaného chromozomu pomocí fluorescenčně značených BAC (umělý bakteriální chromozom) sond. Druhým přístupem byla mikrodisekce chromozomů s následnou sekvenací disekovaného materiálu. Pro tyto experimenty byla použita buněčná linie K562 (ATCC, USA) nesoucí derivovaný chromozom 10 a buňky kostní dřeně pacientů s AL.
Pacienti
V této práci byly analyzovány vzorky kostní dřeně dvou pacientů se stanovenou diagnózou AL. Kostní dřeň byla odebírána v záchytu onemocnění a dále v průběhu léčby pacienta. Vzorky byly odebírány na základě informovaného souhlasu pacienta.
Izolace DNA a RNA a reverzní transkripce
DNA i RNA byla izolována z mononukleární frakce kostní dřeně pacientů s AL v záchytu onemocnění a v jednotlivých bodech sledování MRN. DNA byla izolována pomocí automatického izolátoru MagNA Pure (Roche, Německo); RNA byla izolována pomocí TRI Reagent (Sigma, Německo), podle návodu uvedeného výrobcem. Reverzní transkripce byla provedena systémem VersoRT Blend (AB-gene, UK), podle návodu doporučovaného výrobcem.
PCR v reálném čase
Pro screening chromozomových aberací u pacientů s nově diagnostikovanou AL byl využit multiplexní Real-Time PCR systém AcutePlexX, vyvinutý naší laboratoří (10), který umožňuje detekovat více než 100 známých fúzních transkriptů a jejich sestřihových variant v 10 paralelních PCR reakcích (tab. 1). Vlastní analýza probíhá na platformě Rotor-Gene Q (Qiagen, Německo).
Tab. 1. AcutePlexX. Seznam rekurentních chromozomových abnormalit detekovaných u pacientů s AL. 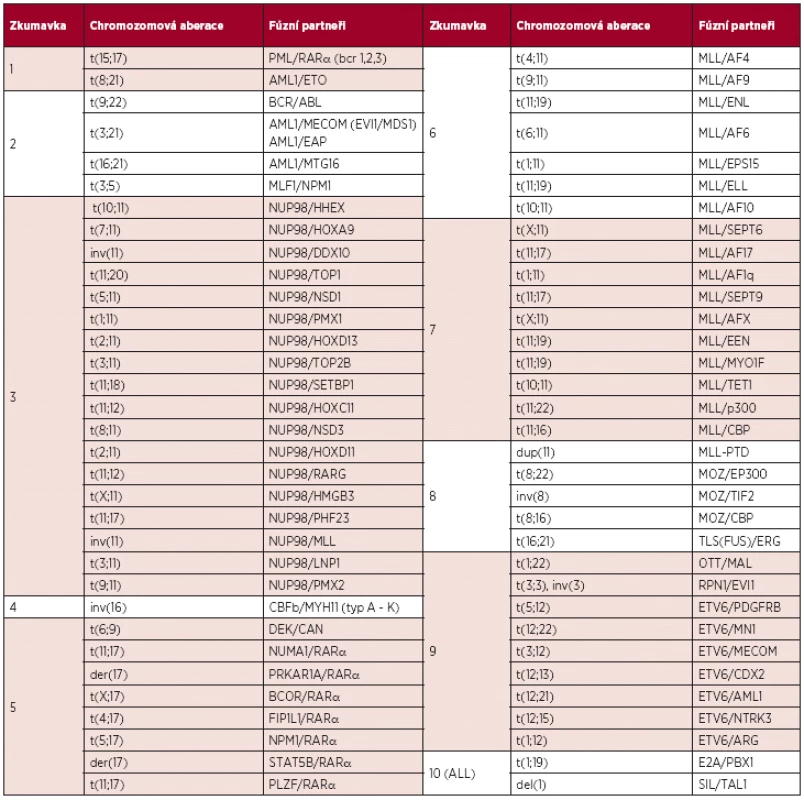
Kultivace a zpracování vzorků kostní dřeně
Vzorky kostní dřeně byly separovány na hustotním gradientu Histopaque (Sigma, Německo). 10M buněk odebraných z mononukleární frakce kostní dřeně bylo kultivováno v kultivačním médiu RPMI 1640 (PAA Laboratories, Rakousko) za přítomnosti 10% fetálního séra (PAA Laboratories, Rakousko), antibiotik (Penicilin/Streptomycin) a L-glutaminu (PAA Laboratories, Rakousko) při 37 °C/5% CO2 po dobu 24 hodin. Vzorek byl dále zpracován dle standardních cytogenetických postupů – přidání kolcemidu (0,1 μg/ml) (Gibco, UK), hypotonie a fixace směsí metanol/kyselina octová.
Buněčná linie K562 byla kultivována v RPMI 1640, s přídavkem 10% fetálního séra, antibiotik (Penicilin/Streptomycin) a glutaminu pro dosažení exponenciálního růstu, a poté zpracována analogicky jako vzorky kostní dřeně pacientů s AL.
Molekulární cytogenetika
Vyšetření karyotypu pacientů s AL, resp. buněčné linie K562, a upřesnění zlomů na derivovaných chromozomech bylo provedeno pomocí technik mnohobarevné fluorescenční in situ hybridizace – mFISH a mnohobarevného pruhování chromozomů – mBAND (MetaSystems, Německo). Veškeré hybridizační a post-hybridizační kroky se řídily dle doporučeného manuálu výrobce. Analýza probíhala s použitím fluorescenčního mikroskopu Axio Imager.Z2 (Zeiss, Německo) a počítačového softwaru Isis (verze 5.4, MetaSystems, Německo). U každého pacienta a buněčné linie bylo analyzováno 20 metafází.
Knihovna BAC
K experimentům byla využita genomická BAC knihovna (GenomicSet 737, ImaGenes, Německo), obsahující cca 25 000 viabilních bakteriálních klonů nesoucích unikátní dlouhé inzerty lidské DNA (~150 kb) klonované ve vektoru BAC (pBACe3.6, pBeloBAC11, pCYPAC2). Knihovna je vlastnictvím naší laboratoře a je po domluvě přístupná pro spolupracující laboratoře.
Izolace DNA z BAC klonů
DNA z klonů BAC byla izolována z 50 ml bakteriální kultury kultivované přes noc v LB (Luria-Bertani) médiu (Sigma, Německo) s přidáním 30 µg/ml chloramfenikolu, příp. kanamycinu (finální koncentrace – 1,5 mg/50 ml LB) pomocí kitu PhasePrepTM BAC DNA (Sigma, Německo) dle modifikovaného protokolu od výrobce, který byl doplněn o extrakci BAC DNA pomocí směsi fenol/chloroform.
Značení BAC DNA
K fluorescenčnímu značení DNA izolované z BAC klonu byla použita metoda „nick-translace“ za použití komerčně dostupného kitu – Nick Translation System (Invitrogen, UK) a komerčních fluorescenčně značených nukleotidů (Jena Bioscience, Německo). Pro značení BAC DNA byly použity nukleotidy s fluorofory TexasRed-dUTP, Fluorescein-dUTP, Atto425-dUTP, Cy5-dUTP a Cy3-dUTP. Fluorescenční značení probíhalo v 50 µl reakčním objemu dle standardního protokolu doporučovaného výrobcem. Koncentrace a čistota DNA byla stanovena spektrofotometricky. Do značící reakce byl vložen 1 µg BAC DNA.
Fluorescenčně značená DNA byla poté standardně precipitována přidáním 1/10 objemu 3M octanu sodného (Sigma, Německo) a 2,5x objemu 96% etanolu. K precipitátu byl přidán 1 µg Cot-1 DNA (BlueGnome, UK) k vyvázání repetitivních sekvencí. Po sražení byl DNA precipitát rozpuštěn v 5 µl Hi-Di formamidu (Applied Biosystems, USA) a uchováván při 4 °C. Pro následnou hybridizaci byl smíchán 1 µl DNA/Hi-Di roztoku s 9 µl hybridizačního pufru (50% formamid, 10% dextran sulfát, 10 mM Tris-HCl, 0,1% Tween20, 2xSSC).
Hybridizace BAC sond
Denaturace sondy a preparátu probíhala zvlášť. Preparát byl denaturován pomocí denaturačního pufru (70% formamid, 2xSSC, H2O) po dobu 2 minut při teplotě 75 °C na vyhřívané ploténce. Poté byl preparát odvodněn etanolovou řadou (70 %, 85 %, 96 %). Sonda byla denaturována 5 minut při teplotě 75 °C v termocycleru (Bio-Rad, USA). Hybridizace probíhala za standardních podmínek při 37 °C v hybridizační komůrce (Boekel Scientific, USA).
Izolace celogenomové DNA
K izolaci celogenomové DNA, pro účely „long-range“ PCR, byl použit automatický izolátor nukleových kyselin MagNA Pure Compact System (Roche, Německo). Vstupním materiálem byla buněčná suspenze.
„Long-range“ PCR (LR-PCR)
Primery pro LR-PCR reakci byly navrženy pomocí počítačového programu Vector NTI (Invitrogen, verze 11.5). K amplifikaci LR-PCR produktů byl použit kit Expand Long Range, dNTPack (Roche, Německo). Postup při PCR reakci se řídil doporučeními výrobce. Reakční směs (celkový objem 50 µl) obsahovala přibližně 200 ng DNA, 5U Expand Long Range Enzyme Mix, DMSO ve finální koncentraci 5 %, 1x Expand Long Range Buffer s MgCl2, směs nukleotidů ve finální koncentraci 500 µM a 0,4 µM každého z navržených primerů. Po úvodní inkubaci (8 min., 92 °C) následovalo 35 cyklů amplifikace (denaturace 30s/92 °C, hybridizace primerů 30s/58 °C a elongace 6 min./68 °C), pro kontrolu úspěšnosti PCR reakce bylo 10 µl PCR produktu separováno na 0,7% agarozovém gelu. Zbylý objem PCR reakce (40 µl) byl purifikován pomocí Gel Extraction Kitu (Qiagen, Německo) a eluován do 30 µl finálního objemu. LR-PCR amplikony byly fluorescenčně značeny „nick-translací“ analogickým postupem jako BAC DNA (viz Značení BAC DNA). LR-PCR sondy byly hybridizovány na metafázní chromozomy společně s kontrolní centromerickou sondou pro konkrétní chromozom (MetaSystems, Německo).
Izolace DNA pro „fiber-FISH“
DNA byla izolována z kultivovaných buněk, které byly promyty roztokem PBS a lyzovány lyzačním pufrem - Tris-EDTA, Sarkosyl (Sigma, Německo) a Proteinasa K (Roche, Německo), lyzát byl ošetřen RNázou (Sigma, Německo) a extrahován směsí fenol/chloroform. DNA byla precipitována pomocí roztoku 3M octanu sodného (NaAc) (Sigma, Německo) a 96% etanolem. Precipitát byl opláchnut 80% etanolem a rozpuštěn v 0,5xTE pufru.
„DNA combing“ a „fiber-FISH“
DNA „česání“ („DNA combing“) s hybridizací fluorescenčně značených sond a následnou imunodetekcí signálů pomocí monoklonálních protilátek bylo provedeno dle protokolu francouzské laboratoře (Institute of Human Genetics Montpellier, France; osobně z laboratoře, prof. Pasero).
Vlákna DNA byla „česána“ na silanizované krycí sklo (Genomic Vision, Francie) pomocí přístroje, který umožňuje tažení skel konstantní rychlostí 300 µm/s (Dip Coater). Použitý přístroj byl ve spolupráci s naší laboratoří navrhnut a vyroben Elektrotechnickou fakultou Západočeské Univerzity v Plzni. Linearizovaná vlákna DNA byla vizualizována cyaninovým barvivem YOYO-1 (Invitrogen, UK).
Imunodetekce
LR-PCR produkty značené fluorofory TexasRed-dUTP a Fluorescein-dUTP byly denaturovány 5 minut při 75 °C. „Načesaná“ skla byla dehydratována ve vzestupné etanolové řadě (70 %, 85 %, 96 %) a denaturovaná v čerstvě připraveném 1M roztoku hydroxidu sodného s následnou neutralizací pufrem PBS. Hybridizace probíhala při 37 °C přes noc. Nespecificky navázaná sonda byla odstraněna odmytím při 44 °C v 50% formamidu a 2 x SSC. Preparáty byly následně vloženy do roztoků 2 x SSC, PBS a ošetřeny 1% roztokem BSA (Albumin z bovinního séra, Sigma). Pro vizualizaci vláken a sond jsme využili možnosti kombinace různě značených protilátek (IgG), které nám umožnily detekci tří barev a amplifikaci signálů. Proti jednovláknové DNA (ssDNA) jsme použili myší protilátky proti ssDNA (Millipore, USA), které sloužily jako antigen pro protilátky králičí značené Alexa Fluor 350 (Invitrogen, UK). Signály hybridizovaných sond byly zesíleny dvěma vrstvami protilátek, proti TexasRed (králičí)/Fluorescein (kozí), a vrstvou fluorescenčně značených protilátek proti králičímu IgG (oslí, Alexa Fluor 594)/kozímu IgG (kuřecí, Alexa Fluor 488) (Invitrogen, UK). Mezi jednotlivými vrstvami byly preparáty promyty v PBS/T (0,1% Triton X 100 solution, Sigma, Německo). Pořízení obrázků a analýza dat byla provedena pomocí fluorescenčního mikroskopu Axio Imager.Z2 (Zeiss, Německo).
Chromozomová mikrodisekce
Mikrodisekce derivovaných chromozomů probíhala ve spolupráci s Institut für Human Genetik, Friedrich-Schiller-Universität Jena, Německo. Buněčné suspenze byly kapány na krycí skla (24 x 60 mm) ošetřená 10% roztokem SDS. Čerstvě nakapaná skla byla inkubována ve fosfátovém pufru (pH 6,88) po dobu 1 min, a poté trypsinizována (40–60 s). Skla byla barvena roztokem Giemsa po dobu 2–3 min a opláchnuta destilovanou vodou.
Vlastní mikrodisekce byla provedena pomocí skleněné jehly (průměr ~500 nm) pod invertovaným mikroskopem (Axiovert-135, Axiovert-10 Zeiss, Německo) vybaveným elektronickým mikromanipulátorem. Po mikrodisekci byly fragmenty chromozomů ošetřeny roztokem proteinázy K (11).
PCR s degenerovanými primery
K amplifikaci disekovaných produktů byla použita metoda PCR s degenerovanými primery (DOP-PCR) s použitím komerčně dostupného WGA - Whole Genome Amplification – kitu (Sigma, Německo), dle návodu doporučeného výrobcem. Specifita disekovaných chromozomových fragmentů byla ověřena reverzní fluorescenční in situ hybridizací na metafázní chromozomy (12).
Sekvenování nové generace (Next Generation Sequencing; NGS)
K sekvenaci byl využit GS Junior pyrosekvenační systém (Roche, Německo). V průběhu celého procesu přípravy DNA knihovny, klonální amplifikace i vlastního sekvenování bylo postupováno dle návodu výrobce, pouze s drobnou změnou – před opravou konců fragmentů bylo 500 ng produktů DOP-PCR nejprve přečištěno pomocí Gel Extraction Kitu (Qiagen, Německo) a následně pomocí MinElute PCR Purification Kitu (Qiagen, Německo). Pro klonální amplifikaci fragmentů sloužil GS Junior Titanium emPCR Lib-L Kit (Roche, Německo).
GeneAnalyzer software
Pro identifikaci zlomových míst na derivovaných chromozomech byl naší laboratoří vyvinut software GeneAnalyzer, který umožňuje jednotlivá čtení (reads) mapovat na referenční sekvence chromozomů (NCBI sestava 37.3). Jako hlavní algoritmus pro určení homologie software využívá BLAST+ verze 2.2.26. Hlavním úkolem řídicí aplikace je shromažďovat získané informace o homologii a pozici jednotlivých mapovaných čtení na referenční sekvenci. Software umožňuje nastavit celou řadu parametrů důležitých pro srovnání čtení s referencí (např. procento homologie, minimální délku pro pozitivní přiřazení, minimální diferenci v homologii) tak, aby využitelnost získaných čtení z přístroje GS Junior byla maximální, ale při tom, aby nedocházelo k falešně pozitivním přiřazením.
Výsledky
K vyřešení uvedených cílů byla klíčová identifikace derivovaných chromozomů v karyotypu pacientů a buněčné linie K562 pomocí technik molekulární cytogenetiky – mFISH a mBAND. K přesné identifikaci chromozomových zlomů byly navrhnuty dva různé přístupy. Prvním přístupem bylo mapování zlomů na derivovaném chromozomu pomocí hybridizace fluorescenčně značených BAC klonů. Pro tento experiment jsme použili chromozomové preparáty vzorku kostní dřeně pacienta s AML (pacient 1). Druhý přístup spočíval v mikrodisekci části derivovaného chromozomu a následné sekvenaci disekovaného materiálu. Pro mikrodisekci byla zvolena buněčná linie K562 a buňky kostní dřeně pacienta s ALL (pacient 2). Hlavní kroky obou strategií jsou znázorněny na obrázku 1.
Obr. 1. Znázornění dvou způsobů identifikace nového markeru pro sledování MRN. A) Mapování derivovaného chromozomu pomocí fluorescenčně značených BAC klonů a LR-PCR produktů. B) Mikrodisekce derivovaného chromozomu následovaná amplifikací a sekvenací disekovaného materiálu. 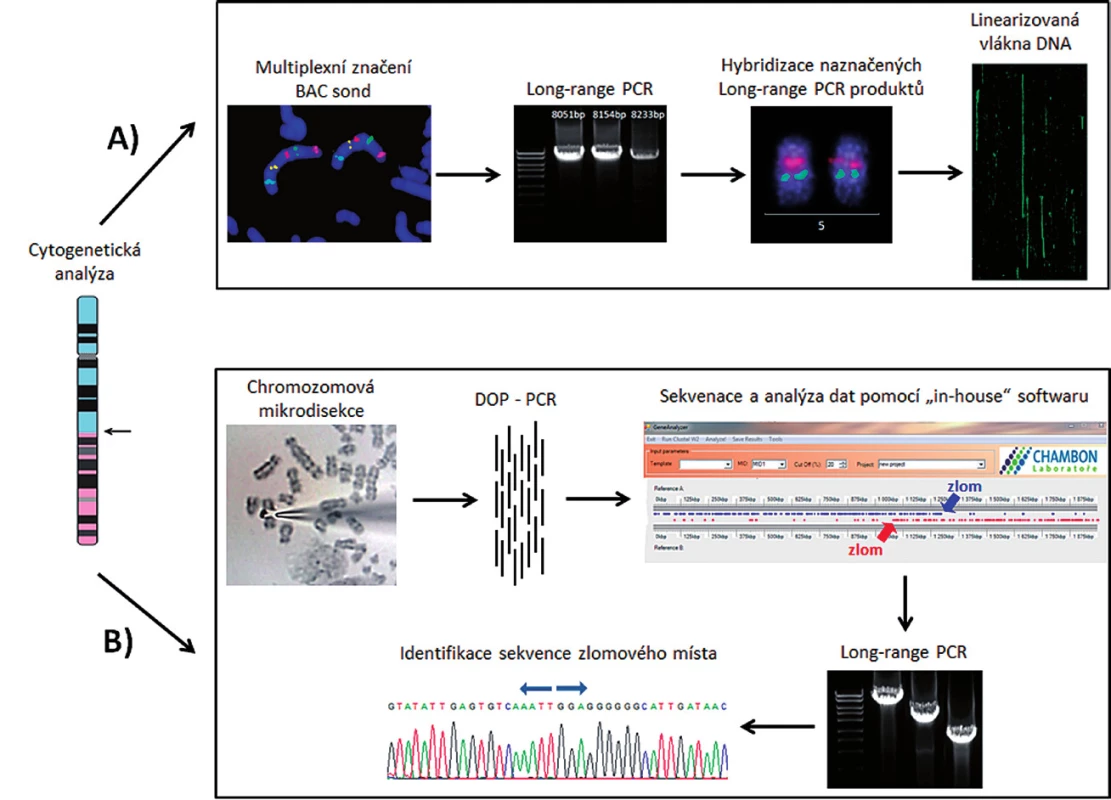
Molekulárně cytogenetickým vyšetřením (mFISH a mBAND) jsme u pacienta 1 nalezli karyotyp s komplexními změnami a pro účely detekce MRN jsme dále analyzovali derivovaný chromozom 5 (obr. 2 A, B). Strategií pozičního klonování jsme dále mapovali zlomové oblasti na der(5) pomocí fluorescenčně značených BAC sond. Při poslední hybridizaci jsme identifikovali BAC klon RP11-461G12 ležící pravděpodobně v místě zlomu chromozomu 5. Tento BAC měl nápadně nižší intenzitu signálu ve srovnání se zdravým chromozomem 5 (obr. 3). Pro upřesnění polohy zlomu jsme sekvenci BAC klonu RP11-461G12 použili k návrhu primerů pro LR-PCR reakci. Amplifikovaný materiál byl dále použit na přípravu vlastních fluorescenčně značených LR-PCR sond. Průměrná délka produktů, které jsme pomocí LR-PCR amplifikovali, byla ~8 kb (6285 bp – 9088 bp). Protože citlivost metody FISH na metafázních chromozomech neumožňuje detekovat sondy o velikosti amplifikovaných produktů (tj. ~8 kb), byly vždy 3 až 4 LR-PCR amplikony spojeny, a sonda tudíž pokrývala oblast ~25 kb. Signály LR-PCR sond na metafázních chromozomech byly obtížně reprodukovatelné, proto jsme k hybridizaci LR-PCR sond použili linearizovaná vlákna DNA (obr. 4).
Obr. 2. Molekulárně cytogenetická analýza karyotypu pacienta 1. A) Mnohobarevná fluorescenční in situ hybridizace (mFISH). B) Mnohobarevné pruhování s vysokou rozlišovací schopností (mBAND) pro chromozomy 5 a 6. 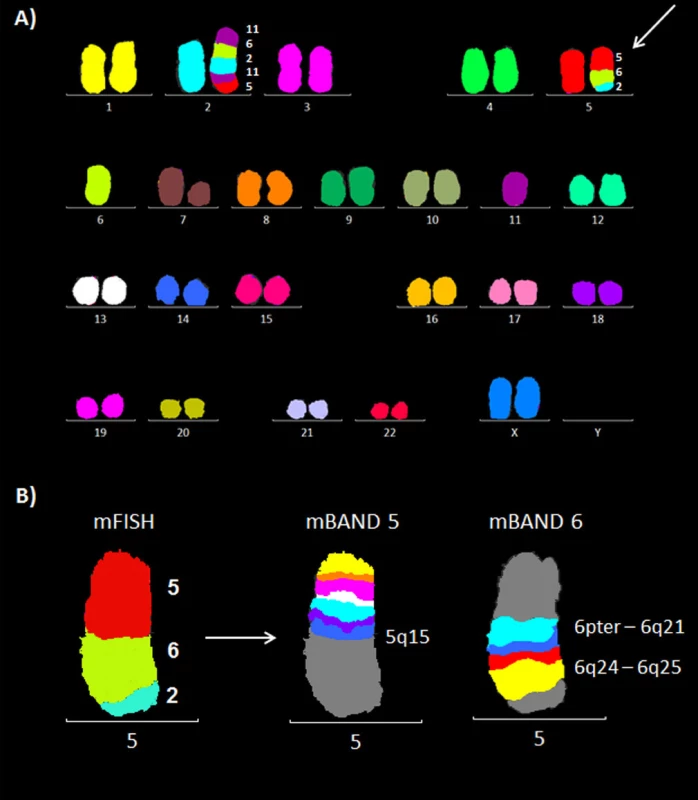
Obr. 3. Analýza pomocí fluorescenčně značených BAC klonů. Hybridizace BAC klonu RP11-461G12 pro chromozom 5q15. Šipka označuje signál o nápadně slabší intenzitě. 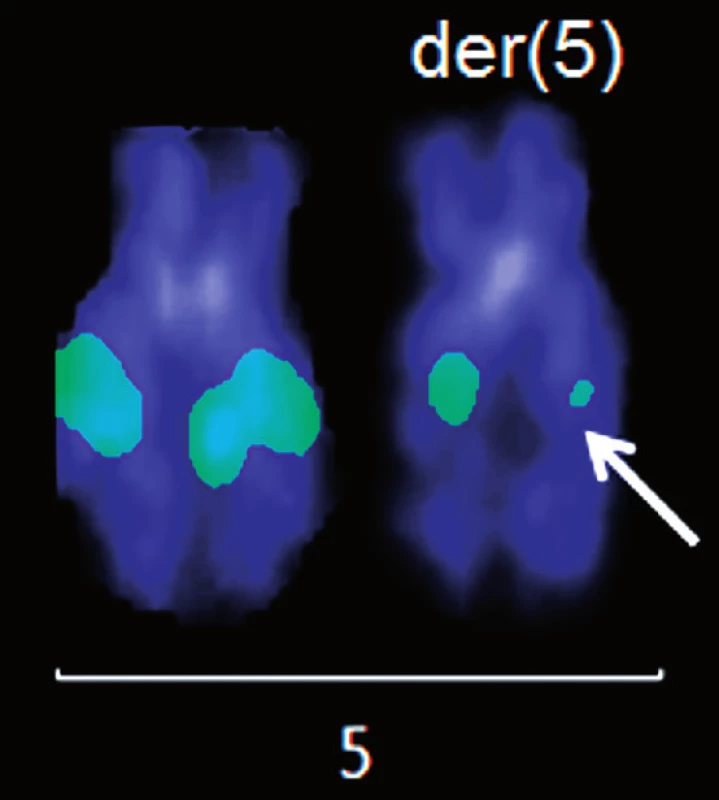
Obr. 4. Schéma trojbarevné detekce pomocí značených protilátek. 
Metodami mFISH a mBAND jsme u buněčné linie K562 identifikovali hypotriploidní karyotyp s přítomností derivovaného chromozomu - der(10)t(3;10)(p21.3;q23) (obr. 5). Část derivovaného chromozomu 10 obsahující materiál z chromozomů 3 a 10 byla disekována pomocí skleněné jehly. Pro zisk dostatečného množství DNA byly disekované fragmenty amplifikovány, a poté sekvenovány (obr. 1 B). Celkem bylo pro der(10) získáno 80 789 čtení, která byla přiřazena k referenčním sekvencím z chromozomů 3 a 10 a analyzována pomocí softwaru GeneAnalyzer, vyvinutého laboratoří Chambon (Plachý R., nepublikováno) (tab. 2). Z takto zmapované sekvence byly vždy po 1000 bp navrženy sady primerů (obr. 6) pro LR-PCR, která vedla k zisku produktu (~ 4,5 kb) (obr. 7 A). Tento amplikon byl poté sekvenován z obou stran standardní Sangerovou metodou. Sekvenační analýza potvrdila přítomnost sekvencí z chromozomů 3 a 10 a identifikovala oba fúzní partnery, gen CDC25A na chromozomu 3 (místo zlomu v intronu 2) a GRID1 na chromozomu 10 (místo zlomu v intronu 4) (obr. 7 B).
Obr. 5. Molekulárně cytogenetická analýza buněčné linie K562. A) Mnohobarevná fluorescenční in situ hybridizace (mFISH). B) Mnohobarevné pruhování s vysokou rozlišovací schopností (mBAND) pro chromozomy 10 a 3. 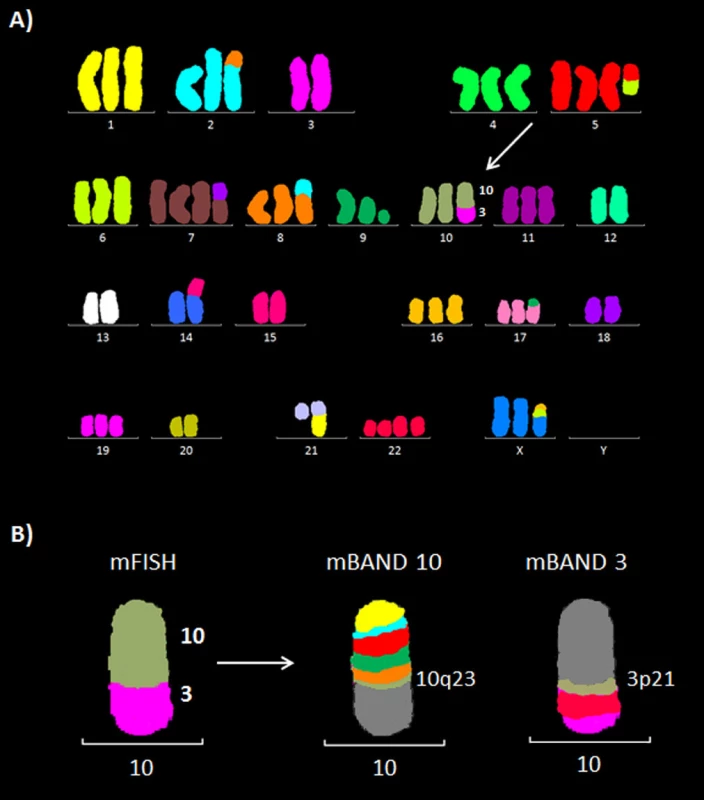
Tab. 2. Základní informace o analýze jednotlivých zlomových míst. 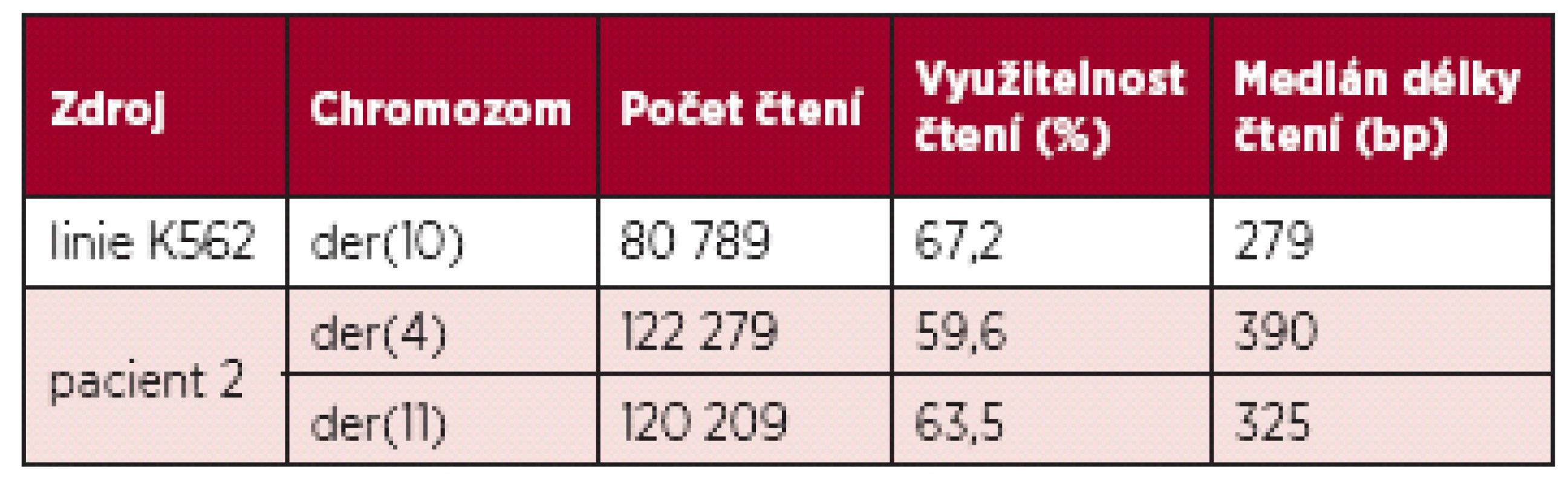
Obr. 6. Schématické zobrazení postupu při identifikaci zlomového místa na derivovaném chromozomu. 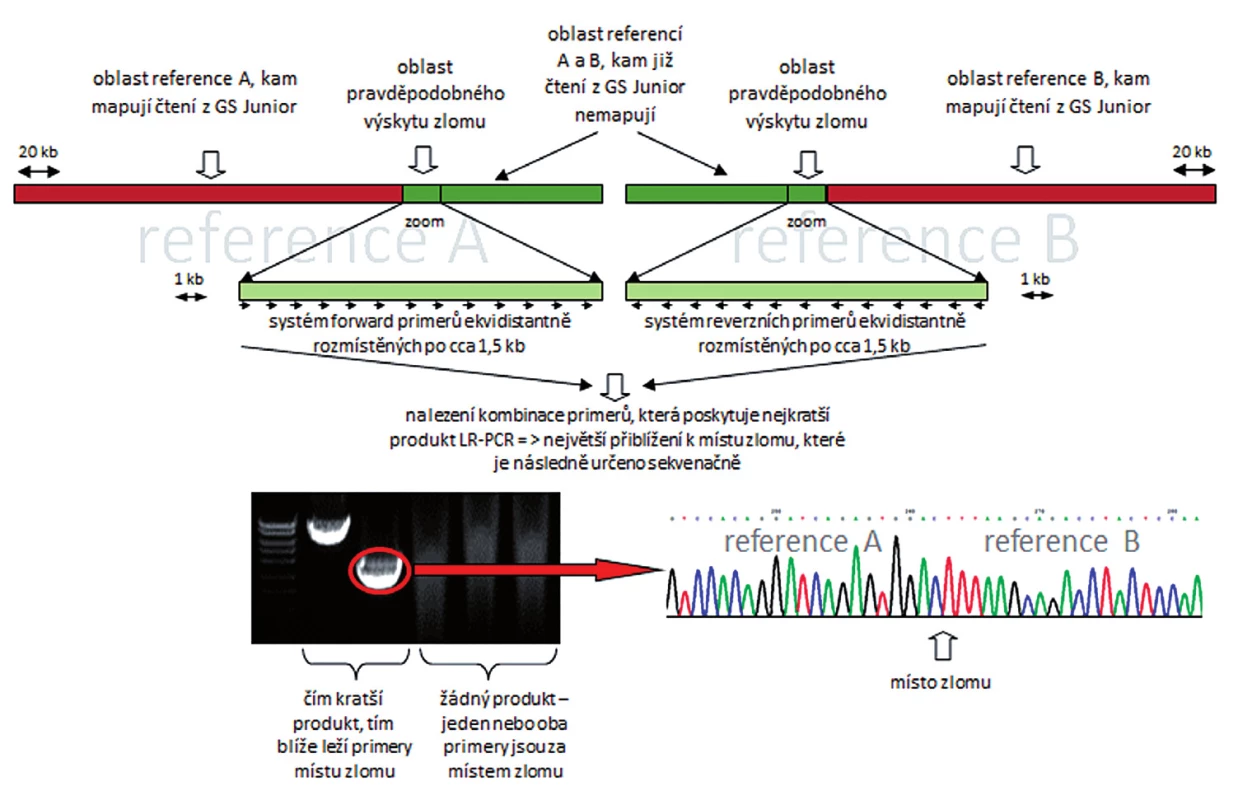
Obr. 7. Buněčná linie K562. A) LR-PCR produkt. B) Nukleotidová sekvence zlomu derivovaného chromozomu 10. 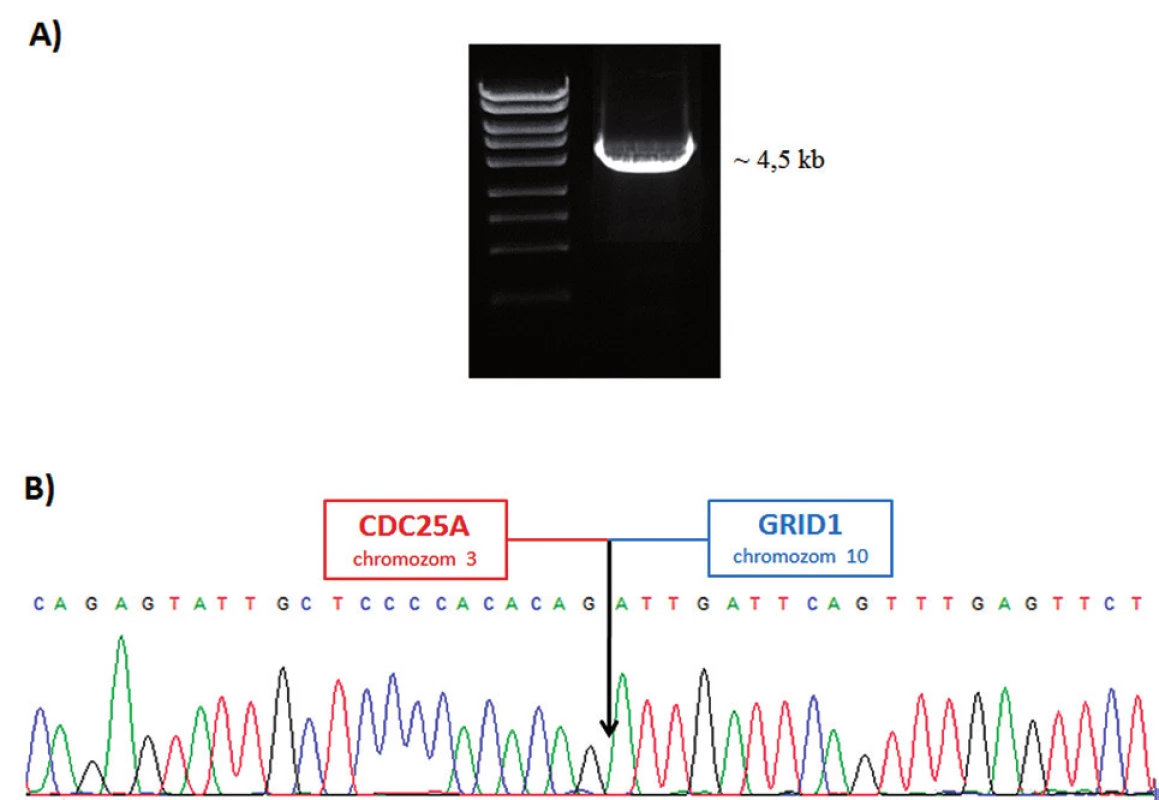
U pacienta 2 byla nalezena balancovaná chromozomová translokace zahrnující gen MLL – 46,XX,t(X;4;11)(q25;q21;q23) (obr. 8). Fúze genů MLL-AF4 byla potvrzena i pomocí Real-Time PCR (AcutePlexX) a sekvenační analýzou (neuvedeno). Stejný postup jako u buněčné linie byl aplikován i na derivované chromozomy 4 a 11 pacienta 2. Z každého derivovaného chromozomu bylo disekováno 10 fragmentů. Jejich specifita byla ověřena hybridizací na metafázní chromozomy negativní kontroly (obr. 9 A, B). Sekvenací disekovaných chromozomových fragmentů bylo získáno 122 279, resp. 120 209 čtení pro chromozomy der(4), resp. der(11) (tab. 2). LR-PCR amplifikace vedla k zisku produktů o velikostech ~ 0,5 kb pro derivovaný chromozom 4, resp. ~ 3 kb pro derivovaný chromozom 11 (obr. 10). Sekvenováním těchto produktů jsme identifikovali DNA sekvence chromozomových zlomů a fúzní partnery. Derivovaný chromozom 4 vznikl fúzí pseudogenu LOC392539 z chromozomu X a genu AF4 na 4q21. Derivovaný chromozom 11 vznikl fúzí genů MLL (intron 9) a AF4 (intron 5) (obr. 11). Návrh MRN eseje pro kvantitativní PCR v reálném čase poté umožnil kvantifikaci nově identifikovaných cílů (zlomy derivovaných chromozomů 4 a 11 – zlomová místa Xq25; 4q21, resp. 4q21; 11q23) a také srovnání s již zavedeným cílem pro sledování MRN (fúzní transkript MLL-AF4) (obr. 12).
Obr. 8. Molekulárně cytogenetická analýza karyotypu pacienta 2. A) Mnohobarevná fluorescenční in situ hybridizace (mFISH). B) Mnohobarevné pruhování s vysokou rozlišovací schopností (mBAND) pro chromozomy X, 4 a 11. 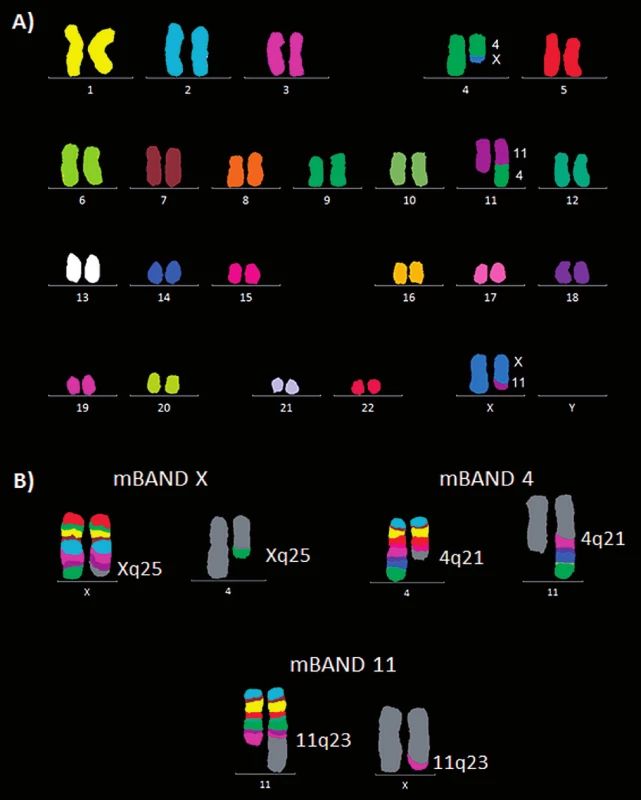
Obr. 9. Reverzní FISH. Ověření specifity disekovaných chromozomových fragmentů u pacienta 2. A) Derivovaný chromozom 4. B) Derivovaný chromozom 11. 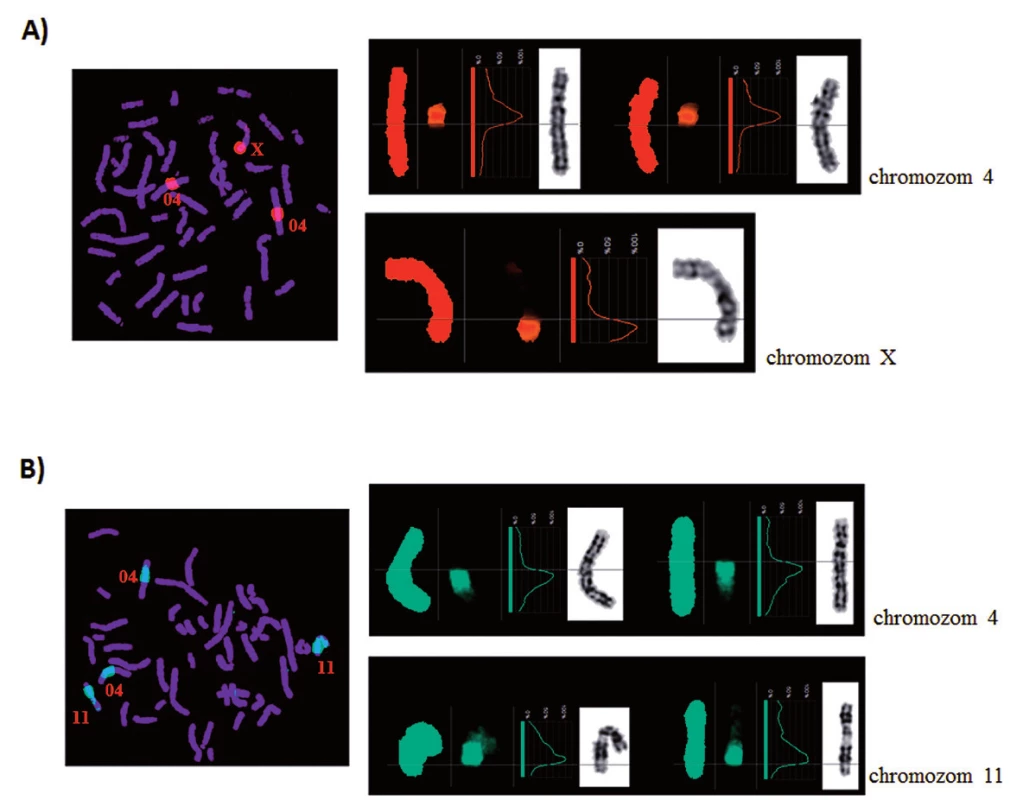
Obr. 10. LR-PCR produkty u pacienta 2. A) Derivovaný chromozom 4. B) Derivovaný chromozom 11. 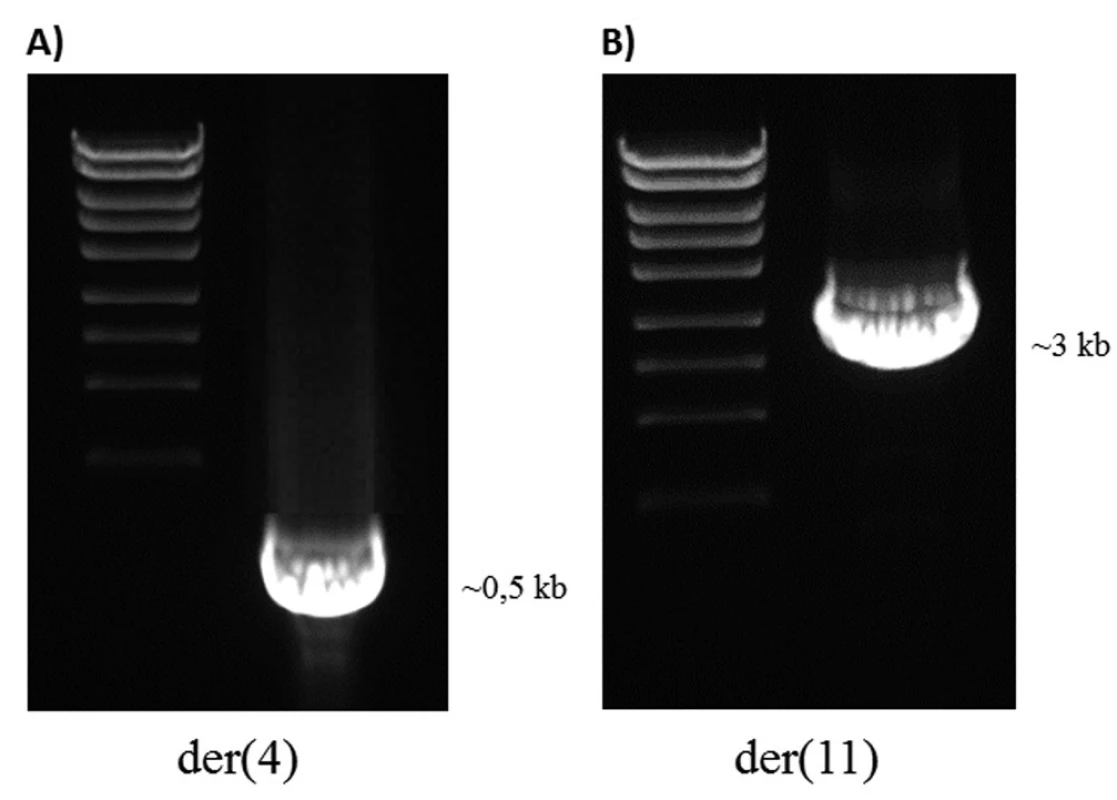
Obr. 11. Sekvence LR-PCR produktů A) derivovaný chromozom 4; fúze genu AF4 (intron 5) a pseudogenu LOC392539 na chromozomu X. B) derivovaný chromozom 11; fúze genů MLL (intron 9) a AF4 (intron 5). 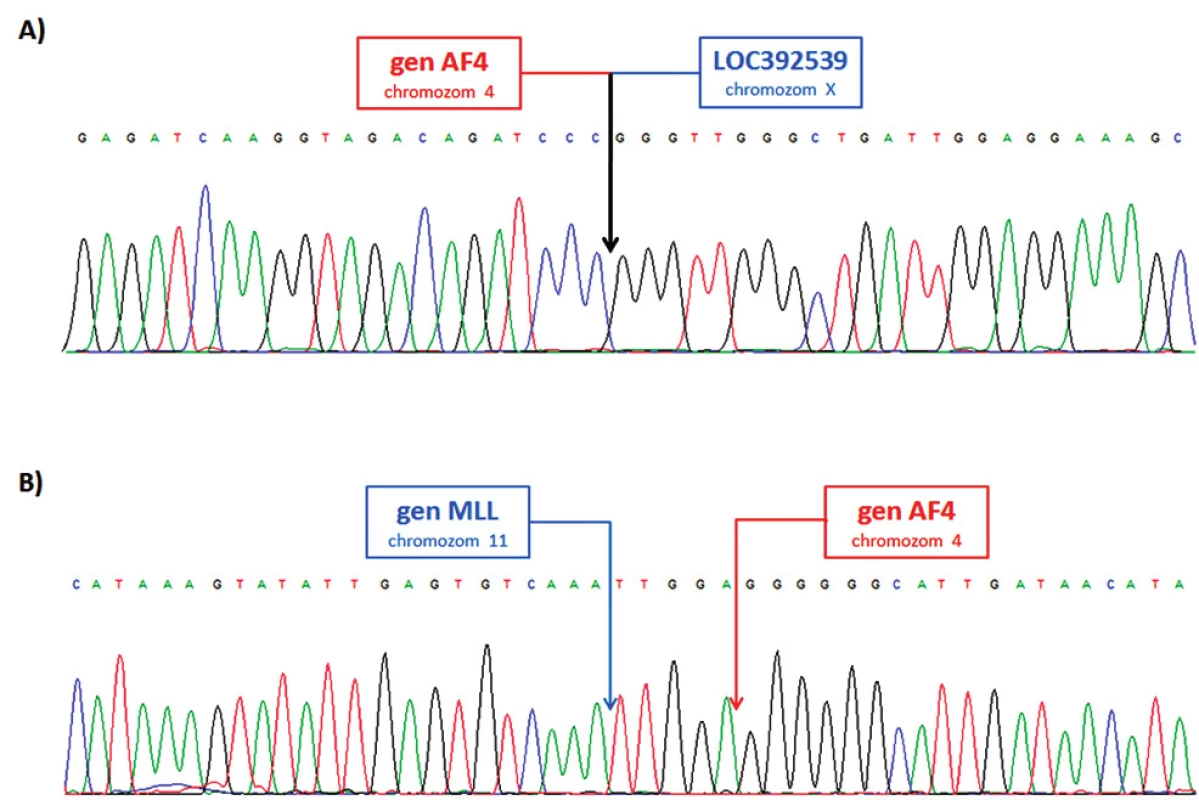
Obr. 12. Kvantifikační grafy. A) Relativní kvantifikace chromozomového zlomu na der(4). B) Relativní kvantifikace chromozomového zlomu na der(11). C) Relativní kvantifikace fúzního transkriptu MLL-AF4. BM – kostní dřeň. 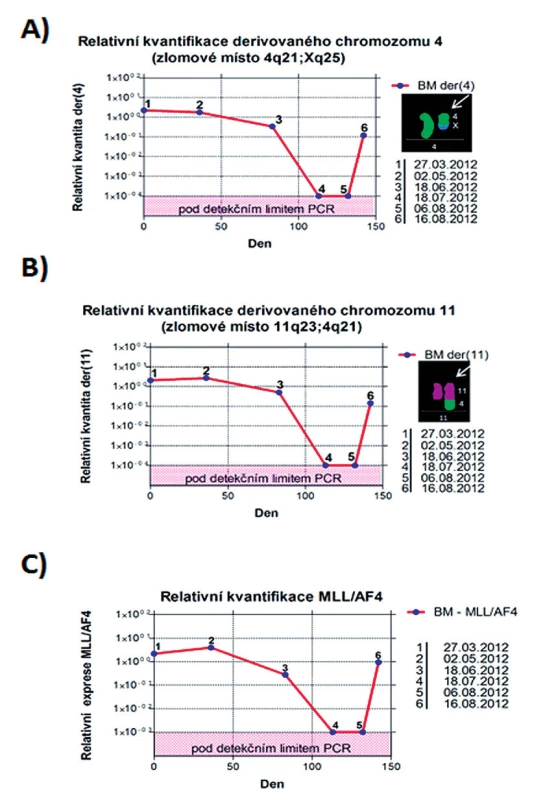
Příklad mapování zlomového místa (derivovaný chromozom 4, pacient 2) pomocí aplikace GeneAnalyzer je uveden na obrázku 13.
Obr. 13. Zlom na derivovaném chromozomu 4 určený pomocí softwaru GeneAnalyzer, vyvinutého laboratoří Chambon. 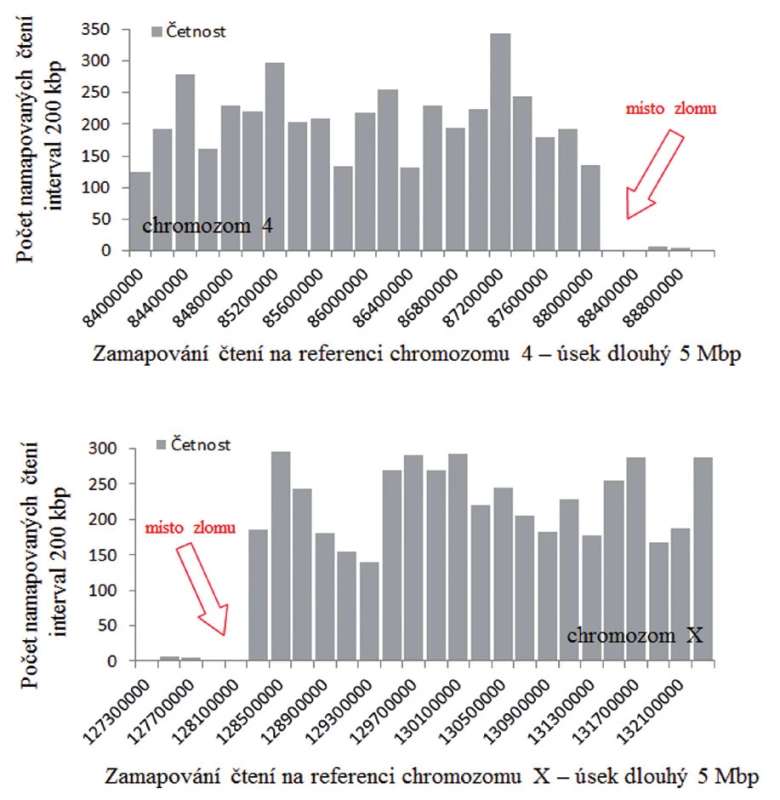
Diskuse
Specifické testy k detekci MRN umožňují precizně monitorovat osud maligní populace buněk v těle pacienta v průběhu léčby a vyhodnotit léčebnou odpověď. Proto má sledování MRN v průběhu léčby pacientů s AL nesporný klinický význam. Cílem naší práce bylo pomocí moderních technik molekulární cytogenetiky, celogenomového sekvenování a standardního Sangerova sekvenování připravit flexibilní metodiku pro identifikaci nových klonálně specifických znaků leukemických buněk u pacientů s AL, u kterých nelze pomocí běžného screeningového panelu analýz v záchytu onemocnění nalézt molekulární marker pro sledování MRN. Námi připravená metodologie pro detekci a identifikaci unikátních klonálně specifických aberací následně umožňuje připravit testy pro kvantitativní PCR v reálném čase ke sledování MRN. Tato technika může pro klinického lékaře představovat cenný nástroj pro sledování dynamiky vývoje maligní populace buněk nesoucích konkrétní unikátní klonální znak, a tudíž poskytnout možnost zhodnocení léčebné odpovědi pacienta tam, kde jiné techniky detekce MRN selhávají. V kombinaci s dalšími vyšetřovacími metodami je molekulární informace důležitým kamínkem v mozaice vyšetřovacích možností s potenciální návazností na léčbu.
Není bohužel možné tyto unikátní MRN testy mezilaboratorně standardizovat, jelikož se jedná o unikátní abnormality. Pro testování funkčnosti unikátních MRN testů (specificita, dynamický rozsah detekce) jsou aplikována identická pravidla jako pro testování funkčnosti MRN testů založených na rekurentních genetických abnormalitách. Proto lze s vysokou mírou pravděpodobnosti považovat výsledky i těchto unikátních molekulárních analýz za spolehlivé.
Imunofenotypizační analýza exprese CD antigenů na leukemických blastech je nedílnou součástí diagnostického procesu u pacientů s AL. Vyšetření pomocí průtokové cytometrie je důležité nejen při stanovení diagnózy, ale také k monitorování účinnosti léčby a stanovení prognózy onemocnění. Nicméně, pro účely detekce MRN u pacientů s AL může být využití průtokové cytometrie limitováno nestabilitou imunologických profilů leukemických buněk v průběhu léčby. Senzitivita průtokové cytometrie je 10-3 až 10-4, v závislosti na typu leukemie, použitém přístrojovém vybavení a detekčním panelu protilátek (13, 14, 15). Stanovení MRN pomocí kvantitativní PCR v reálném čase dosahuje obvykle senzitivity 10-4–10-5 (8, 15). Vyšetření pomocí cytogenetických metod je diagnosticky i prognosticky velmi užitečné, ale není dostatečně senzitivní k detekci zbytkových leukemických buněk u pacientů s AL. Základní cytogenetické vyšetření je ovlivněno ziskem adekvátního počtu analyzovatelných metafází. Molekulárně cytogenetické vyšetření pomocí FISH je sice citlivější, nezávislé na proliferačním potenciálu buněk a umožňuje analyzovat větší počet buněk než je tomu u klasické cytogenetiky, nicméně, pro účely stanovení MRN je nesrovnatelně vhodnější a citlivější metodika PCR. Prognostický význam detekce MRN pomocí PCR byl potvrzen mnoha pracemi (15, 16, 17, 18, 19). V naší laboratoři slouží jako cíl pro monitorování MRN pacientů s akutními leukemiemi klonální přestavby těžkých řetězců imunoglobulinu (IGH) a T-buněčného receptoru (TCR), fúzní transkripty a klonálně specifické mutace prognostických markerů asociovaných s leukemiemi identifikované pomocí konvenční PCR s následným sekvenováním. Fúzní transkripty jsou zahrnuty v unikátním diagnostickém systému AcutePlexX, díky kterému jsme schopni detekovat více než 100 známých fúzních transkriptů a jejich sestřihových variant (tab. 1). Uvedenými přístupy identifikujeme cíl pro sledování MRN přibližně u 50 % dospělých pacientů s AML. Popsaný postup identifikace nových klonálně specifických markerů maligních buněk umožňuje využívat i dosud nepopsané chromozomové aberace jako specifický cíl pro detekci a kvantifikaci MRN na molekulární úrovni.
K dosažení uvedeného cíle byly navrženy dva postupy vycházející ze stanovení karyotypu a identifikace derivovaného chromozomu. Prvním přístupem je mapování zlomových oblastí derivovaného chromozomu pomocí mFISH, mBAND a hybridizace fluorescenčně značených BAC sond. Druhým přístupem je mikrodisekce derivovaného chromozomu spojená s celogenomovým sekvenováním.
Mapování chromozomových zlomů lze považovat za konvenční metodu identifikace genomových abnormalit nejen u leukemických pacientů, ale také u pacientů se solidními tumory a jedinců s balancovanými translokacemi (20, 21, 22). Základním předpokladem je sestavení karyotypu a přesná identifikace derivovaného chromozomu. Klasická karyotypová analýza je limitována rozlišením a morfologií chromozomů. Z uvedených důvodů využíváme k identifikaci karyotypu pacientů kromě klasických „pruhovacích“ technik navíc i technik molekulární cytogenetiky – mFISH, resp. mBAND, které umožňují přesně a velmi rychle charakterizovat materiál aberovaných chromozomů. Uvedené metody dosahují rozlišení 2-3 Mb, resp. 1-1,5 Mb, zatímco klasická cytogenetická analýza poskytuje rozlišení 7-10 Mb (23, 24). Nicméně, i po zavedení metod molekulární cytogenetiky do rutinní praxe není úroveň rozlišení dostačující k precizní identifikaci zlomových oblastí derivovaných chromozomů. Proto je velmi cenným nástrojem dostupnost genomických knihoven, např. knihovna BAC klonů, kterou máme k dispozici v naší laboratoři. Knihovna čítá ~ 25 000 klonů a pokrývá celý lidský genom. Máme tedy možnost přípravy kvalitních BAC sond takřka v neomezeném množství. Díky DNA knihovně se můžeme vyhnout limitujícím podmínkám komerčně dodávaných sond, a to nejen časovým – dodání vybraných sond do 14 dnů, ostatních až za jeden měsíc; ale také kvalitativním – komerčně dostupné jen dva fluorofory, kdy v naší laboratoři dokážeme značit až pěti různými fluorofory. Díky multiplexnímu značení BAC klonů je možné v jedné hybridizaci použít až pět různých sond, tím se mapování zlomů výrazně urychluje. Pro ověření specifity BAC sond rehybridizujeme chromozomový preparát, na kterém byla aplikována metoda mFISH, příp. mBAND, tudíž není nutné používat kontrolní sondu. Navíc díky společné hybridizaci pěti BAC sond je velmi nepravděpodobné, že by všechny sondy nespecificky hybridizovaly ke stejnému chromozomu. U pacienta 1 byl BAC klon ležící v místě zlomu rozpoznán díky slabšímu signálu na derivovaném chromozomu ve srovnání se zdravým chromozomem (obr. 3), což bylo dál potvrzeno mapováním pomocí techniky NGS (neuvedeno). V případě mapování reciproké translokace jsme pozorovali „rozštěpený“ signál na obou translokovaných chromozomech (neuvedeno). Protože délka sekvence tohoto BAC (RP11-461G12) neumožňovala konstrukci PCR testu, bylo nutné použít metodik pro ještě vyšší rozlišení. Toho jsme dosáhli pomocí LR-PCR. Získané amplikony jsme fluorescenčně označili a použili jako sondy. Signály na metafázních chromozomech byly jasné a specifické, ale často byly přítomny jen na jednom z homologických chromozomů. To mohlo být způsobeno odlišným stupněm kondenzace obou homologů a také epigenetickými faktory (25, 26, 27). Pro lepší reprodukci signálů sond je proto vhodné aplikovat metodu s vyšším rozlišením, např. metodu „fiber-FISH“, která spočívá v uniformním a rovnoběžném natažení vláken DNA pomocí ustupujícího menisku při konstantní rychlosti 300 µm/s, kde přibližně 1 µm odpovídá oblasti 2 kb. K takto připraveným vláknům DNA hybridizujeme fluorescenčně značené sondy (28, 29, 30). Cílová oblast, kterou pokrývají LR-PCR produkty, příp. BAC sondy, je však velmi malá vzhledem k templátové DNA na mikroskopickém skle. V takto připraveném preparátu je proto velmi obtížné hybridizované cíle najít (31).
V naší studii jsme použili přístup separace derivovaného chromozomu pomocí mikrodisekce. Tato metodika se běžně využívá k přípravě chromozomově specifických knihoven DNA, malovacích sond, a také k identifikaci marker chromozomů (32, 33). Předpokladem pro úspěšnou disekci je dostatek dobře rozptýlených chromozomů a jejich rozlišení na základě klasických barvicích technik (barvení Giemsou, G-pruhování) (32), příp. použitím fluorescenčních sond (osobní sdělení, prof. Thomas Liehr, Německo). Laserová mikrodisekce sice umožňuje rychlou a precizní disekci cílového chromozomu bez kontaminace nebo poškození genetického materiálu, nicméně pro účely izolace intersticiálních částí chromozomu nemusí být příliš vhodná (osobní sdělení, Dr. Anya Hunt, MMI, Švýcarsko). Proto jsme v naší práci zvolili konvenční mikrodisekci s použitím skleněné mikrokapiláry ovládané elektronicky řízeným mikromanipulátorem. Ve srovnání s laserovou mikrodisekcí je konvenční mikrodisekce finančně dostupnější alternativou pro rutinní využití v laboratorní praxi (34). K izolaci derivovaných chromozomů může být použita i průtoková cytometrie, ale tento přístup je závislý na velikosti chromozomů, a proto nemusí být vždy s úspěchem využit, navíc neumožňuje studium invertovaných chromozomů (20). Podobně jako při použití laserového mikrodisektoru, průtokovou cytometrií není možné separovat specifické intersticiální fragmenty derivovaných chromozomů. Pořízení průtokového cytometru umožňujícího separaci jednotlivých chromozomů je velmi nákladnou investicí, což také limituje jeho použití ve standardní laboratoři.
Disekované fragmenty chromozomů byly dále amplifikovány a sekvenovány na platformě GS Junior (Roche). Chen et al. ve studii popisují použití celogenomového sekvenátoru Illumina 1G (Solexa), který v průběhu jedné analýzy dokáže získat až miliony čtení o průměrné délce ~200 bp (20, 35). Námi použitý systém GS Junior sice poskytuje přibližně sto tisíc čtení, ale jejich medián je přibližně 400 bp, což umožňuje preciznější mapování repetitivních sekvencí, čímž stoupá využitelnost jednotlivých čtení.
Finálním krokem je poté navržení MRN testu pro kvantitativní PCR v reálném čase pro konkrétní místo zlomu derivovaného chromozomu. V případě pacienta 2 byly navrženy testy pro chromozomové zlomy na der(4) a der(11), jejich účinnost byla ověřena srovnáním s již zavedeným testem k detekci fúzního transkriptu MLL-AF4. Ověření specificity reakce pro kvantitativní PCR v reálném čase bylo provedeno pomocí směsné DNA negativních kontrol. Rozsah senzitivity testu byl stanoven desítkovým ředěním DNA z období diagnózy leukemie.
Možnou limitací popsané metody k identifikaci nových molekulárních markerů (obr. 1 B) je nedostupnost referenční sekvence (NCBI, sestava 37.3) v místě přechodu jednotlivých kontigů některých chromozomů, zejména pak v subtelomerických oblastech a v repetitivních oblastech centromer, nebo molekulárně komplikované aberace (např. inzerce pod rozlišením molekulárně cytogenetických metod nebo paracentrické inverze). Další možnou limitací je stabilita nově identifikovaného molekulárního markeru v průběhu onemocnění. Z tohoto hlediska pro mikrodisekci volíme takovou chromozomovou aberaci, která je, v případě přítomnosti více klonů v karyotypu pacienta, zastoupena v každém klonu. V případě identifikace komplexních změn v karyotypu pacienta disekujeme všechny vhodné chromozomové aberace (viz der(4) a der(11) u pacienta 2), čímž je zajištěno monitorování MRN pomocí několika nezávislých cílů. U každého pacienta může být stabilita markeru (a to i již zavedeného) ovlivněna klonální evolucí. Z tohoto důvodu je vždy nutná korelace takto získaných výsledků s dalšími klinickými, biochemickými a hematologickými parametry, průtokovou cytometrií a cytogenetickou analýzou.
Kombinace metod cytogenetiky a molekulární biologie nám umožnila převést cytogenetický znak (derivovaný chromozom) leukemických buněk pacientů s AL až na molekulární (DNA sekvence) cíl určený k detekci a kvantifikaci MRN. Díky zde popsanému přístupu je možné použít nově nalezenou chromozomovou translokaci k vytvoření specifického MRN testu pro kvantitativní PCR v reálném čase, a umožnit tak senzitivní sledování úspěšnosti léčby pacientů s AL.
Závěr
Předložená práce popisuje zcela unikátní způsob identifikace molekulárního markeru pro sledování MRN u pacientů s AL. Jedná se o laboratorní přístup „šitý na míru“ nemocných s AL, který naplňuje naši představu o personalizované medicíně. V optimálním případě je MRN test v reálném čase na podkladě unikátního klonálně specifického znaku leukemické populace připraven již za 6 týdnů od stanovení diagnózy, což je zcela v souladu s potřebami rutinního diagnostického procesu u pacientů s AL, kdy první vzorky ke sledování MRN dostává laboratoř po doznění indukční terapie, což je přibližně 1 měsíc od záchytu onemocnění.
Poděkování
Tato práce byla podpořena grantem Ministerstva průmyslu a obchodu ČR (FR-TI2/579).
Podíl autorů na přípravě rukopisu
J. T. – návrh studie a provádění experimentů, sepsání rukopisu;
P. R. – návrh studie a provádění experimentů, bioinformatická práce, programování analyzačního sotware, kritická revize rukopisu;
Š. J. – sekvenování nové generace, revize rukopisu;
K. L. – provádění experimentů, revize rukopisu;
H. D. W. – návrh studie a provádění experimentů, kritická revize rukopisu;
L. T. – návrh experimentů a provádění mikrodisekcí chromozomů;
K. N. – provádění mikrodisekcí chromozomů;
Č. R. – návrh experimentů, experimentální práce, kritická revize rukopisu;
Ž. L. – experimentální práce, revize rukopisu;
K. T. – léčba pacientů s AL, sběr klinických dat, revize rukopisu;
Ž. P. – léčba pacientů s AL, sběr klinických dat, revize rukopisu;
K. M. – léčba pacientů s AL, sběr klinických dat, revize rukopisu;
P. S. – návrh studie, návrh experimentů, experimentální práce, finální revize rukopisu.
Seznam zkratek a vysvětlení pojmů
AL – akutní leukemie
ALL – akutní lymfoblastová leukemie
AML – akutní myeloidní leukemie
BAC – umělý bakteriální chromozom (Bacterial Artificial Chromosome) je DNA konstrukt nesoucí rozsáhlé úseky lidské DNA. Fluorescenčně značených BAC konstruktů je ve velké míře využíváno jako hybridizačních sond v molekulární cytogenetice pro mapování chromozomálních zlomů a definovaných genomických abnormalit
DNA – deoxyribonukleová kyselina
Degenerace primeru – proces, kdy při syntéze oligonukleotidu jsou v definovaném místě zařazovány do vznikajícího řetězce kombinace bazí. Kompletní degenerace v konkrétním místě primeru například znamená přítomnost všech čtyř bazí A, T, C, G v daném místě, v takto vzniklé směsi oligonukleotidů
emPCR – emulzní PCR, při které dochází k individuálním PCR reakcím v jednotlivých tukových kapičkách, drobných nanoreaktorech. Výsledná směs je potom nanášena na pikotitrační destičku, na které probíhá vlastní proces sekvenování nové generace
Fiber-FISH – fluorescenční in situ hybridizace na linearizovaných DNA vláknech
FISH – fluorescenční in situ hybridizace
mFISH – mnohobarevná fluorescenční in situ hybridizace
Kontig – sada překrývajících se DNA fragmentů, které společně reprezentují konsenzní úsek konkrétního chromozomu
LR-PCR – Long-range PCR; jedna z velmi náročných PCR aplikací, kdy jsou za specifických chemických podmínek amplifikovány dlouhé fragmenty DNA v rozsahu tisíců až desetitisíců párů bazí
mBAND – mnohobarevné fluorescenční pruhování chromozomů
MRN – minimální reziduální nemoc
Molecular combing / DNA combing – „česání“ chromozomů do podoby linearizovaných vláken DNA na silanizovaném skle
NGS – next generation sequencing; sekvenování nové generace, kdy je v průběhu jedné sekvenační reakce sekvenováno statisíce i více DNA vláken najednou. Přístroje s největší kapacitou umožňují de novo sekvenování celých genomů
Nick-translace – metoda (v našem případě fluorescenčního) značení DNA, při které jsou do jednoho z řetězců DNA enzymem DN-áza I zavedeny zlomy. Zlomy jsou rozeznávány enzymem DNA Polymeráza I, který v průběhu značící reakce do nově vznikajícího vlákna DNA inkorporuje standardní i fluorescenčně značené nukleotidy. Výsledkem je DNA fluorescenčně naznačená fluoroforem s definovanou vlnovou délkou, která je potom používána jako specifická sonda pro FISH
PCR – polymerázová řetězová reakce
Real-Time PCR – PCR v reálném čase
RNA – ribonukleová kyselina
Doručeno do redakce: 17. 9. 2012
Přijato po recenzi: 11. 1. 2013
Mgr. Tereza Jančušková
Laboratoř molekulární diagnostiky
Laboratoře CHAMBON s.r.o.
Evropská 176/16
160 00 Praha 6
Zdroje
1. Mrózek K, Heerema NA, Bloomfield CD. Cytogenetics in acute leukemia. Blood Rev 2004; 18 : 115-136.
2. Kern W, Haferlach C, Haferlach T, Schnittger S. Monitoring of minimal residual disease in acute myeloid leukemia. Cancer 2008; 112 : 4-16.
3. Grimwade D, Hills RK, Moorman AV, et al. Refinement of cytogenetic classification in acute myeloid leukemia: determination of prognostic significance of rare recurring chromosomal abnormalities among 5876 younger adult patients treated in the United Kingdom Medical Research Council trials. Blood 2010; 116 : 354-365.
4. Bassan R, Spinelli O, Oldani E, et al. Improved risk classification for risk-specific therapy based on the molecular study of minimal residual disease (MRD) in adult acute lymphoblastic leukemia (ALL). Blood 2009; 113 : 4153-4162.
5. Brüggemann M, Gökbuget N, Kneba M. Acute lymphoblastic leukemia: monitoring minimal residual disease as a therapeutic principle. Semin Oncol 2012; 39 : 47-57.
6. Cairoli R, Beghini A, Grillo G, et al. Prognostic impact of c-KIT mutations in core binding factor leukemias: an Italian retrospective study. Blood 2006; 107 : 3463-3468.
7. Schlenk RF, Döhner K, Krauter J, et al. Mutations and treatment outcome in cytogenetically normal acute myeloid leukemia. N Engl J Med 2008; 358 : 1909-1918.
8. Rossi G, Minervini MM, Carella AM, et al. Comparison between multiparameter flow cytometry and WT1-RNA quantification in monitoring minimal residual disease in acute myeloid leukemia without specific molecular targets. Leuk Res 2012; 36 : 401-406.
9. Campana D. Minimal residual disease in acute lymphoblastic leukemia. Hematology Am Soc Hematol Educ Program 2010; 7-12.
10. Plachy R, Zejskova L, Cmejla R, et al. Five-color multiplex Real-Time PCR technology to detect over 75 recurrent chromosomal abnormalities in acute myeloid leukemia; benefits for minimal residual disease detection. Blood 2011; 118 : 1083 (Abstract 2526).
11. Weise A, Timmermann B, Grabherr M, et al. High-throughput sequencing of microdissected chromosomal regions. Eur J Hum Genet 2010; 18 : 457-462.
12. Liehr T, Weise A, Heller A, et al. Multicolor chromosome banding (MCB) with YAC/BAC-based probes and region-specific microdissection DNA libraries. Cytogenet Genome Res 2002; 97 : 43-50.
13. Craig FE, Foon KA. Flow cytometric immunophenotyping for hematologic neoplasms. Blood 2008; 111 : 3941-3967.
14. Peters JM, Ansari MQ. Multiparameter flow cytometry in the diagnosis and management of acute leukemia. Arch Pathol Lab Med 2011; 135 : 44-54.
15. Brüggemann M, Raff T, Kneba M. Has MRD monitoring superseded other prognostic factors in adult ALL? Blood 2012; 120 : 4470-4481.
16. Schnittger S, Weisser M, Schoch C, et al. New score predicting for prognosis in PML-RARA+, AML1-ETO+, or CBFBMYH11+ acute myeloid leukemia based on quantification of fusion transcripts. Blood 2003; 102 : 2746-2755.
17. Weisser M, Kern W, Schoch C, et al. Risk assessment by monitoring expression levels of partial tandem duplications in the MLL gene in acute myeloid leukemia during therapy. Haematologica 2005; 90 : 881-889.
18. Perea G, Lasa A, Aventín A, et al. Prognostic value of minimal residual disease (MRD) in acute myeloid leukemia (AML) with favorable cytogenetics [t(8;21) and inv(16)]. Leukemia 2006; 20 : 87-94.
19. Abdelhamid E, Preudhomme C, Helevaut N, et al. Minimal residual disease monitoring based on FLT3 internal tandem duplication in adult acute myeloid leukemia. Leuk Res 2012; 36 : 316-323.
20. Chen W, Kalscheuer V, Tzschach A, et al. Mapping translocation breakpoints by next-generation sequencing. Genome Res 2008; 18 : 1143-1149.
21. Lu CM, Kwan J, Baumgartner A, et al. DNA probe pooling for rapid delineation of chromosomal breakpoints. J Histochem Cytochem 2009; 57 : 587-597.
22. De Braekeleer E, Douet-Guilbert N, Basinko A, et al. Using bacterial artificial chromosomes in leukemia research: the experience at the university cytogenetics laboratory in Brest, France. J Biomed Biotechnol 2011; 2011 : 329471.
23. Bickmore WA. Karyotype analysis and chromosome banding. 2001; eLS. disease in acute myeloid leukemia. Cancer 2008; 112 : 4-16.
24. Mundle SD, Koska RJ. Fluorescence In Situ Hybridization: A major milestone in luminous cytogenetics. In: Coleman WB, Tsongalis GJ. Molecular diagnostics for the clinical laboratorian. 2nd Edition, New Jersey, Humana Press, 2006; 189-201.
25. Woodcock CL, Dimitrov S. Higher-order structure of chromatin and chromosomes. Curr Opin Genet Dev 2001; 11 : 130-135.
26. Weissmann F, Lyko F. Cooperative interactions between epigenetic modifications and their function in the regulation of chromosome architecture. Bioassays 2003; 25 : 792-797.
27. Baxter J, Aragón L. A model for chromosome condensation based on the interplay between condensin and topoisomerase II. Trends Genet 2012; 28 : 110-117.
28. Weier HU. DNA fiber mapping techniques for the assembly of high-resolution physical maps. J Histochem Cytochem 2001; 49 : 939-948.
29. Lebofsky R., Bensimon A. Single DNA molecule analysis: Applications of molecular combing. Brief Funct Genomic Proteomic 2003; 4 : 385-396.
30. Labit H, Goldar A, Guilbaud G, et al. A simple and optimized method of producing silanized surfaces for FISH and replication mapping on combed DNA fibers. Biotechniques 2008; 45 : 649-652.
31. Greulich-Bode KM, Wang M, Rhein AP, Weier JF, Weier HU. Validation of DNA probes for molecular cytogenetics by mapping onto immobilized circular DNA. Mol Cytogenet 2008; 23 : 28.
32. Engelen JJ, Albrechts JC, Hamers GJ, et al. A simple and efficient method for microdissection and microFISH. J Med Genet 1998; 35 : 265-268.
33. Shim SH, Kyhm JH, Chung SR, et al. Generation of FISH probes using laser microbeam microdissection and application to clinical molecular cytogenetics. J Microbiol Biotechnol 2007; 17 : 1079-1082.
34. Grossmann V, Höckner M, Karmous-Benailly H, et al. Parental origin of apparently balanced de novo complex chromosomal rearrangements investigated by microdissection, whole genome amplification, and microsatellite-mediated haplotype analysis. Clin Genet 2010; 78 : 548-553.
35. Chen W, Ullmann R, Langnick C, et al. Breakpoint analysis of balanced chromosome rearrangements by next-generation paired-end sequencing. Eur J Hum Genet 2010; 18 : 539-543.
Štítky
Hematológia Interné lekárstvo Onkológia
Článok vyšiel v časopiseTransfuze a hematologie dnes
Najčítanejšie tento týždeň
2013 Číslo 1- Nejasný stín na plicích – kazuistika
- Těžké menstruační krvácení může značit poruchu krevní srážlivosti. Jaký management vyšetření a léčby je v takovém případě vhodný?
- I „pouhé“ doporučení znamená velkou pomoc. Nasměrujte své pacienty pod křídla Dobrých andělů
- Parazitičtí červi v terapii Crohnovy choroby a dalších zánětlivých autoimunitních onemocnění
-
Všetky články tohto čísla
- Rizikové monoklonální gamapatie nejasného významu – léčit nebo neléčit?
- Klinické aspekty „pravé“ nesekretorické formy mnohočetného myelomu
- Výskyt venózního tromboembolismu u žen v časové souvislosti s užíváním hormonální antikoncepce
- Přístupy k antimykotické terapii: od profylaxe k léčbě
- Kryokonzervace krve – historie, metody a současnost
- Úvodník
- Myeloproliferative neoplasms in Budd-Chiari syndrome and portal vein thrombosis: a meta-analysis
- Cardiovascular risk factors in hematopoietic cell transplantation survivors: role in development of subsequent cardiovascular disease
- A randomized trial of dasatinib 100 mg versus imatinib 400 mg in newly diagnosed chronic-phase chronic myeloid leukemia
- Genome-wide association study identifies germline polymorphisms associated with relapse of childhood acute lymphoblastic leukemia
- A randomized phase II study to evaluate tacrolimus in combination with sirolimus or methotrexate after allogeneic hematopoietic cell transplantation
- Soutěž
- Realita vyšetřování minimální reziduální nemoci u akutních leukemií aneb kolik laboratorní akrobacie je skutečně třeba?
- Identifikace nových molekulárních markerů pro sledování minimální reziduální nemoci u akutních leukemií
- Transfuze a hematologie dnes
- Archív čísel
- Aktuálne číslo
- Iba online
- Informácie o časopise
Najčítanejšie v tomto čísle- Rizikové monoklonální gamapatie nejasného významu – léčit nebo neléčit?
- Kryokonzervace krve – historie, metody a současnost
- Přístupy k antimykotické terapii: od profylaxe k léčbě
- Klinické aspekty „pravé“ nesekretorické formy mnohočetného myelomu
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



